 近日,武汉大学生命科学学院朱玉贤院士、周宇教授课题组合作在国际学术期刊Nature Communications在线发表了题为”Multi-strategic RNA-seq analysis reveals a high-resolution transcriptional landscape in cotton”的研究论文。该研究通过整合运用四种高通量RNA测序技术,系统地研究了亚洲棉16个不同组织器官的RNA转录情况,建立了亚洲棉基因组转录全景图。生命科学学院王坤副研究员和周宇实验室王得和博士研究生为论文共同第一作者,朱玉贤院士和周宇教授为论文的共同通讯作者。武汉未来组承担了本研究中PacBio Iso-seq转录组的测序和部分分析工作。棉花是一种重要的天然纤维作物,也是研究细胞分化、伸长和细胞壁发育调控的重要模式植物,构建棉花全面的高分辨率转录景观,有利于对其各种生物过程分子机制的研究。本研究整合了四种互补的高通量技术,包括长读长Pacbio Iso-seq,链特异性RNA-seq,CAGE-seq和PolyA-seq,对亚洲棉16个不同组织或器官类型的转录情况进行测序,从而系统性的解析亚洲棉基因组转录全景。研究者通过编写的IGIA算法整合了四种技术的集成数据,从中鉴定出包括TSS,TES和内含子在内的的基因元件,对亚洲棉基因组的基因结构进行准确注释。最终,从36,826个基因中鉴定出94,170个转录本,其中有56.7%的基因只有一个转录本,而17,101个基因有两个以上转录异构体(图1c)。通过随机挑选的170多对注释差异位点的RT-PCR验证表明IGIA的准确率达到98%。
近日,武汉大学生命科学学院朱玉贤院士、周宇教授课题组合作在国际学术期刊Nature Communications在线发表了题为”Multi-strategic RNA-seq analysis reveals a high-resolution transcriptional landscape in cotton”的研究论文。该研究通过整合运用四种高通量RNA测序技术,系统地研究了亚洲棉16个不同组织器官的RNA转录情况,建立了亚洲棉基因组转录全景图。生命科学学院王坤副研究员和周宇实验室王得和博士研究生为论文共同第一作者,朱玉贤院士和周宇教授为论文的共同通讯作者。武汉未来组承担了本研究中PacBio Iso-seq转录组的测序和部分分析工作。棉花是一种重要的天然纤维作物,也是研究细胞分化、伸长和细胞壁发育调控的重要模式植物,构建棉花全面的高分辨率转录景观,有利于对其各种生物过程分子机制的研究。本研究整合了四种互补的高通量技术,包括长读长Pacbio Iso-seq,链特异性RNA-seq,CAGE-seq和PolyA-seq,对亚洲棉16个不同组织或器官类型的转录情况进行测序,从而系统性的解析亚洲棉基因组转录全景。研究者通过编写的IGIA算法整合了四种技术的集成数据,从中鉴定出包括TSS,TES和内含子在内的的基因元件,对亚洲棉基因组的基因结构进行准确注释。最终,从36,826个基因中鉴定出94,170个转录本,其中有56.7%的基因只有一个转录本,而17,101个基因有两个以上转录异构体(图1c)。通过随机挑选的170多对注释差异位点的RT-PCR验证表明IGIA的准确率达到98%。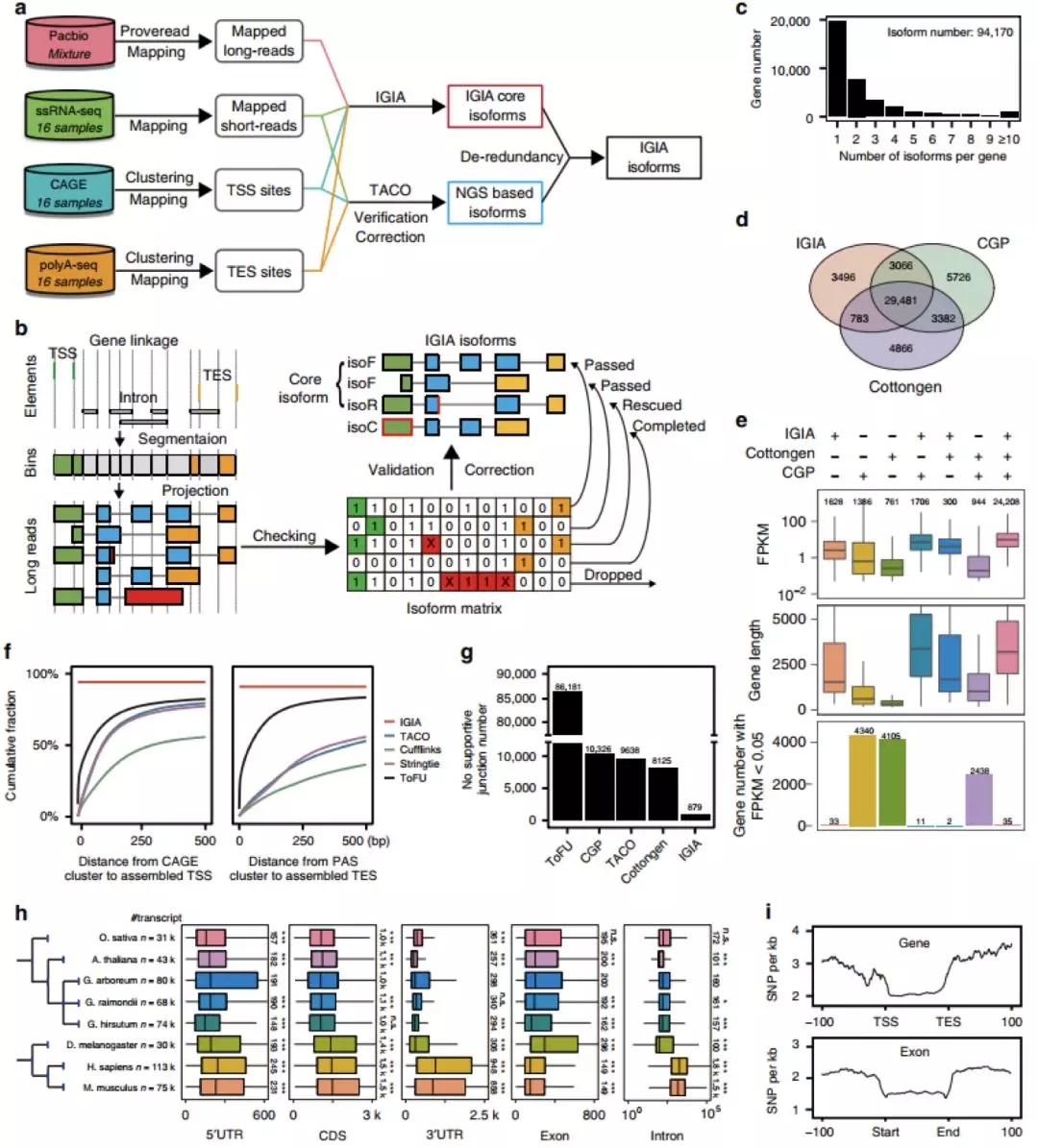
图1 针对亚洲棉高分辨率转录组景观设计的多重RNA-Seq策略。
研究者通过分析来自于22,863个亚洲棉基因的44,728个TSS cluster发现38.4%的基因有两个及以上转录起始位点(图2a)。多启动子基因的不同TSS中,远端TSS使用更为频繁(图2c)。可变启动子产生的RNA的异质性显着增加了转录组多样性,从而产生蛋白组的可塑性,本研究数据表明可变启动子可能分别改变了5,888个基因的UTR长度和2,800多个基因的编码蛋白长度(图2d)。进一步发现了具有胚珠组织或发育阶段特异性的TSS可变基因如NRT1.2,编码基因TSS可变造成NRT蛋白四个跨膜TM的丢失,产生NRT-L和NRT-S两种长度的蛋白异构体,造成结构变化影响硝酸根转运能力。上述结果表明,差异调控的选择性TSSs是棉花mRNA的一个共同特征,通常在mRNA或蛋白中产生选择性的N-端来调控发育。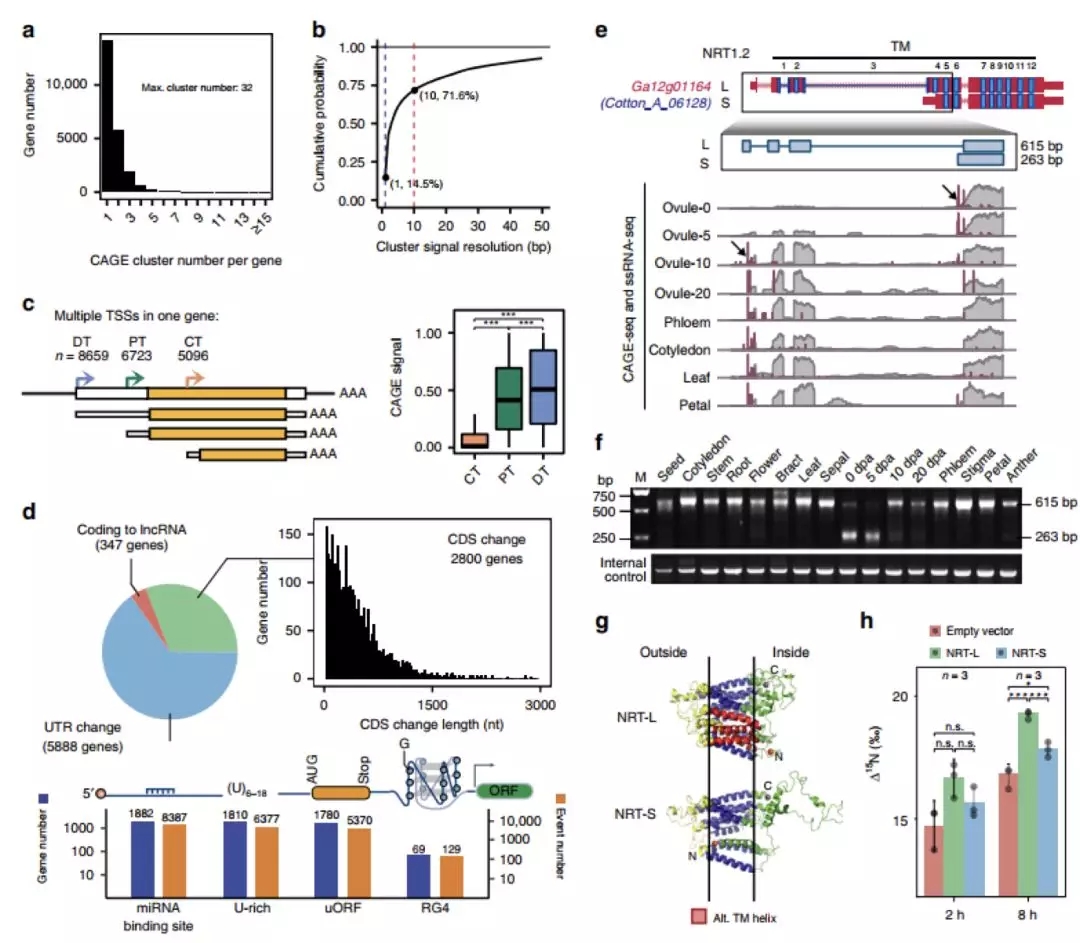
图2 亚洲棉中多转录起始位点和可变启动子的使用。
于PolyA-seq的3’末端信息,我们对16个组织中所有表达基因的全基因组TES进行了分析。揭示了它们的序列特征、发育过程中的动态规律和组织规范。结果表明在棉花中所有表达的基因中,有40.2%的人至少有两个TES,基因的3’末端同样存在多TES调控的现象,在发育和组织分化过程中,很多基因的转录终止通过可变的TES调节其3’UTR的长度。
本研究基于IGIA注释,对亚洲棉的23,451个多外显子基因进行了可变剪接(AS)的系统分析。结果表明,所有AS事件中内含子保留(RI)占62.2%(图3a),在所有报道的植物中是最高的。此外,某些基因中的几个区域显示出高度丰富的AS事件,研究者将其称为AS热点。进一步分析表明,大多数AS热点影响保守的蛋白质结构域(图3g-h)。微外显子(micro-exon)是动物中首先报道的一种微小外显子,其长度短至仅3nt,该研究通过系统分析,首次在棉花中鉴定到微外显子的存在,还通过在多个植物物种的比对,发现了一个具有潜在重要作用的45nt的保守微外显子。
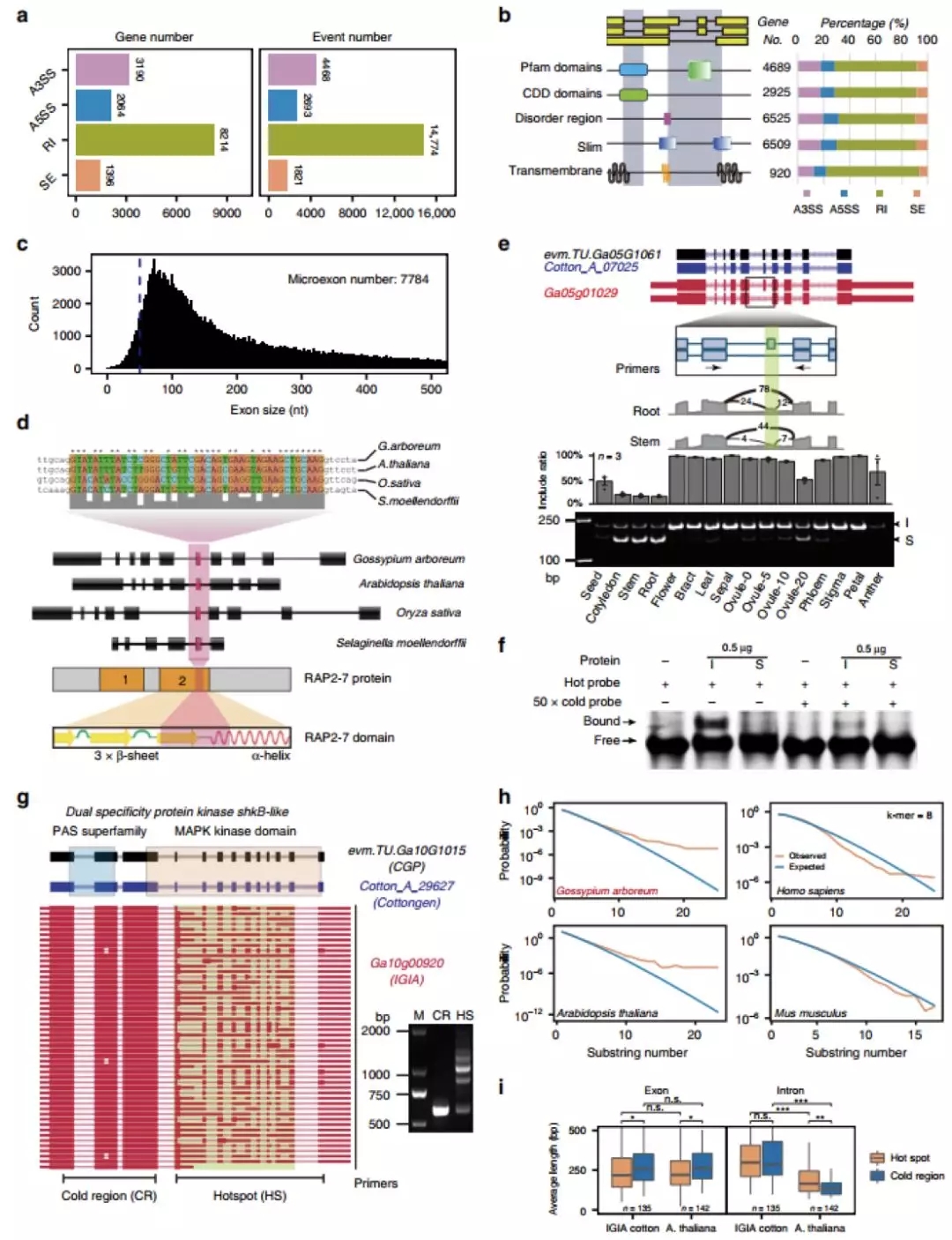
图3 亚洲棉中选择性剪接调控及热点
最后,该研究通过分析三代测序的全长转录本,发现约5%的亚洲棉基因存在转录通读现象,形成类似原核生物的多顺反子(Polycistron)转录本(图4a)。这些基因相互临近,平均距离明显小于其他相邻的独立转录基因之间的距离(图4c)。另外通过对这些多顺反子上的基因对的功能分析发现,他们往往倾向于执行相同功能或位于同一个分子作用网络(图4f-h)。该结果表明这些基因对的转录水平的共同调控现象可能利于其共同完成某一生物学过程。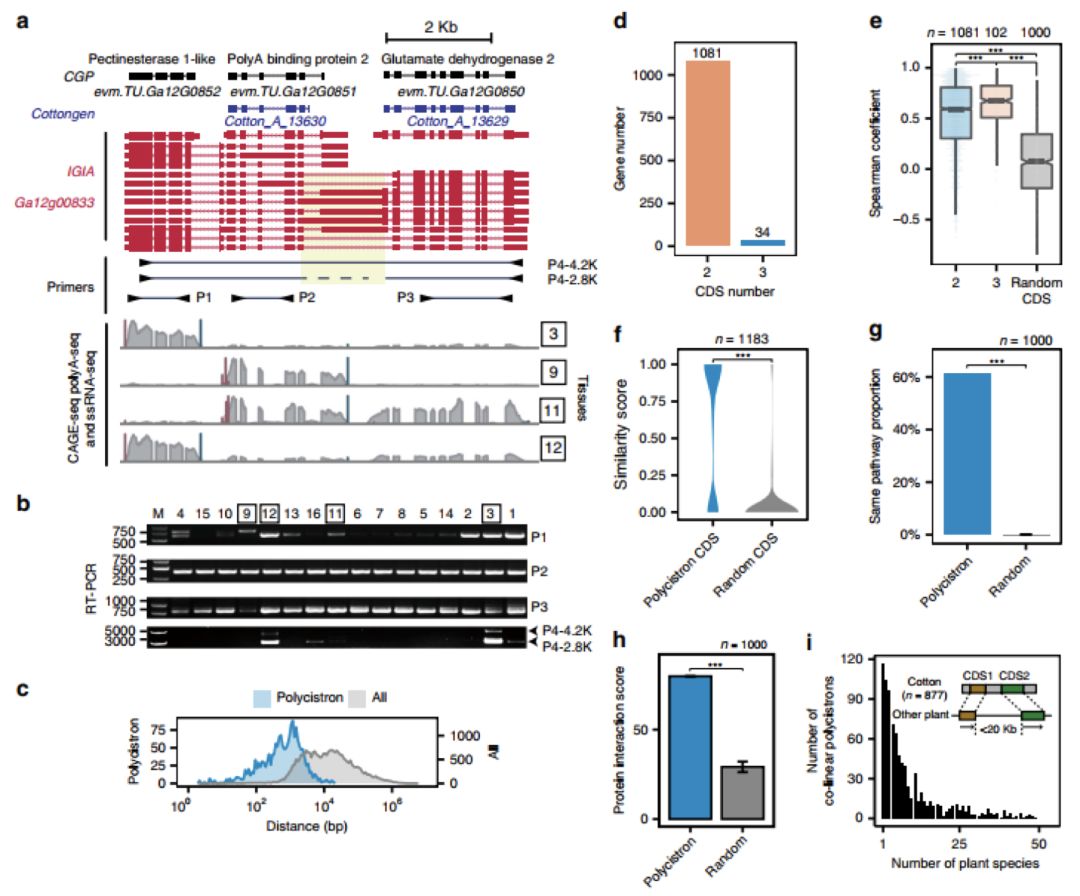
本研究通过IGIA算法整合了四种互补的高通量技术:用于直接读取全长转录本异构体的PacbioIso-seq,用于定量表达和剪接的链特异性RNA-seq,准确定义转录起始和终止位点的CAGE-seq和检测聚腺苷酸化位点的PolyA-seq,生成了高分辨率的亚洲棉转录组景观图谱。发现并验证了棉花发育中基因表达调控的不同模式,如可变启动子和终止子调节、微外显子剪接、多顺反子转录通读和RNA选择性剪接热区等复杂现象,对未来棉花功能基因组学的进一步发展意义重大。
参考文献:
Wang K, Wang D, Zheng X, et al. Multi-strategic RNA-seqanalysis reveals a high-resolution transcriptional landscape in cotton[J].Nature communications, 2019, 10(1): 1-15.












发表评论
想参加讨论吗?请尽情讨论吧!