Next系列软件应用 | 世界首个草莓T2T,NextDenovo完成图必备工具

二倍体森林草莓Fragaria vesca(2n=14)原产于欧洲和亚洲,由于其植株较小、杂合度低以及容易遗传转化被当作草莓研究的模式物种。2011年早期发布了F.vesca cv.‘Hawaii 4’的基因组序列草图(v1.0),2018年报道了基于PacBio测序和光学图谱的染色体水平组装。然而,目前F.vesca基因组仍然没有达到完整的T2T水平,表明其基因组质量有继续提升的空间。
本研究使用ONT和PacBio HiFi测序组装了一个高质量的T2T F.vesca基因组,填补了目前可用参考基因组中的所有剩余空白,并构建了染色体核型演化模型,探究了八倍体草莓的祖先二倍体。
本研究对二倍体草莓测序产生的约32.67 Gb的ONT超长测序reads、27.31 Gb的PacBio HiFi reads和32.10 Gb的Illumina reads以及44.56Gb的Hi-C数据进行基因组组装。使用NextDenovo软件对ONT数据进行组装(https://github.com/Nextomics/NextDenovo),使用NextPolish(1.4.1版本)软件对其进行纠错。
NextDenovo/NextPolish软件是由希望组自主研发的三代测序基因组组装工具,在极大减少计算资源和运行时间的情况下,仍然能够组装出高质量基因组,具有高纠错、高效组装、高准确度的优势,已帮助众多科研人员进行基因组的组装以及文章的发表。
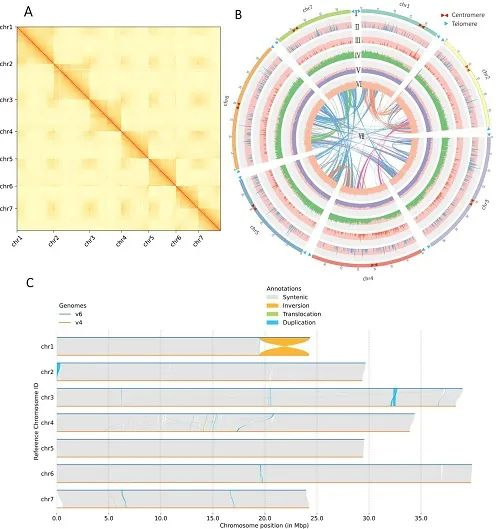
图1 F.vesca的全基因组组装
最终组装得到的无gap基因组大小为220.8Mb,Contig N50达到了34.34Mb,BUSCO值为98.8%,注释到了36173个蛋白质编码基因,其中1153个为新注释的基因,鉴定到7条染色体上所有14个端粒和7个着丝粒。系统发育分析表明,F.vesca和F.viridis是栽培的八倍体草莓F.×ananassa的祖先,而F. iinumae和F. nipponica与其亲缘关系较远。
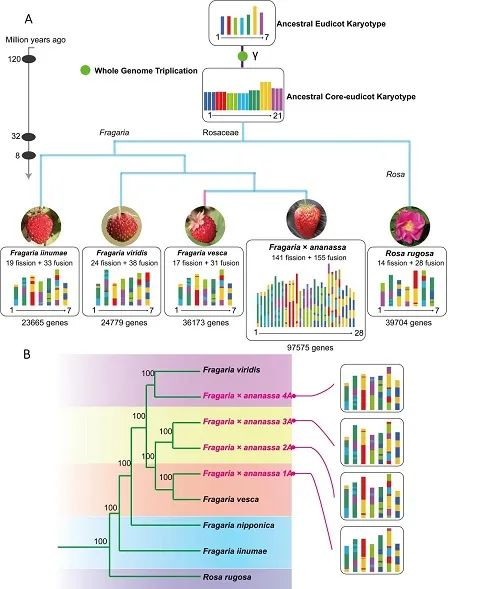
图2 Fragaria vesca对栽培八倍体草莓的贡献
综上,这一高质量无gap的T2T F.vesca基因组,结合对栽培草莓起源的系统发育推断,提供了对Fragaria基因组进化的深入了解,并促进了草莓遗传学和分子育种的发展。





发表评论
想参加讨论吗?请尽情讨论吧!