项目文章|三代测序(TGS)技术助力水稻泛基因组研究再攀高峰
基因组研究热点+前沿测序技术=基因组学顶级期刊!
泛基因组是一个群体中所有基因组的集合,在众多领域(尤其作物科学领域)的基因组研究中显示出巨大的潜力,泛基因组的构建和基因存在-缺失变异(PAVs)分析是基因组研究的新热点。目前基于二代测序技术(SGS),初步构建了主要作物(如水稻、玉米、大豆)的泛基因组,其中水稻泛基因组大小比水稻参考基因组(NipRG)日本晴要大近270 Mb。然而,使用二代测序数据构建的泛基因组仍存在基因组不完整和基因注释不准确的缺点。这些问题在很大程度上可以通过前沿测序技术——长读长的三代测序解决。
近日,上海交通大学生命科学技术学院韦朝春团队和中国农业科学院作物科学研究所合作完成基于三代测序数据的水稻泛基因组构建及分析,相关成果论文 “Long-read sequencing of 111 rice genomes reveals significantly larger pan-genomes”在基因组学顶级期刊《Genome Research》发表。希望组为本研究提供了二代和三代测序服务,自主研发的NextDenovo软件被应用于项目的关键环节——水稻基因组组装。
研究概要
研究挑选了111个代表性水稻品种,结合二代和三代测序技术进行全基因组测序,获得了连续性和完整性很高的个体基因组,并在此基础上构建了高质量水稻泛基因组,同时填补了9个代表性水稻群体的高质量参考基因组,其中包括5个gapless水稻基因组。
研究结果
1. 水稻泛基因组构建及特征分析
结合111个水稻基因组构建的高质量泛基因组有879Mb的非冗余新序列(序列相似度<90%,长度> 500bp)。新序列中转座因子占一半以上,其中逆转录因子52.71%,DNA转座子16.05%,名为Gypsy的长末端重复(LTRs)逆转录元件占比达47.83%。新序列分布于每条染色体上,Chr1新序列数量最多,Chr11新序列长度最长。除了 Chr4 和 Chr11 的端粒附近的两个峰以外,含高密度新序列的基因组区域倾向于位于着丝粒附近。另外,在所有水稻基因组中,野生稻包含的重复序列 LTRs 明显多于栽培稻。泛基因组变异分析的结果显示缺失和易位为主要的结构变异(structural variations,SVs)模式。
在水稻泛基因组中一共发现19319个新编码基因(分布于2132个新基因家族),其中89.5%包含至少一个功能区域。进一步统计分析,在所有基因中65.7%为核心基因,14.4%为候选核心基因(存在于90%的样本),19.6%为非核心基因家族基因(少于90%但不仅在一个样本中存在),0.2%为特有基因。
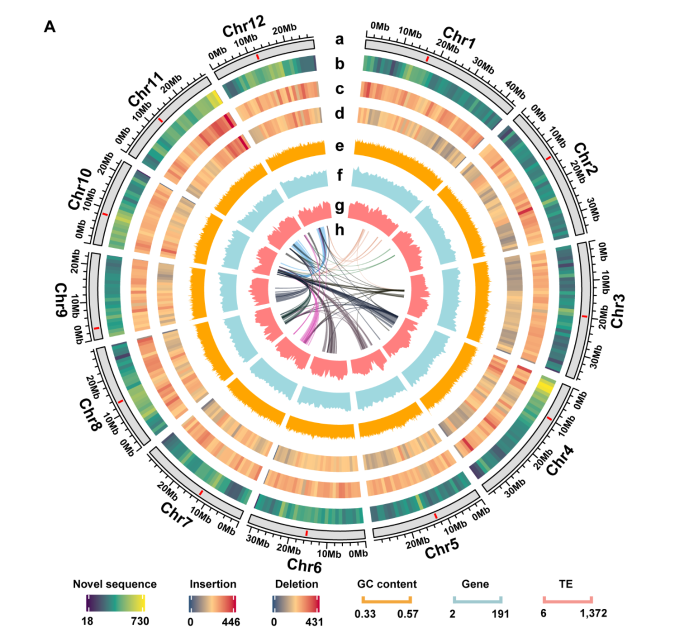
图一 水稻泛基因组的基因组特征
2.测序技术及品种数量对构建泛基因组的影响
后续分析中分别构建了相同品种水稻的SGS和TGS泛基因组及品种数量不等的TGS泛基因组,比较分析结果显示:同样的测序技术,加入品种数量不同时,品种数量越多得到新序列越多;品种数量相同时,基于TGS技术构建的泛基因组比基于SGS技术构建的泛基因组得到的新序列多。
进一步对同品种SGS和TGS构建的泛基因进行PAVs分析,将全部基因分为TGS偏好、SGS偏好及无偏好三组。对比分析发现TGS偏好基因的GC含量更高,CDS长度更短;多数SGS偏好基因CDS区与重复元件重叠,与DNA转座子和LTR相关;具有较高LINEs和RC/Helitron比例的基因在TGS中比SGS中更频繁。这些结果表明,SGS数据在检测基因PAVs时往往会得到更高的假阳性率,尤其是对于包含重复序列的基因。
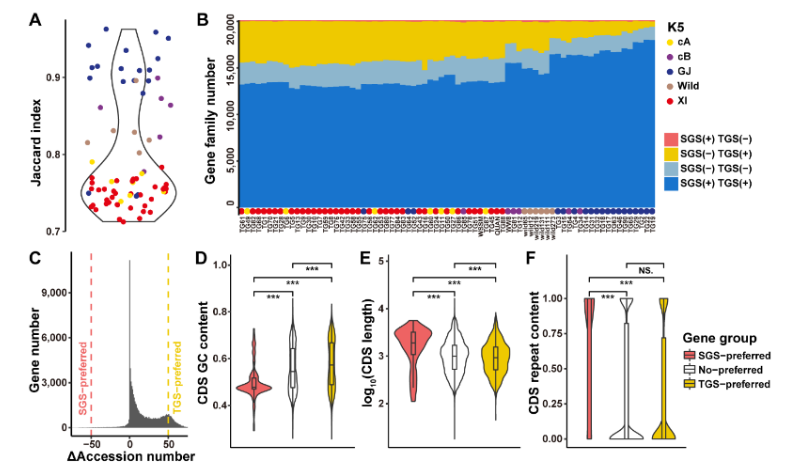
图二 SGS和TGS基因家族比较分析
3. 栽培稻表型关联分析
通过栽培稻基因PAV和表型关联分析,检测到14,471个显著的基因PAVs和表型的关联(8130个基因和9个表型)。例如LOC_Os01g27930(一种反转录转座子蛋白)的缺失与籽粒长宽比增加相关,而它的存在也与籽粒宽度增加相关;SD-1的缺失会导致植株高度的降低。这些结果表明,基因PAVs对水稻表型变异可能有重要贡献。
小结
本研究结合SGS和TGS数据构建了水稻高质量泛基因组,全面解析水稻基因组相关特征。研究结果将全面推动水稻的功能基因组学研究,为水稻品种改良提供重要参考资源。




发表评论
想参加讨论吗?请尽情讨论吧!