硬核爆发-希望组2020三代基因组文章集锦-植物篇
高质量基因组揭示棉花A亚基因组起源[1]
影响因子:25.455
发表日期:2020.04.13
三代测序平台:PacBio RSII&Sequel
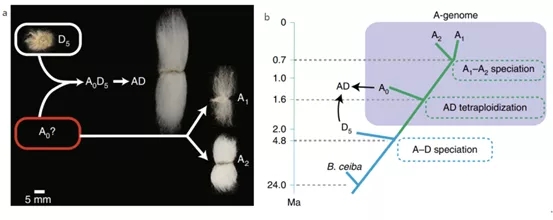
图1 棉花A基因组起源与进化模型(a)和重要进化事件(b)
ONT测序助力攻克首个高质量角苔参考基因组[2]
影响因子:13.297
发表日期:2020.02.10
三代测序平台:Nanopore PromethION
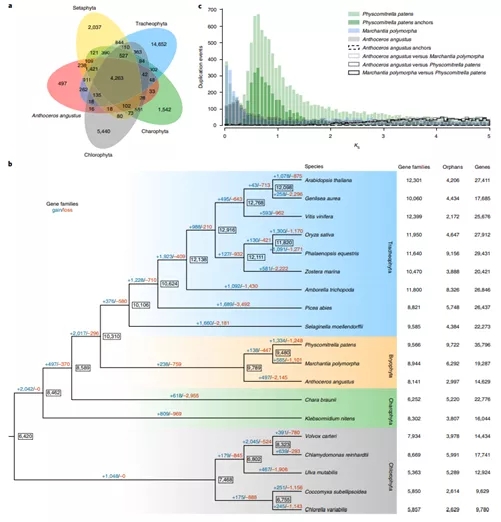
图2 芽胞角苔与18个绿色植物的比较基因组分析。a, 基于OrthoMCL的基因家族聚类比较。b, 19个绿色植物的基因家族获得(+)/丢失(-)情况比较,红框标注苔藓类群分支。c, 芽胞角苔、小立碗藓和地钱的全基因组加倍事件分析。
高质量油桐基因组,荣登GPB期刊“封面故事”[3]
影响因子:6.597
发表日期:2020.04.07
三代测序平台:PacBio RSII
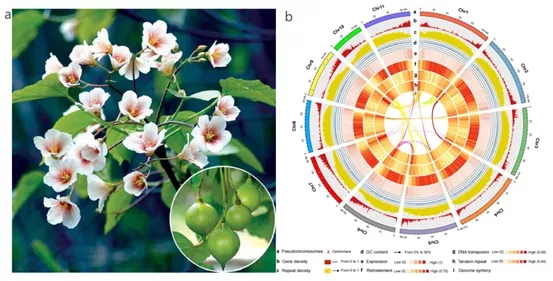
图3 油桐(a)及其基因组景观(b)
白木香—瑞香科第一个染色体水平基因组[4]
影响因子:4.688
发表日期:2020.03.02
三代测序平台:Nanopore GridION
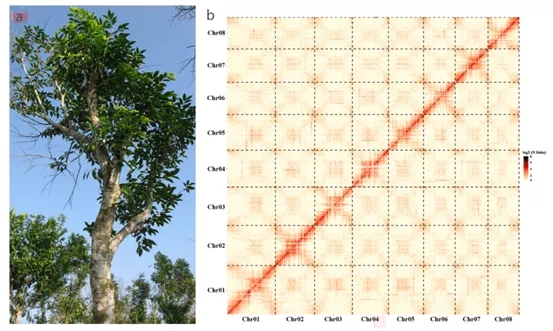
图4 白木香(a)及其基因组Hi-C热图(b)
园艺观赏植物文竹染色体水平基因组[5]
影响因子:3.368
发表日期:2020.04.01
三代测序平台:Nanopore GridION
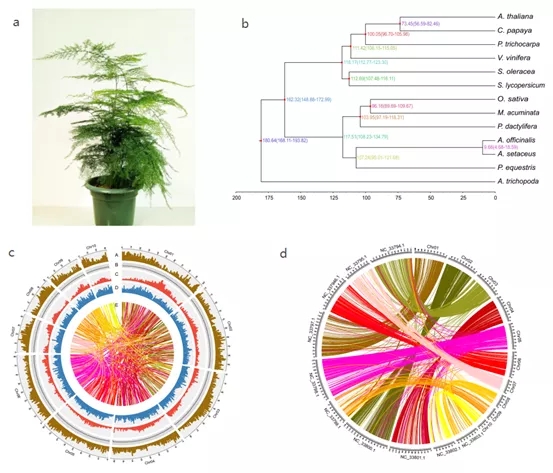
图5 文竹高质量基因组。(a)文竹。(b)基于1002个单拷贝直系同源基因的系统发生树。(c)文竹基因组景观。(d)文竹与芦笋基因组线性比较
影响因子:4.688
发表日期:2020.02.26
三代测序平台:Nanopore GridION
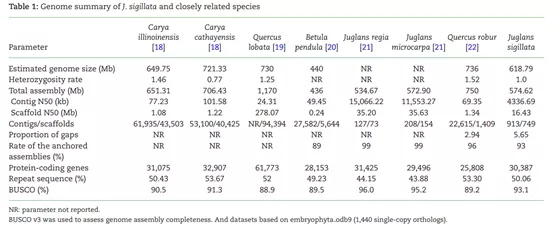
图6 铁核桃与其近源种基因组比较
 ▼
▼参考文献:
1. Huang, G. et al. Genome sequence of Gossypium herbaceum andgenome updates of Gossypium arboreum and Gossypium hirsutumprovide insights into cotton A-genome evolution. Nat Genet (2020)doi:10.1038/s41588-020-0607-4.
2. Zhang, J., Fu, X., Li, R. et al. The hornwort genome and early landplant evolution. Nat. Plants 6, 107–118 (2020).https://xs.scihub.ltd/https://doi.org/10.1038/s41477-019-0588-4
3. Zhang, L. et al. Tung Tree (Vernicia fordii) Genome ProvidesA Resource for Understanding Genome Evolution and Improved Oil Production.Genomics, Proteomics & Bioinformatics S167202291830216X (2020)doi:10.1016/j.gpb.2019.03.006.
4. Ding, X. et al. Genome sequence of the agarwood tree Aquilariasinensis (Lour.) Spreng: the first chromosome-level draft genome in theThymelaeceae family. GigaScience 9, giaa013 (2020).
5. Li, S.-F. et al. Chromosome-level genome assembly, annotation andevolutionary analysis of the ornamental plant Asparagus setaceus. HorticRes 7, 48 (2020).
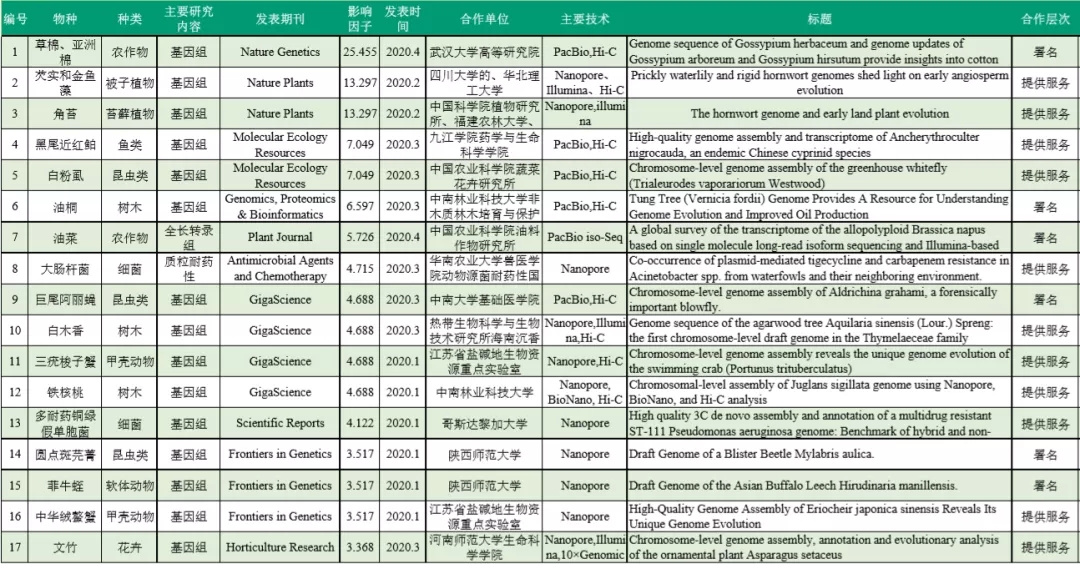
6. Ning, D.-L. et al. Chromosomal-level assembly of Juglanssigillata genome using Nanopore, BioNano, and Hi-C analysis. GigaScience 9,giaa006 (2020).





发表评论
想参加讨论吗?请尽情讨论吧!