项目文章 | 深圳基因组所商连光团队基于结构变异图谱挖掘到重要耐盐优异基因
近日,中国农业科学院深圳农业基因组研究所(岭南现代农业科学与技术广东省实验室深圳分中心)联合福建省农业科学院生物技术研究所、崖州湾国家实验室、厦门大学生命科学学院等单位在植物学顶级学术期刊 New Phytologist (IF= 9.4) 上发表了题为“Identification of salt tolerance-associated presence-absence variations in the OsMADS56 gene through the integration of DEGs dataset and eQTL analysis”的研究论文,该研究全面揭示了结构变异对水稻盐胁迫下基因表达和耐盐性状的重要影响,结合结构变异挖掘到关键耐盐基因OsMADS56,为水稻耐盐育种改良提供了新的优异靶位点。希望组为本研究提供优质的三代测序服务。

目前,土壤盐碱化已成为全球范围中威胁作物生长和生产力的主要环境因素之一。水稻作为全球最重要的谷类作物,时常受到盐胁迫的危害,挖掘耐盐优异等位基因,提高水稻在盐胁迫下的生产力成为农业育种的关键挑战,是实现“以种适地”的关键环节。结构变异(Structural variations,简称SVs)是遗传多样性的重要来源,对基因组的影响比起SNP更大,与许多表型变异和环境适应有关。插入缺失变异PAVs是SV一种主要类型,过去由于短读长测序的限制,PAVs很难被高效挖掘和鉴定,是未被广泛挖掘的“隐藏”的基因组变异。由于PAV和SNP并不是紧密连锁,PAV作为SNP的补充可以挖掘到更多优异变异资源。
该团队前期利用全球核心种质资源构建了群体规模最大、基因组充分注释、稻属中最为系统的图形超级泛基因组,解析了全面的基因组序列变异图谱(Shang et al., 2022);构建了核心种质群体在正常和盐胁迫下的表达谱,结合水稻超级泛基因组图谱在全基因组水平系统分析了耐盐性相关的SNP-eQTL,并成功克隆了关键耐盐新基因STG5(Wei et al., 2024),该基因优异单倍型导入到主栽品种中可以提高耐盐性,为耐盐水稻品种的培育奠定了良好的理论基础和种质材料。进一步本研究利用PAV变异挖掘耐盐新基因,评估正常和盐胁迫条件下影响基因表达的PAV,进行PAV-eQTL分析并分别鉴定到2427个和2898个正常和盐胁迫条件下的PAV影响的基因,其中盐胁迫下特异性响应的基因有1206个,为挖掘由结构变异引起的耐盐相关新等位基因提供了有价值的数据集。
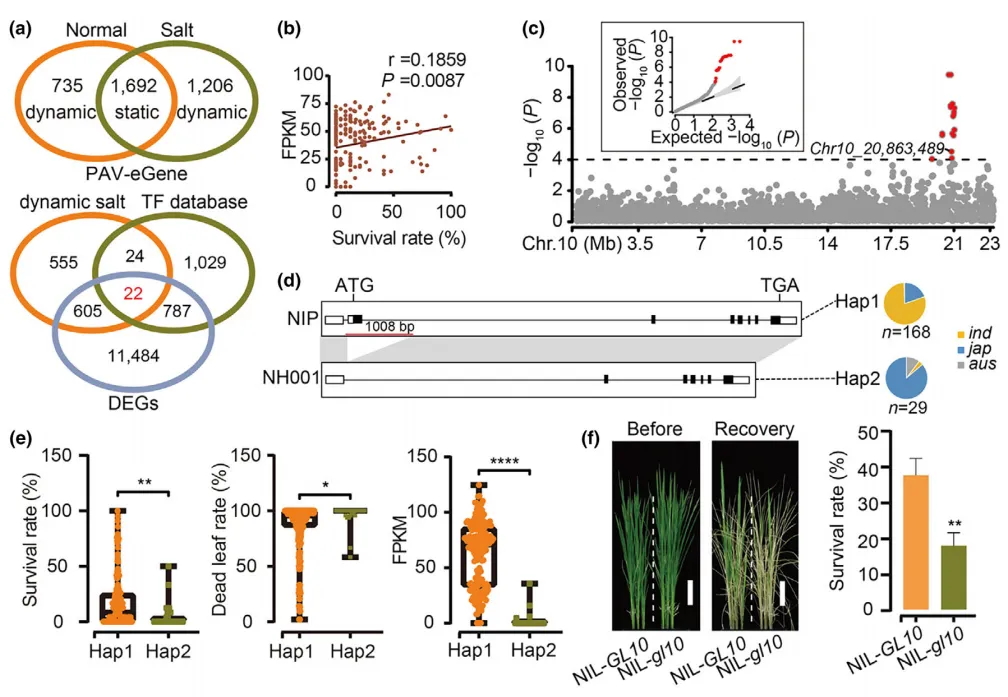
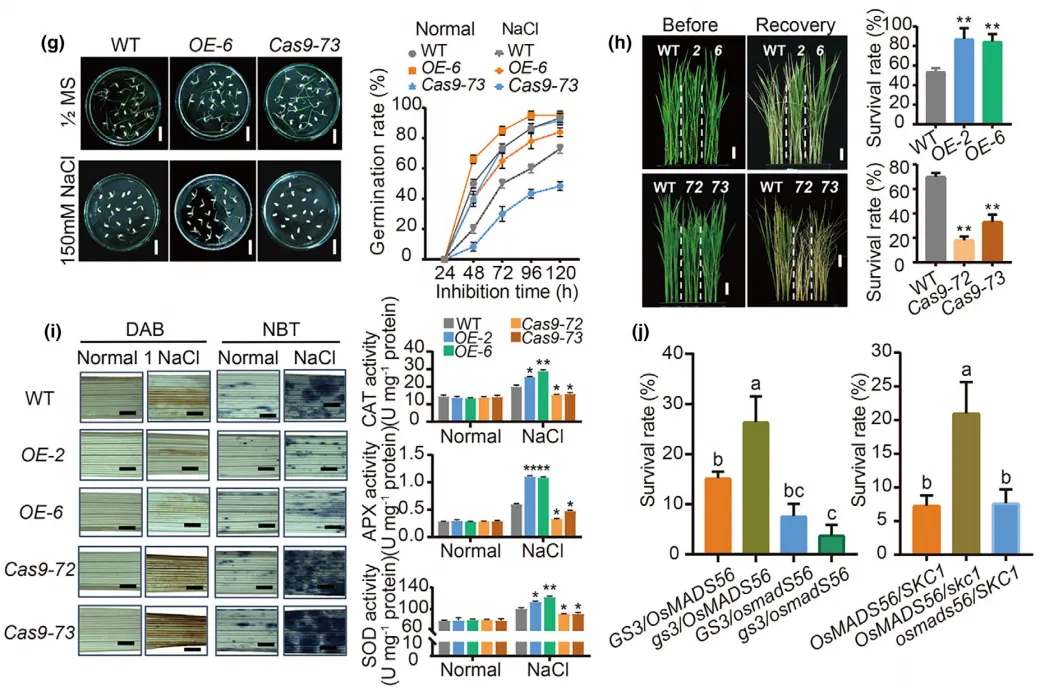
图1 基于结构变异挖掘耐盐基因OsMADS56及其耐盐分子机制
利用盐胁迫下特异性响应的PAV-eGene结合群体水平的差异表达基因集,挖掘了一个位于OsMADS56基因上的PAV。这个PAV的存在导致了起始密码子ATG和第一外显子的缺失,从而降低了该基因的耐盐性。通过该基因的近等基因系、基因编辑突变体和过表达材料的耐盐性分析,表明OsMADS56基因在响应盐胁迫上发挥正向调控作用,并通过协调抗氧化酶活性调节体内活性氧的积累影响耐盐性。单倍型分析发现,在大多数耐盐品种中检测到1.0 Kb的存在-缺失变异,表明该PAV等位基因在水稻耐盐性中发挥了重要作用。另外,该基因优异的单倍型耐盐性与其他关键耐盐基因STG5、SKC1表现出加性效应,为后续水稻耐盐模块耦合设计育种以应对盐胁迫提供了参考。
该研究利用水稻图形超级泛基因组结合转录组学技术,为鉴定具有功能的PAV-eQTL提供了有效的方法,这一技术使得过去难以发现的耐盐相关的PAV变异得以揭示,为耐盐基因挖掘和耐盐优异种质资源的创新利用提供了新的解决方案。同时为耐盐水稻的多基因聚合策略提供了全新的见解,使得高效、精准的水稻耐盐定向改良成为可能。这一成果将有助于提升水稻品种的耐盐性和推动水稻耐盐全基因组设计育种。





发表评论
想参加讨论吗?请尽情讨论吧!