季度回顾|希望组第二季度客户项目成果盘点
发表期刊:Nature genetics (IF=31.7)
发表日期:2025.04
通讯作者:潍坊现代农业山东省实验室/北京大学现代农业研究院和小麦育种全国重点实验室的邓兴旺院士、何航研究员、李博生研究员为论文共同通讯作者。
该研究首次成功绘制了六倍体小麦的端粒到端粒(T2T)完整基因组图谱。通过采用PacBio HiFi和ONT超长测序技术,结合Hi-C、Illumina和Bionano数据,通过混合组装策略,成功构建六倍体小麦(CS-IAAS)端粒到端粒(T2T)无缺口基因组。其大小为14.51 Gb,contig N50达到723.78 Mb,包含21条染色体、42个端粒和21个着丝粒。新组装版本CS-IAAS填补了CS RefSeq(v2.1)中565.66 Mb的缺口,包括着丝粒卫星序列(16.05%)、转座元件(68.66%)、rDNA阵列(0.75%)等。这一成果不仅深化了对小麦基因组结构和进化机制的理解,还为解析其他复杂多倍体作物基因组提供了范例。未来,依托这一高质量参考基因组,科学家将更精准地挖掘与产量、品质、抗病性相关的关键基因,为小麦品种改良带来革命性突破。
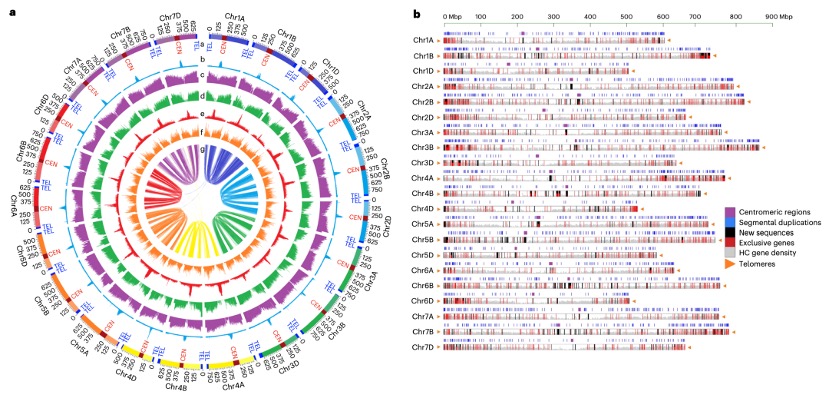
图1 六倍体小麦(CS-IAAS)完整基因组组装
Gap-free telomere-to-telomere assembly of the Mangifera persiciforma genome and its evolutionary insights on resistance
发表期刊:Plant Biotechnology Journal(IF=11.2)
发表日期:2025.05
通讯作者:广西大学李柏君副教授和谢亮助理教授为论文通讯共同作者
该研究首次绘制了天桃木(Mangifera persiciforma)无间隙端粒到端粒(T2T)基因组图谱,是杧果属植物中的首个T2T基因组。研究发现,基因家族的扩张与收缩与物种性状的增强或减弱密切相关,天桃木中扩张的基因家族主要与环境适应性通路相关,有利于植物对生物和非生物胁迫的抗性。此外发现TPS基因在天桃木进化过程中异常活跃,其扩张更倾向于通过近端复制和串联复制实现,而非全基因组复制。比较基因组分析发现天桃木有特有1009个基因,且主要富集在与植物-病原体互作相关的环境适应通路中。进一步通过组织特异性表达分析筛选出19个高表达基因,其中17个属于NLR基因家族。这些结果表明天桃木在进化和环境适应过程中的独特机制,也为芒果抗性育种提供了潜在靶点。
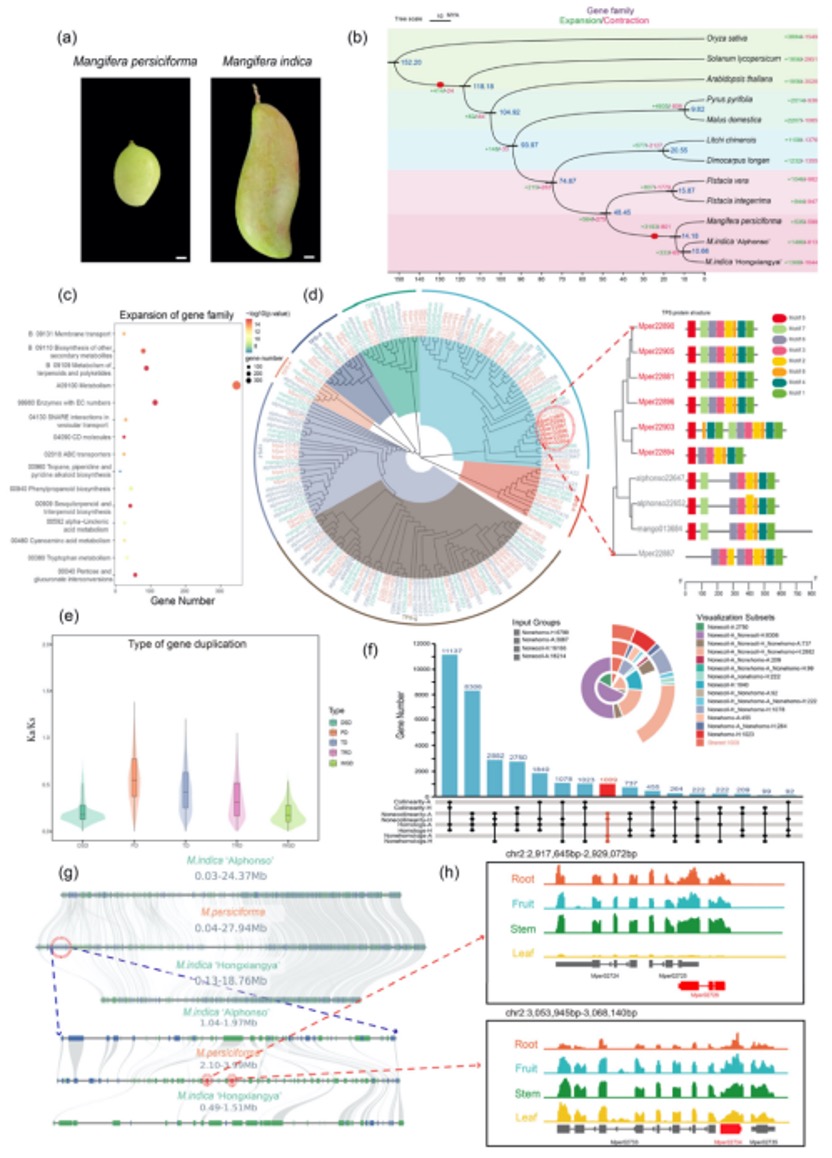
图2 天桃木T2T基因组解析
发表期刊:Nature (IF=50.5)
发表日期:2025.04
通讯作者:中国科学院分子植物科学卓越创新中心韩斌院士和赵强研究员为论文共同通讯作者
普通野生稻(Oryza rufipogon)作为亚洲栽培稻(Oryza sativa)的野生祖先,是水稻育种的重要资源。为了全面捕捉栽培稻和野生稻的固有遗传多样性,该研究首次完成了145份具有地理和遗传多样性材料(包含129份普通野生稻和16份栽培稻)的高精度基因组组装,绘制了迄今为止分辨率最高的“野生稻-栽培稻泛基因组图谱”。构建的泛基因组包含69,531个基因,其中发现13,728个为野生稻特有基因。研究发现相较栽培稻,野生稻蕴含丰富的抗病资源。群体结构分析表明,粳稻最早由中国南方Or-IIIa群体驯化,向东南亚扩散后与当地Or-Ia和Or-Ib杂交形成籼稻和aus稻,南亚栽培稻频繁杂交产生intro-indica(籼稻×aus稻),intro-indica与粳稻杂交形成basmati稻。这些结果强有力支持亚洲栽培稻单次驯化假说。该成果不仅为水稻基因组辅助育种提供了前所未有的遗传资源,为培育抗病耐逆、适应气候变化的优质水稻品种奠定了坚实的科学基础。
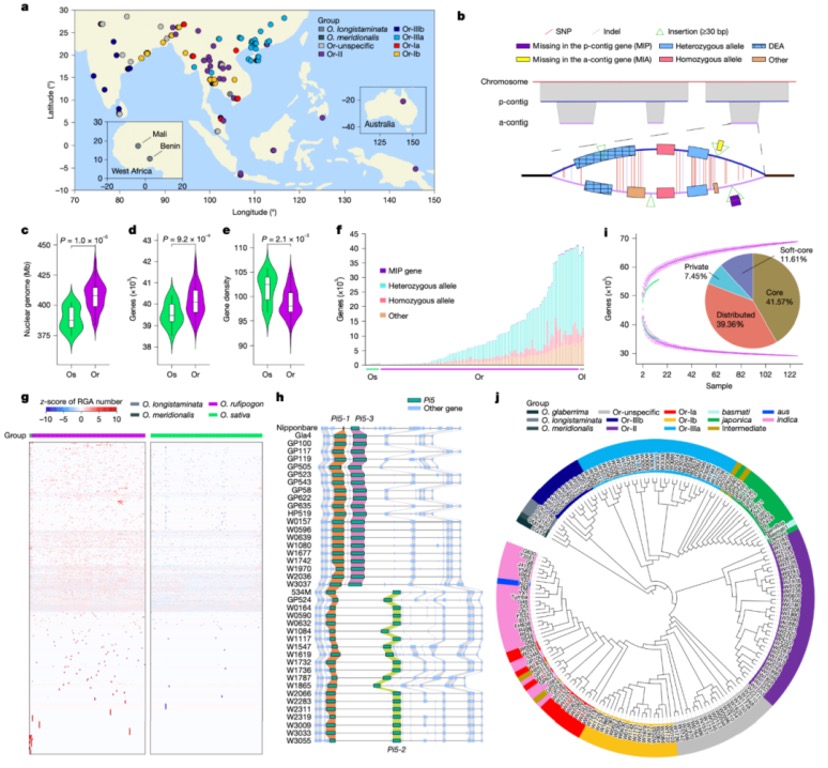
图3 149个代表性野生和栽培水稻品种及泛基因组构建分析
Pangenome analysis reveals structural variation associated with seed size and weight traits in peanut
发表期刊:Nature Genetics (IF=31.7)
发表日期:2025.04
通讯作者:河南农业大学殷冬梅教授、上海交通大学韦朝春教授和澳大利亚莫道克大学Rajeev K. Varshney院士为论文共同通讯作者
花生(Arachis hypogaea L.)作为一种重要的油料与经济作物,其种子大小和重量是驯化与育种的关键性状。该研究通过整合8个代表性品种高质量基因组及269份不同籽粒大小和重量的重测序数据,开展了全面泛基因组分析。研究鉴定出22,222个核心或软核心基因家族、22,232个分布基因家族和5,643个私有基因家族。发现A亚基因组SV频率高于B亚基因组,并鉴定出1,335个与驯化相关的SVs和190个与籽粒大小和重量相关的SVs。特别是挖掘出AhCKX6和AhARF2-2基因的结构变异与籽粒重量和大小的关系,为未来花生基因组学辅助改良育种提供了理论基础和新思路。
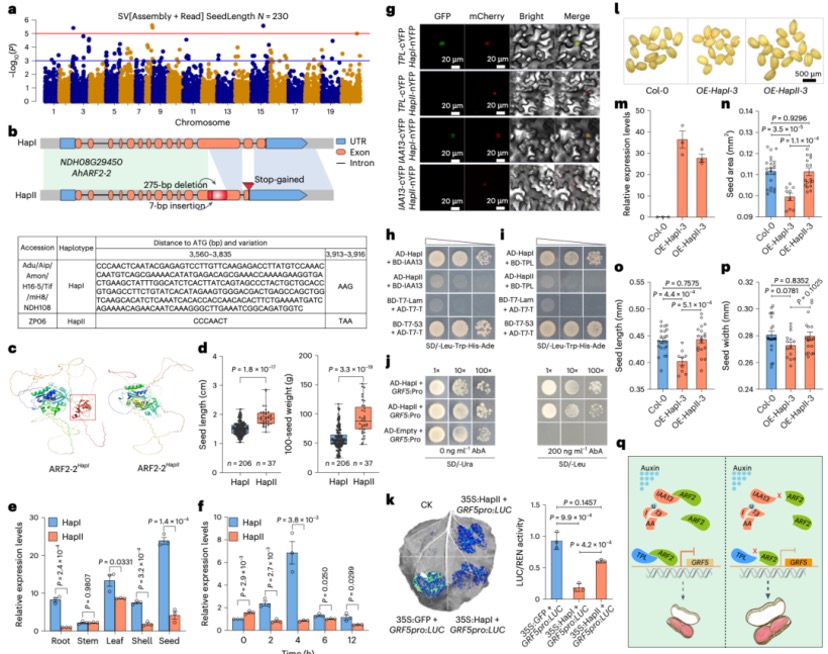
图4 AhARF2-2负调控种子大小
发表期刊: Genome Biology(IF=10.1)
发表日期:2025.04
通讯作者:中国科学院动物研究所胡义波研究员和中国科学院动物研究所/江西农业大学魏辅文院士为论文共同通讯作者
该研究基于Nanopore、Hi-C及Illumina测序技术成功构建了一只野生雌性雪豹的染色体级别基因组及52份野生雪豹全基因组数据进行全面的关于雪豹种群基因组学研究。研究发现全球雪豹可以划分为两个主要遗传谱系,即北部谱系和南部谱系,这一发现得到了生物地理学证据的支持。末次盛冰期促使两个谱系发生分化,而喜马拉雅山脉中段与西段间的微气候差异及大型河流很可能是维持谱系分化的关键因素。南方谱系中EPAS1 基因的两个错义突变在南部谱系中几乎固定,且等位基因频率随海拔升高而增加。与南方谱系相比,北方谱系表现出更低的基因组多样性,但近亲繁殖水平更高,遗传负荷也更重,这与其近期的种群数量下降一致。研究还发现雪豹具有极低的基因组多样性,其近交程度高于其他食肉目物种;但历史种群瓶颈和近亲繁殖过程有效清除了雪豹群体中的强有害突变,这可能是该物种种群存续的重要遗传机制,也为雪豹保提供了重要的理论基础。
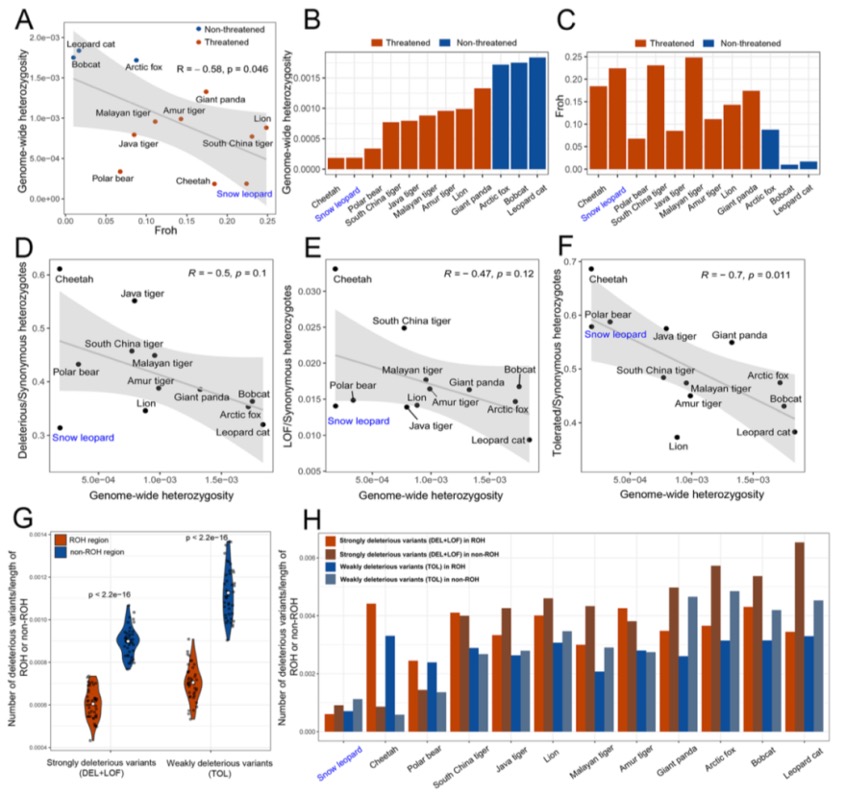
图5 食肉目种间比较揭示雪豹低遗传多样性背景下的有害变异清除机制
Vitellogenin receptor mediates heat adaptability of oocyte development in mud crabs and zebrafish
发表期刊:Nature Communications(IF=14.7)
发表日期:2025.04
通讯作者:中山大学何建国教授和王牧骅副教授为论文共同通讯作者
气候驱动的变暖影响着卵生变温动物的繁殖。然而,卵生变温动物是否具有卵黄发生的高温保护机制,以及该保护机制的遗传基础仍未可知。研究发现,在高温环境下,多数雌性拟穴青蟹能形成发育良好的卵巢,但少数个体会出现卵母细胞发育障碍。为探究青蟹卵母细胞发育的高温保护机制,该研究构建了该物种染色体级别基因组,并鉴定出卵黄蛋白原受体(VtgR)基因的一个增强子,该增强子能在高温条件下激活基因表达。异常个体因内含子缺失导致该增强子缺失,使得VtgR表达水平低下,最终造成这些个体在高温胁迫下卵黄发生异常。此外,研究在斑马鱼中也发现了类似的高温保护机制,破环斑马鱼卵黄蛋白原受体基因Lrp13会导致所有敲除个体在高温胁迫下卵黄发生异常。这项研究发现卵生变温动物也具有卵巢发育高温适应的遗传机制,这一发现对理解卵生变温动物如何适应气候变暖具有重大意义。
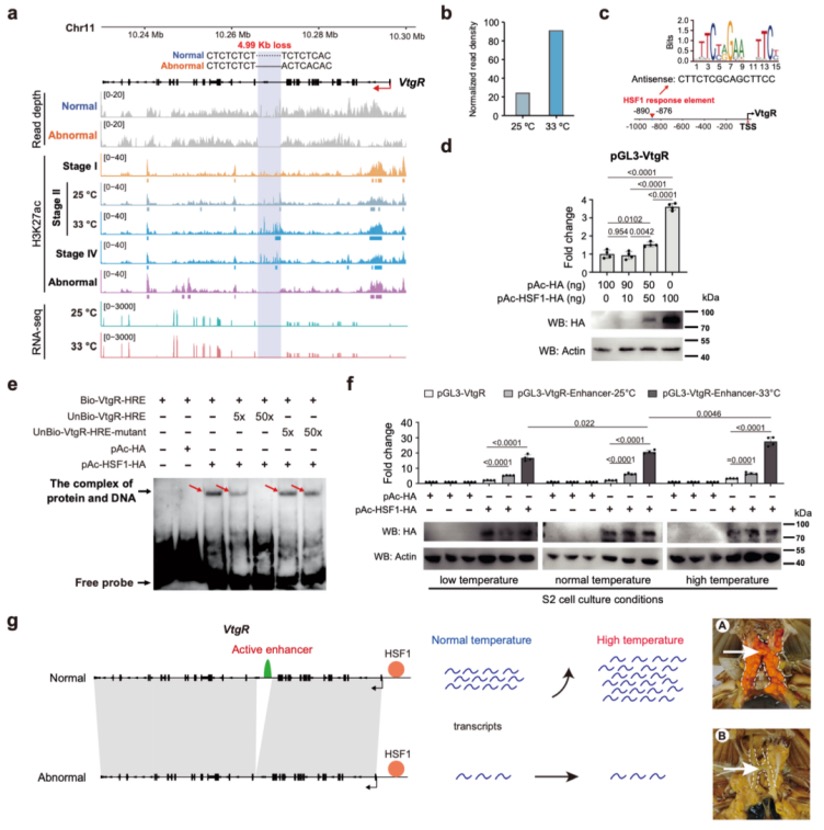
图6 VtgR介导拟穴青蟹的卵黄发生高温保护机制
发表期刊:Science(IF=44.7)
发表日期:2025.05
通讯作者:中国科学院分子植物科学卓越创新中心詹帅研究员为论文通讯作者
该研究以受温度诱导的兼性滞育品系(二化,bi-voltinism)和非滞育品系(多化,poly-voltinism)为亲本构建作图群体,将决定滞育变异的主效基因座定位在Z染色体末端。研究结合255个具有不同化性特征的家蚕品系进行全基因组关联分析(GWAS),进一步将关键变异位点定位在该区域内的核心生物钟基因Cycle(脊椎动物BMAL1的昆虫同源基因)的5′端。研究发现家蚕Cycle基因通过可变剪接编码3个亚型(Isoforms A-C);相较于滞育品系,非滞育品系在C亚型特异的N端外显子上存在1-bp的碱基缺失,导致该亚型发生整体性移码突变,但不影响其它亚型(A/B)的编码。通过开展基因编辑、生理生化实验、共表达网络分析、行为学实验等,研究人员证明CYC-C亚型可能作用于GABAergic-CRZ-DH信号通路调控家蚕滞育的环境诱导,而A/B亚型发挥CYC本身的昼夜节律调控功能。此外,研究人员还发现这些功能分化的CYC亚型以相对保守的形式普遍存在于鳞翅目昆虫中,并进一步在亚洲玉米螟(Ostrinia furnacalis)中验证了CYC-C对其幼虫滞育(主要受光周期诱导)的调控作用,从而提示了CYC-C对鳞翅目昆虫滞育调控的普适作用。该研究揭示的分子机制为基于遗传学手段打破滞育、高效利用资源昆虫,以及在全球变暖大背景下预测昆虫种群潜在动态变化等提供理论依据。
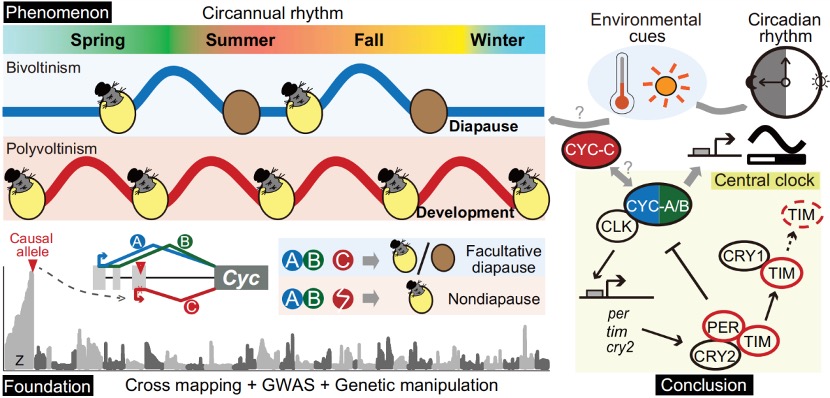
图7 生物钟基因Cycle通过不同亚型的功能多态性同时调控昼夜节律和季节节律
发表期刊:BMC Genomics(IF=3.5)
发表日期:2025.05
通讯作者:云南农业大学动物科学技术学院赵桂英教授和霍金龙教授为论文通讯作者
该研究通过整合Illumina和Nanopore单细胞RNA测序技术,绘制了保山猪睾丸单细胞全面的转录图谱。研究鉴定出9个显著不同的细胞群及确定了保山猪睾丸中精子发生过程中的6个明确发育阶段:精原细胞(SPG)、早期精母细胞(Early SPC)、晚期精母细胞(Late SPC)、球形精子细胞(RS)、长型精子细胞(ES)、成熟精子(Sperm)。研究鉴定出8条关键信号通路并发现六种生殖细胞类型之间存在显著通讯网络。此外,长读长测序技术进一步捕获了RNA转录本的完整性和多样性,其中约60%为新型异构体,且FSM异构体在转录本长度、编码序列长度、开放阅读框长度和外显子数量等方面均表现出优势。该研究为理解猪精子发生的分子机制提供了新的视角。有助于推动猪繁殖技术的发展,为提高保山猪的繁殖效率提供理论依据,对保护和利用这一珍贵的种质资源具有重要意义。
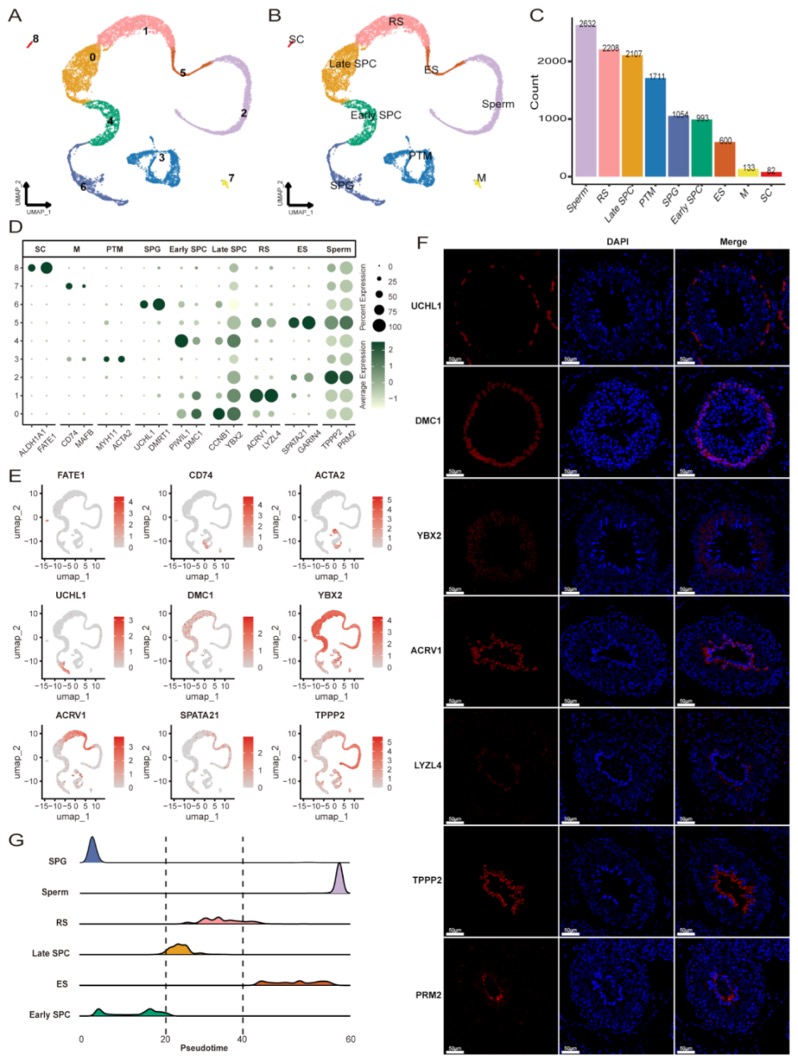
图8 保山猪睾丸组织细胞类型分析
1. Liu S, Li K, Dai X, et al. A telomere-to-telomere genome assembly coupled with multi-omic data provides insights into the evolution of hexaploid bread wheat[J]. Nature Genetics, 2025: 1-13.
2. Wu J, Bao R X, Liu Y, et al. Gap‐free telomere‐to‐telomere assembly of the Mangifera persiciforma genome and its evolutionary insights on resistance[J]. Plant Biotechnology Journal, 2025.
3. Guo D, Li Y, Lu H, et al. A pangenome reference of wild and cultivated rice[J]. Nature, 2025: 1-10.
4. Zhao K, Xue H, Li G, et al. Pangenome analysis reveals structural variation associated with seed size and weight traits in peanut[J]. Nature genetics, 2025, 57(5): 1250-1261.
5. Yang L, Jin H, Yang Q, et al. Genomic evidence for low genetic diversity but purging of strong deleterious variants in snow leopards[J]. Genome Biology, 2025, 26(1): 94.
6. Zhang L, Wu K, Li H, et al. Vitellogenin receptor mediates heat adaptability of oocyte development in mud crabs and zebrafish[J]. Nature Communications, 2025, 16(1): 3722.
7. Zheng S, Wang Y, Li G, et al. Functional polymorphism of CYCLE underlies the diapause variation in moths[J]. Science, 2025, 388(6750): eado2129.
8. Lin W, Zhang X, Liu Z, et al. Isoform-resolution single-cell RNA sequencing reveals the transcriptional panorama of adult Baoshan pig testis cells[J]. BMC genomics, 2025, 26(1): 1-17.





