自主创新丨NextDenovo:一种高效且精确的长读长纠错与组装工具
ONT平台的测序可产生>100 Kb的超长reads用于填充基因组组装中串联或高度同源的多拷贝重复区域,但其同时伴随着准确度不高的问题。使用ONT数据组装基因组,有两种常使用策略即“先矫正后组装”(CTA)和“先组装后矫正”(ATC),对于大型植物基因组在组装重复序列时,基于CTA策略通常能产生更准确和连续的组装。对此,希望组自主研发基于ONT数据进行高效纠错和CTA组装的NextDenovo 软件,用于组装出一个完整、准确的基因组。NextDenovo 软件历经多年打磨以及在动植物基因组组装中的成功应用,于2024年4月26日在《Genome Biology》发表题为《NextDenovo:an efficient error correction and accurate assembly tool for noisy long reads》的文章。

NextDenovo 包含五个主要步骤:1、对原始reads进行成对重叠;2、过滤重叠结果避免错误配对以影响纠错的准确性;3、基于过滤后的重叠结果进行纠错;4、需要两步迭代成对矫正reads重叠;5、使用重叠结果构建一个组装图,然后进行图形清理并输出结果。 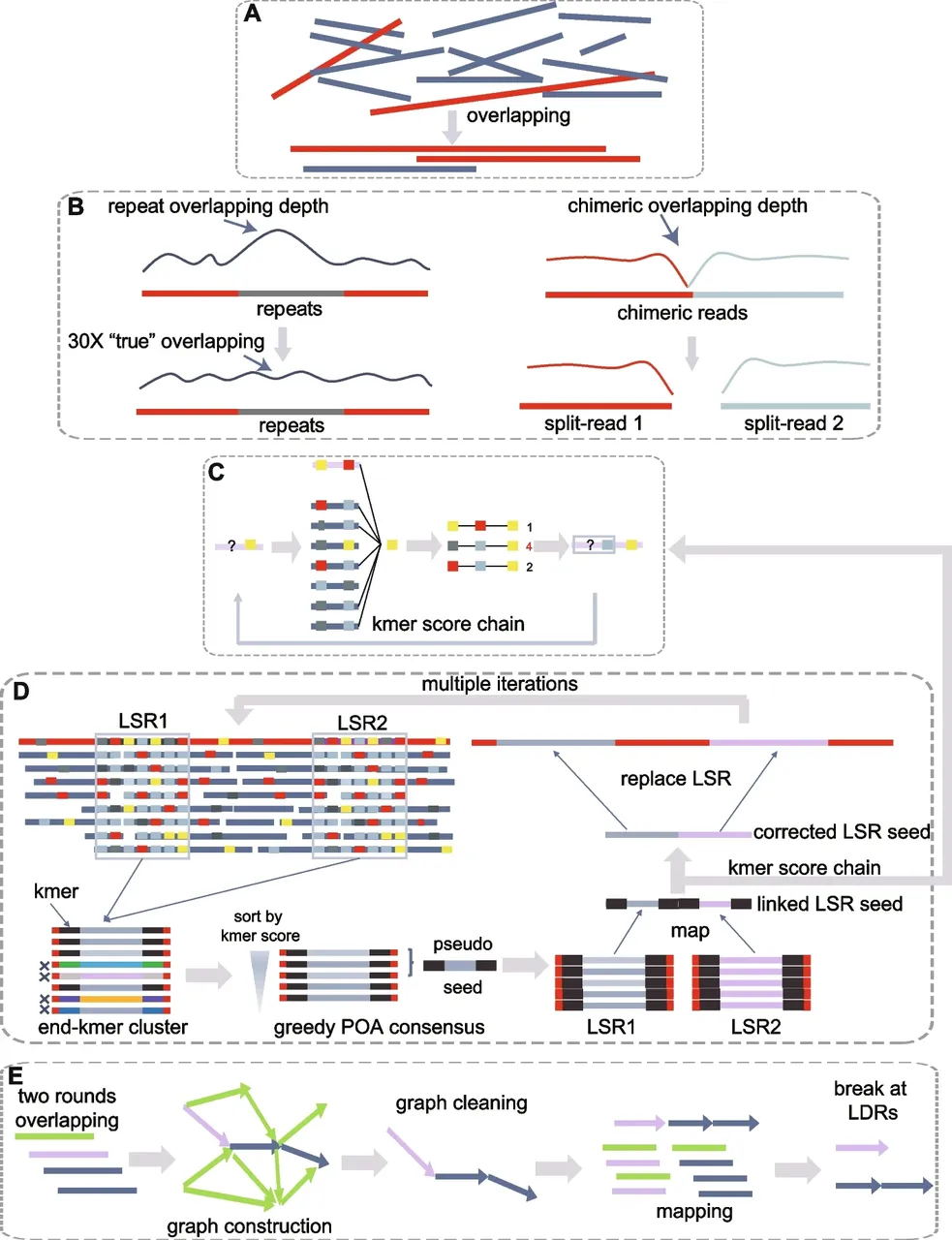
图1. NextDenovo 组装流程图
在纠错速度方面,NextDenovo与 Consent、Canu 和 Necat 相比,在模拟数据上分别快 3.00 倍、7.44 倍和 1.13 倍;在真实数据上则分别快 9.51 倍、69.25 倍和 1.63 倍。
表1. ONT reads纠错统计 
将NextDenovo软件与其他纠错、组装软件在4个非人类基因组(果蝇、拟南芥、水稻、玉米)和35个人类基因组的组装方面进行了比较,结果显示NextDenovo可快速、高效地对ONT数据进行纠错并产生高准确度的基因组组装,特别是对于含有大量重复序列的基因组。 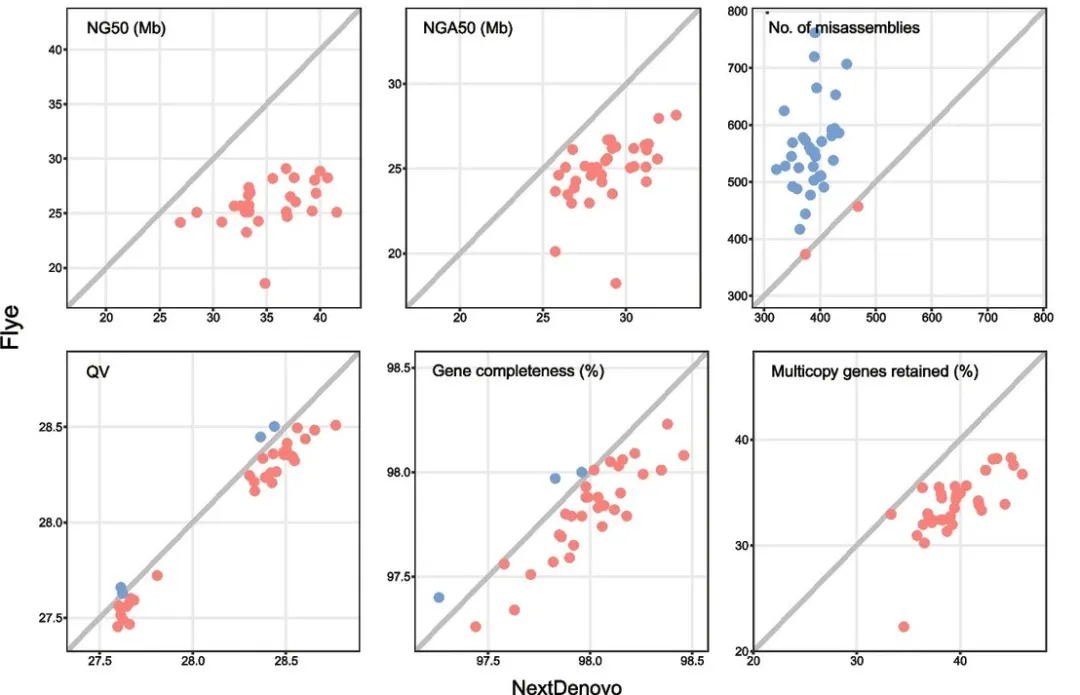
图2. 35个人类基因组的De novo组装
NextDenovo 已成功多次用于大基因组组装,例如约 10.5 Gb 的苏铁(Cycas panzhihuaensis)基因组组装(contig N50 = 12 Mb)、约 10.76 Gb 的六倍体燕麦基因组组装(contig N50 = 75.27 Mb)、约 40 Gb 的非洲肺鱼基因组组装(contig N50 = 1.60 Mb)和约 48 Gb 的南极磷虾基因组组装(contig N50 = 178.99 kb)。
通过使用 ONT超长reads,NextDenovo 可以产生部分或几乎达到染色体水平的组装。在约 4.59 Gb 罂粟基因组中,NextDenovo 使用约 19X ONT超长reads和约 86X ONT常规reads组装了 contig N50 为 65.57 Mb的基因组,最长长度为 178.776 Mb ;类似地,对于 3.69 Gb 的西瓜基因组,NextDenovo 使用约 57X ONT超长reads,组装出11 条最长 contig 表示 11 条染色体;在约 10.76 Gb 的六倍体燕麦基因组中,NextDenovo 使用约 100X ONT超长reads 组装了contig N50 为 75.27 Mb,最长长度为 313.87 Mb的基因组。
总的来说,NextDenovo 是一种针对长读长的高效纠错和组装工具,它可以快速提供高度准确的纠错reads,并从这些reads中产生准确的组装。特别是当使用 ONT 的超长reads进行组装时,NextDenovo 可以生成部分或接近染色体级的组装。ONT测序具有低成本、高通量、周期快的特点,因此NextDenovo 还是一种用于群体规模的ONT长读长测序数据的优秀组装工具。
希望组一直致力于自主创新、开发优质软件以便为客户交付更优质的高质量数据用于后续的科学研究,以助力各位专家学者在基因组学领域取得更多的突破和进展!除NextDenovo外,希望组自研软件NextPolish可高效矫正三代(Nanopore 、Pacbio)下机数据组装得到基因组的单碱基错误,进一步提高单碱基准确性。该工具采用 K-mer 得分链和 K-mer 计数算法,在运行速度、校正精度及消耗资源等方面均优于同类软件,NextPolish 目前已在《Bioinformatics》 期刊正式发表《NextPolish: a fast and efficient genome polishing tool for long-read assembly》。





发表评论
想参加讨论吗?请尽情讨论吧!