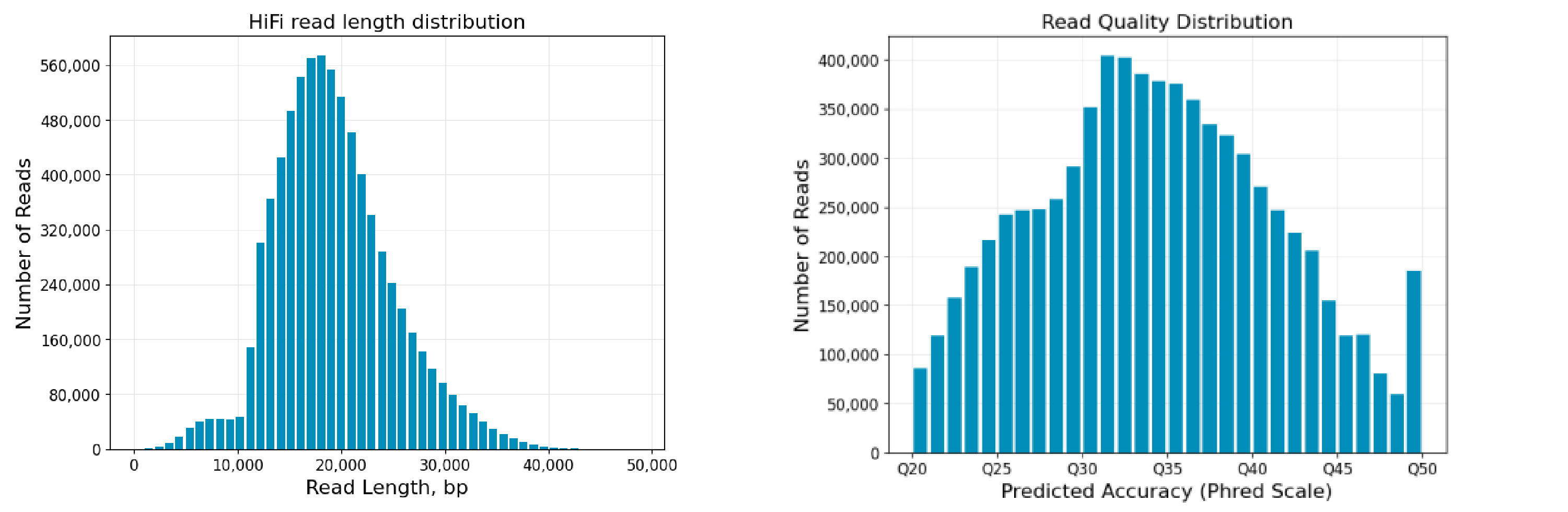
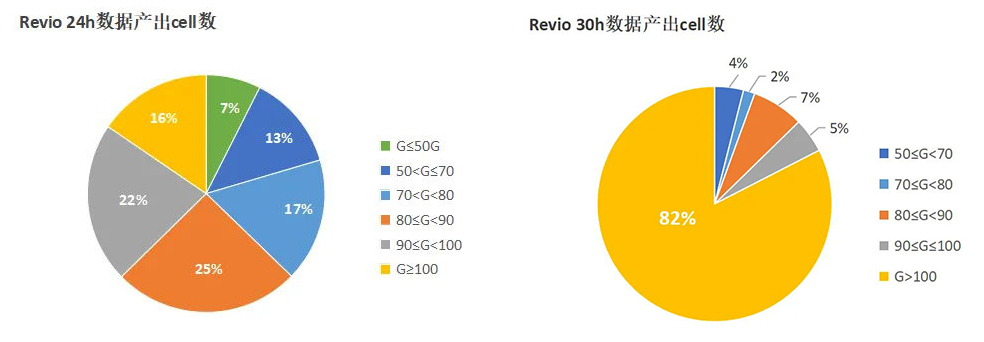
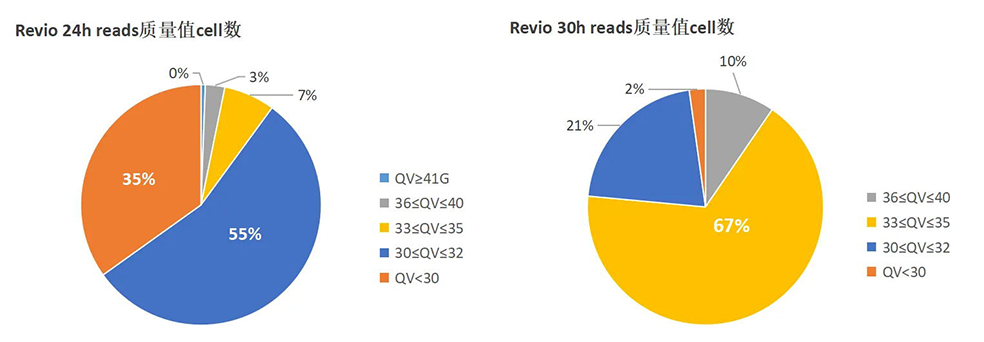
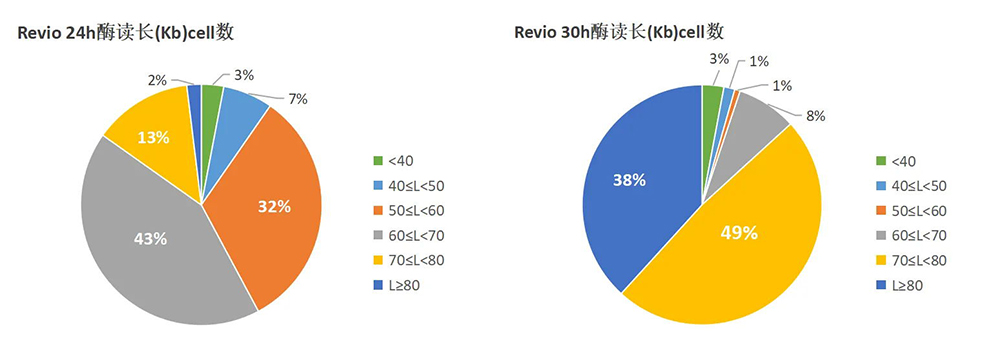
16S 扩增子测序:通过对特定环境中的微生物(细菌)DNA特定区域进行扩增测序,以研究微生物群落组成、物种丰度以及样本间群落组成差异情况。
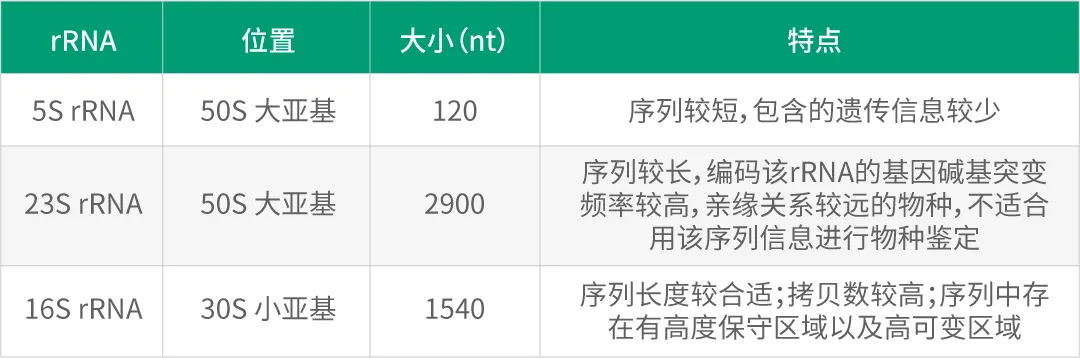
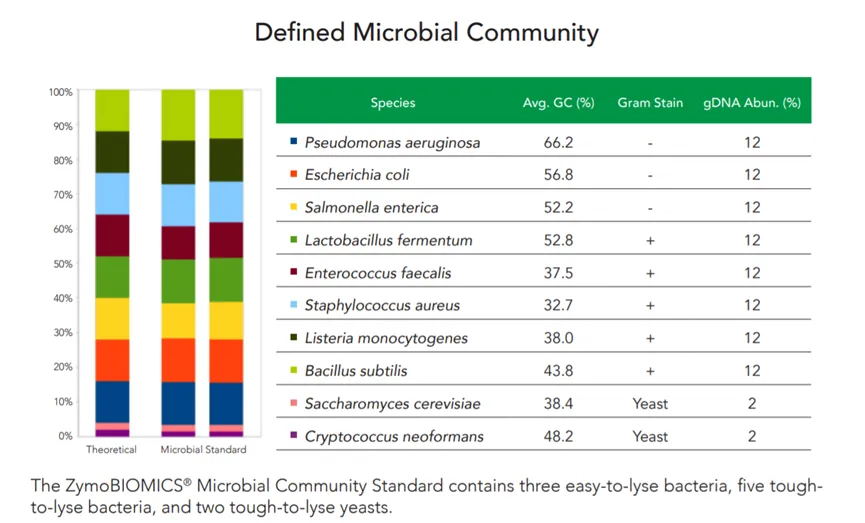
细菌核糖体RNA(rRNA)按沉降系数分为3种,分别为5S、16S和23S rRNA。16S rRNA是细菌核糖体中30S亚基的组成部分,与5S rRNA、23S rRNA相比,其序列长度合适、拷贝数较高以及序列中存在有高度保守区域以及高可变区域。因而,16S rDNA 是目前最适于细菌系统发育和分类鉴定的指标。
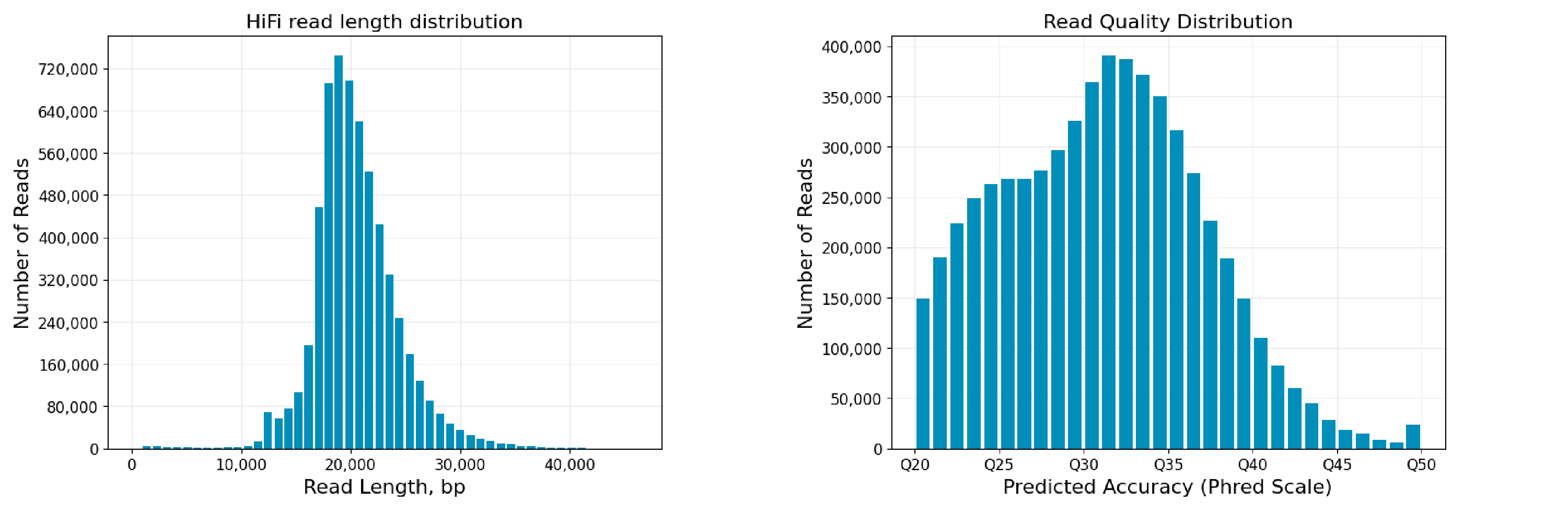
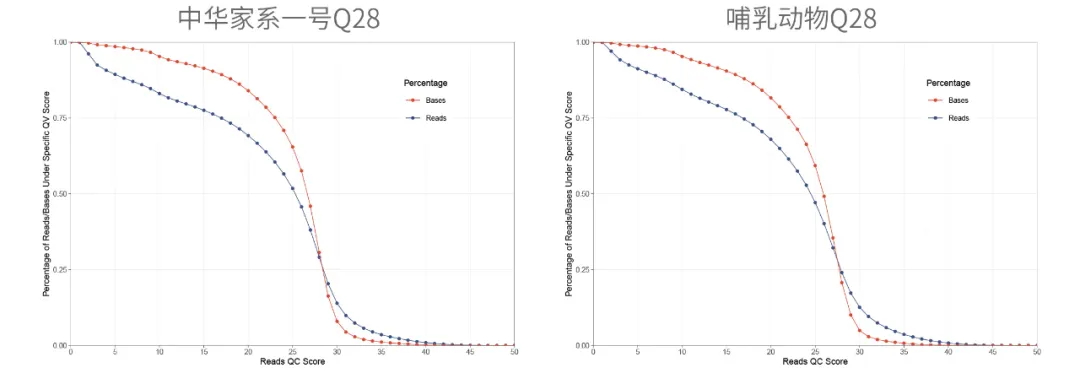
二代扩增子测序因测序读长的限制,无法对原核生物16S中所有可变区进行测序,因此一般只能针对1-2个可变区进行测序分析和物种鉴定。部分高可变区所携带的变异信息有限,无法实现种水平的物种注释,导致二代微生物多样性通常在属及属以上的水平进行研究。而三代扩增子主要基于PacBio Revio测序平台,利用单分子实时测序(SMRT)的方法,基于HiFi模式对全长16S(V1-V9)进行测序,获取更多的高可变区信息以了解样本中的物种组成以及相对丰度等,显著提高物种注释的分辨率和准确性,更全面的反映微生物的群落结构。
全长16S扩增子的产品优势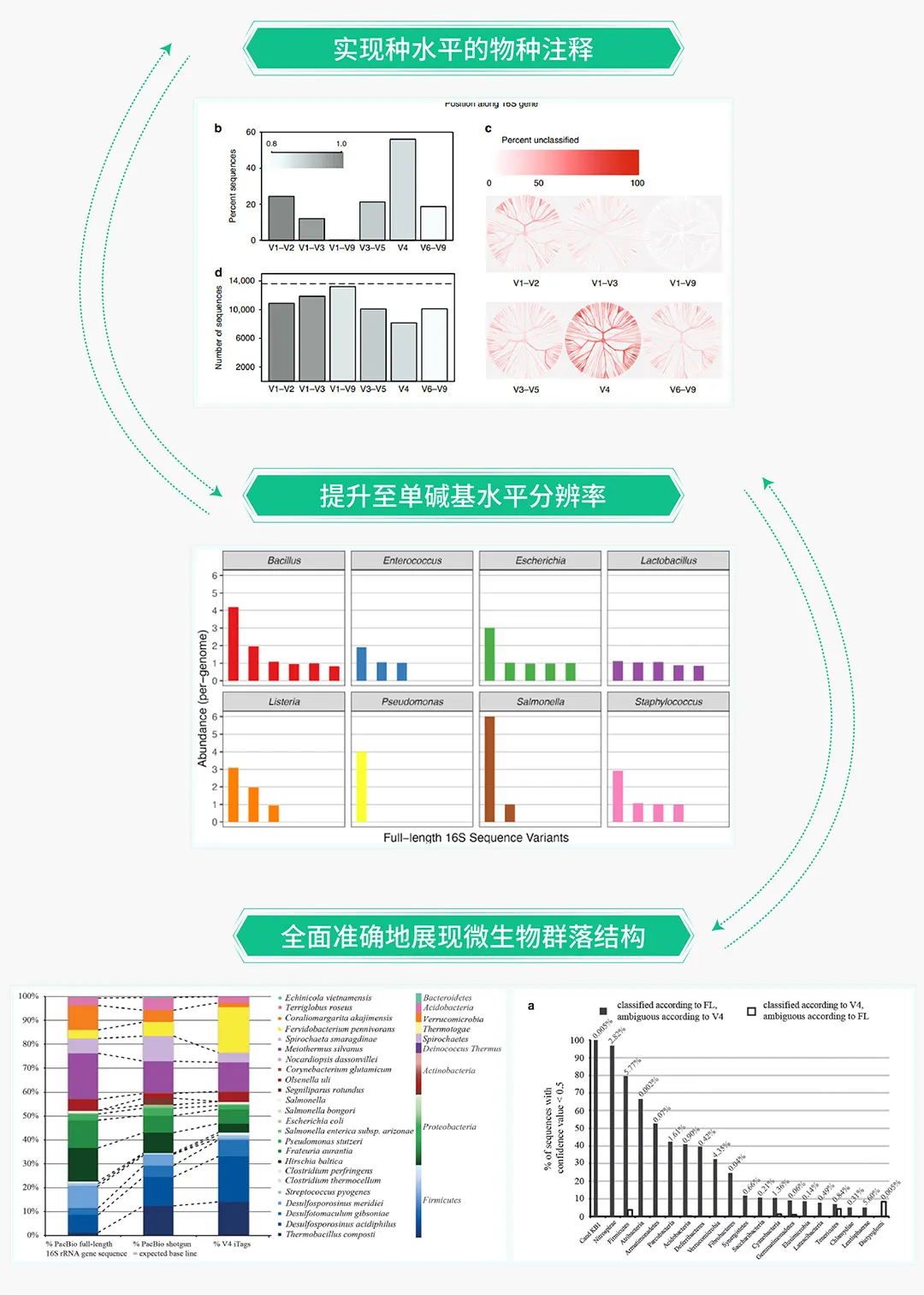
希望组全长16S扩增子全面升级
希望组通过三重技术保障,来提高生产效率、降低生产成本、提高产品精度,从而推动三代扩增子生产的转型升级和高质量交付。
· 自动化生产线设备

希望组通过搭建一整套自动化流程生产线,可实现单日流转样本量>1000个。
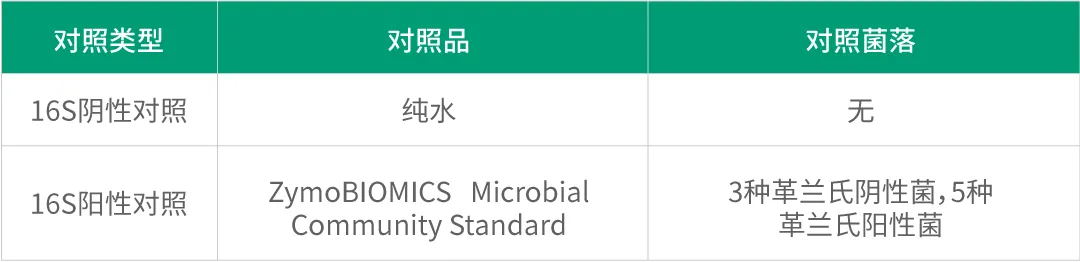
· 阴性和标准品阳性质控
希望组通过每个生产批次独立匹配阴性对照和阳性对照,可以有效地监控实验过程的各个环节,确保数据的准确性和可靠性。其中阴性对照用于检测实验过程中是否存在非特异性扩增或污染,而阳性对照则用于验证实验条件和扩增效率是否正常。
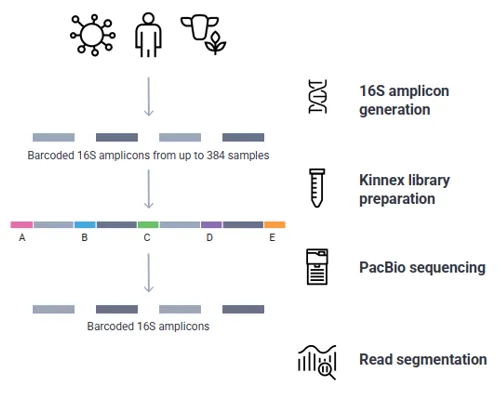
· 串联建库测序流程
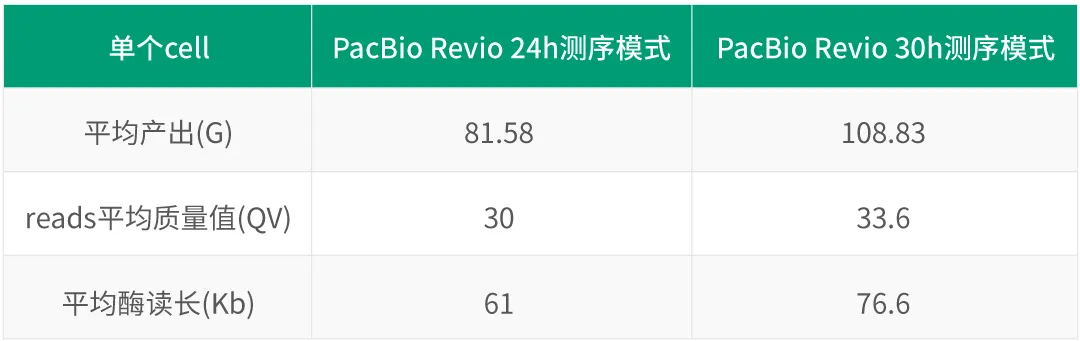
PacBio Revio测序平台的全长16S扩增子是基于Kinnex 16S rRNA试剂盒的方法,首先对扩增产物添加特异性barcode序列,将多样本进行混样,可混样的样本数高达384个。kinnex试剂盒还可以将1.5K大小的扩增产物短片段串联成15K以上的长片段,用这种方法构建的文库,可以适配Revio的HiFi测序模式,更具成本效益。
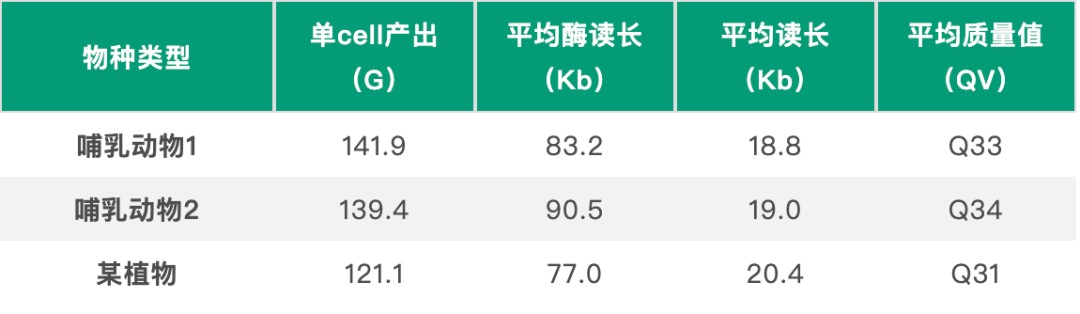
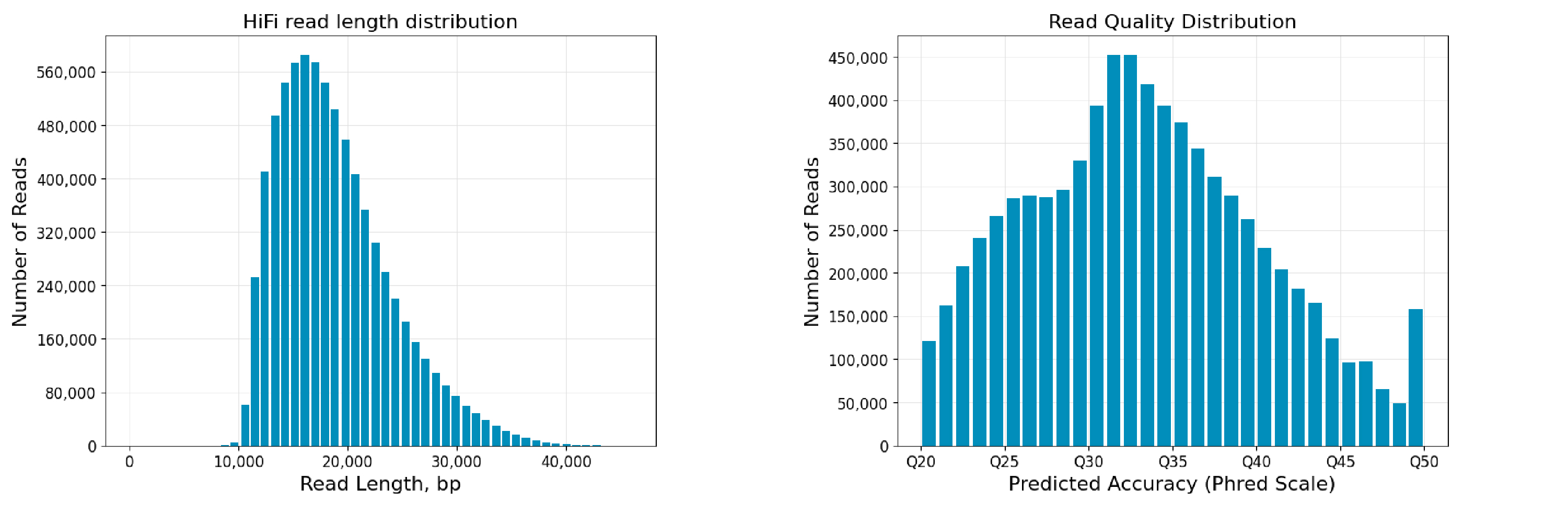
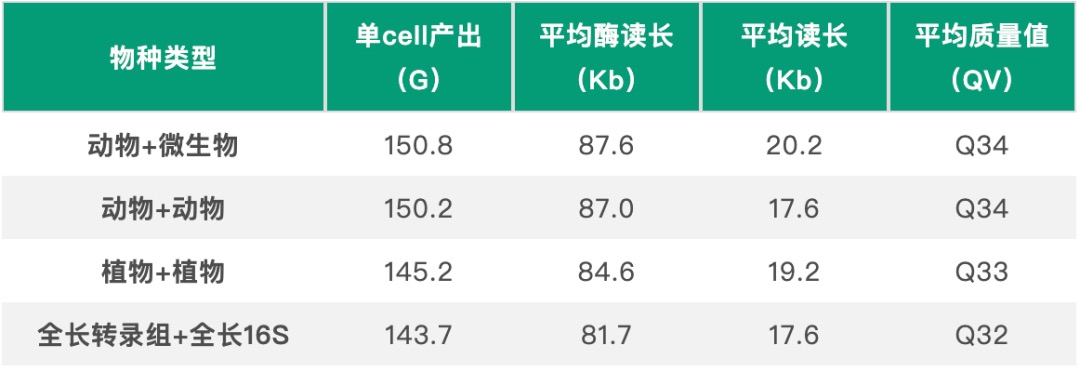
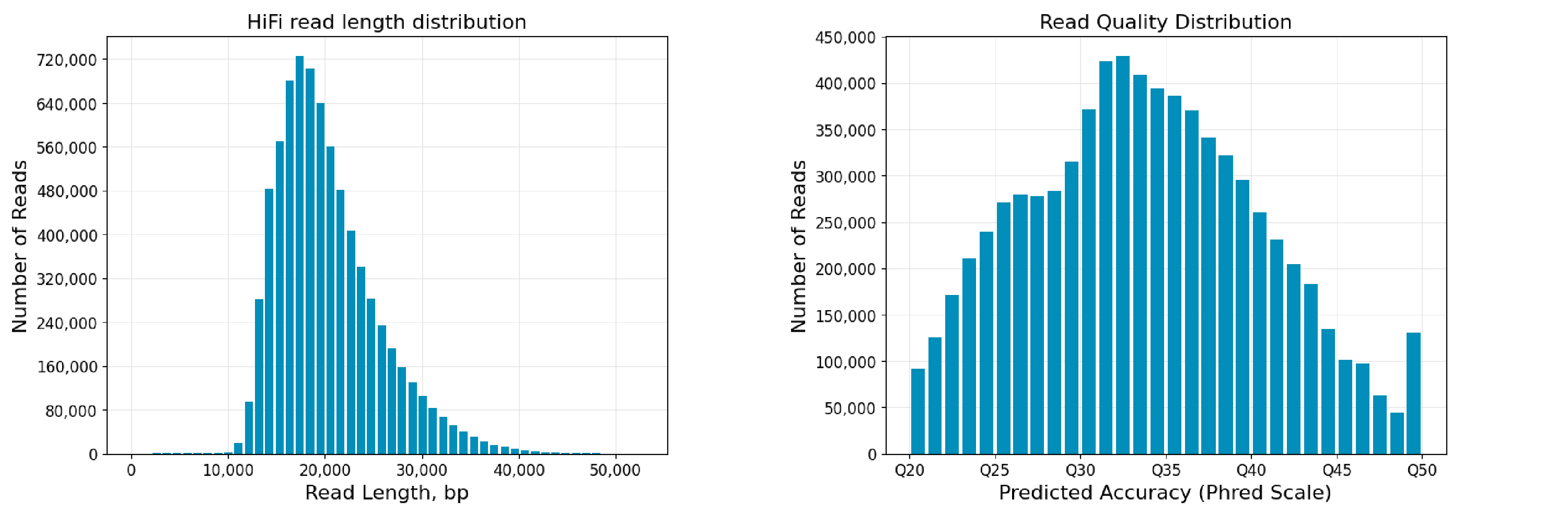
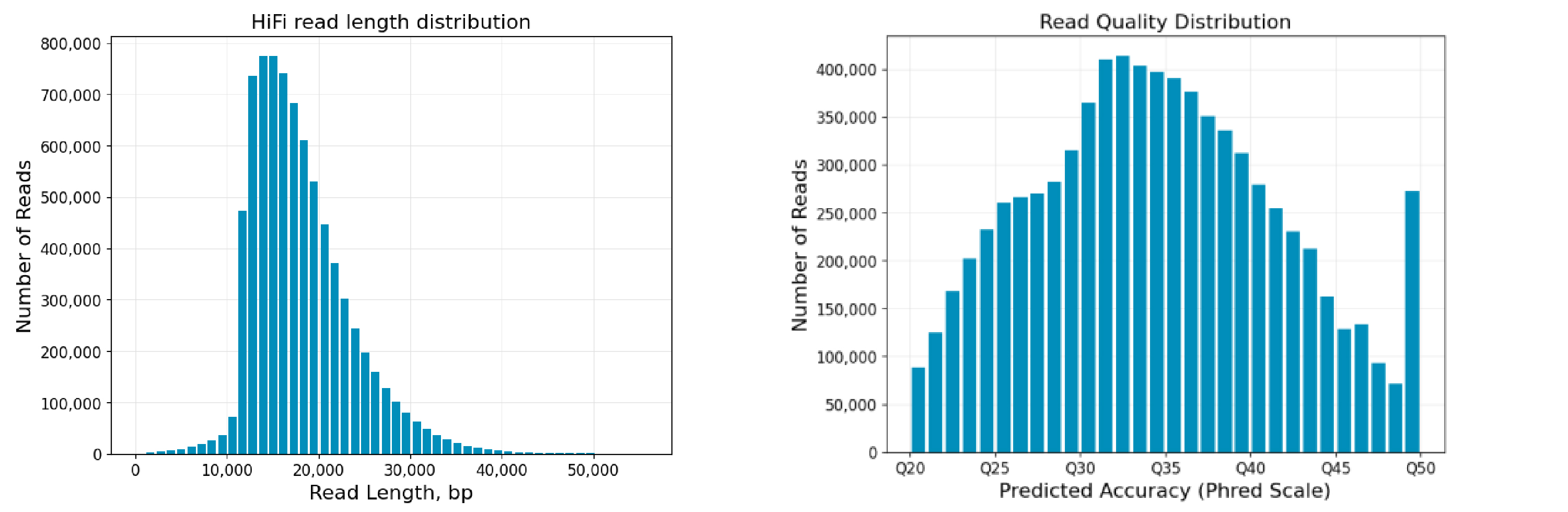
希望组实测数据
· 不同样本类型在不同测序深度条件下检出的物种数量
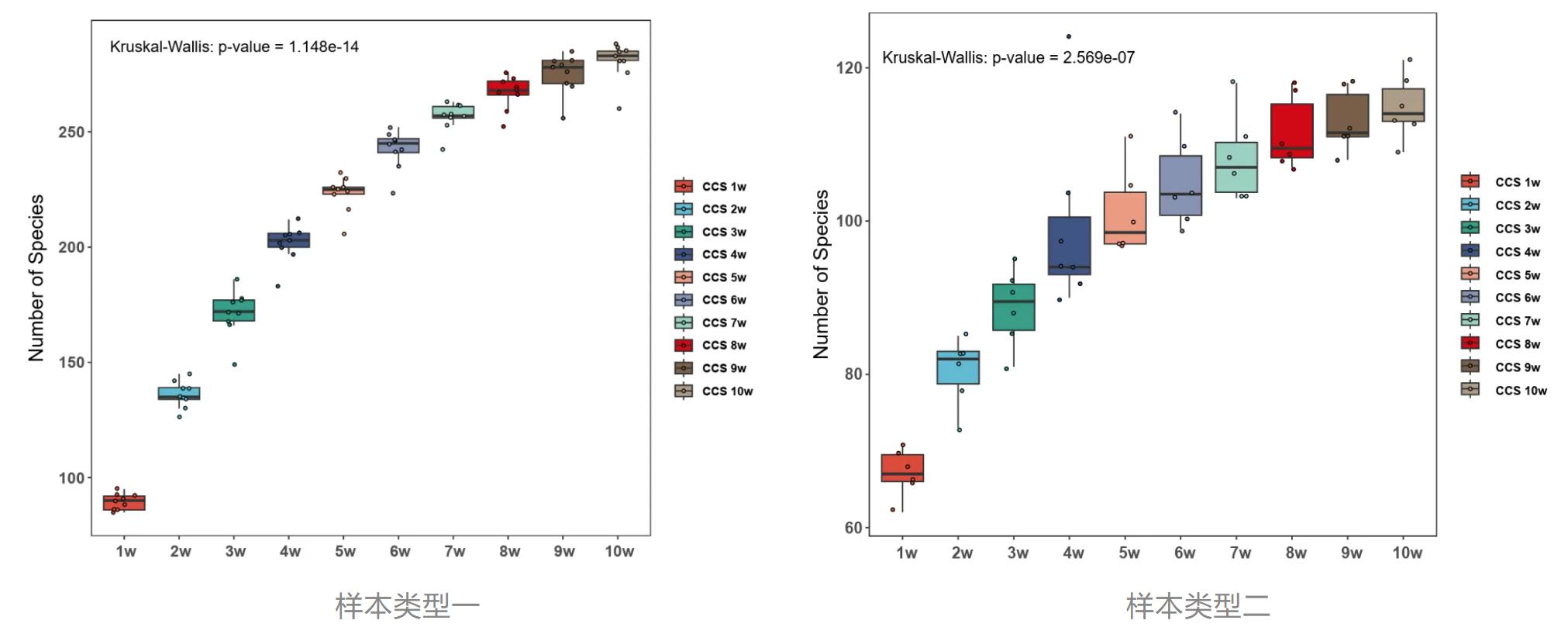
希望组选取不同的样本类型开展梯度测试,结果发现:随着测序深度不断提升,样本中的物种检出数量逐步增加,在10w CCS的测序深度下物种检出数量基本达到饱和。希望组建议:对于中高丰度物种,5w CCS的测序深度可基本满足科研工作者的需求。若您关注低丰度物种的检出,推荐10w CCS的测序深度。应用方向
三代全长16S测序技术的应用范围极为广泛,在基础科研、环境监测、工农业生产等领域展现其独特的优势!
01.环境检测
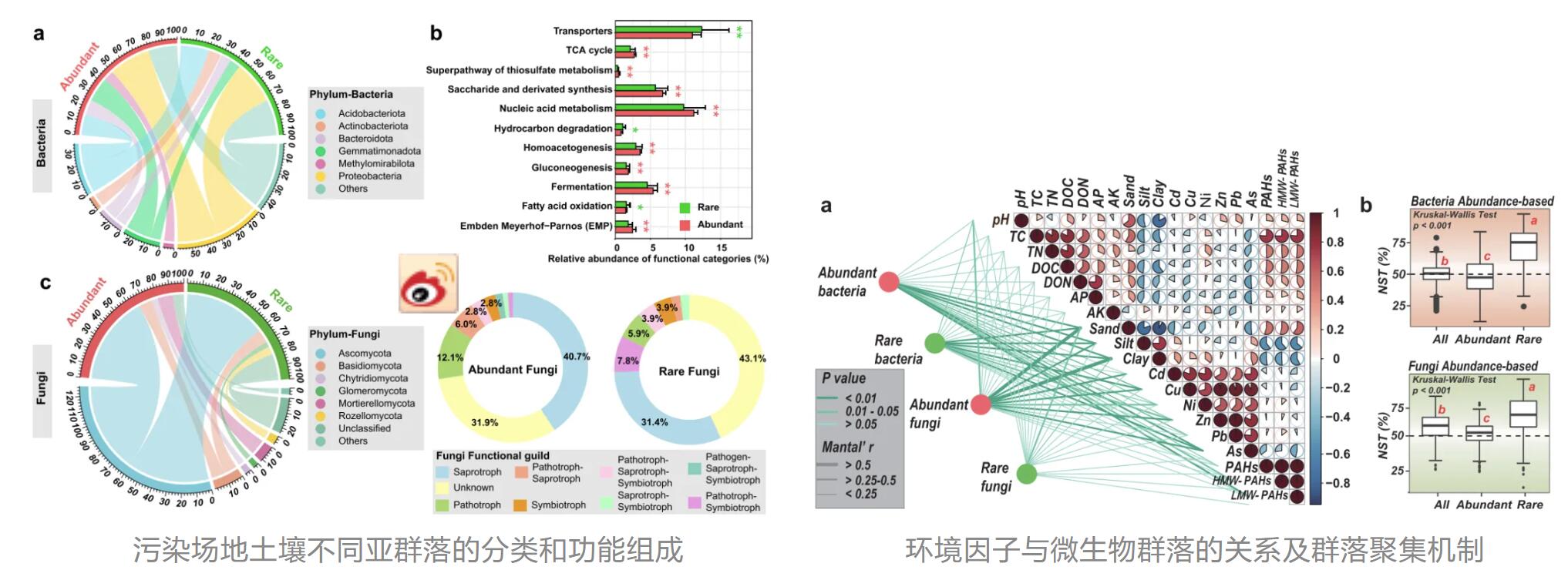
题目:Abundant fungi dominate the complexity of microbial networks in soil of contaminated site: High-precision community analysis by full-length sequencing
期刊:Science of The Total Environment
影响因子:IF=10.753
发表时间:2023年2月
样本类型:土壤
该研究对鞍山和台州的6个土壤样本同时进行16S、ITS全长测序以及16S V3-V4、ITS短读长测序,在高分辨率条件下解析受污染土壤中不同微生物类群的群落组成和生态状况。结果表明全长16S rRNA基因测序在所有水平上都能提供更好的细菌鉴定分辨率,在某些样品中的真菌鉴定上没有显著差异。丰富的分类群对于由全长和短读长测序数据构建的微生物共生网络至关重要。上述研究发现有助于了解土壤生态系统中的生态机制和微生物相互作用,并证明全长测序有可能提供更多微生物群落的细节。
02.工农业生产
题目:Correlation between microbial diversity and flavor metabolism in Huangshui: A by-product of solid-state fermentation Baijiu期刊:LWT
影响因子:6.056
发表时间:2023年4月
样本类型:发酵产物
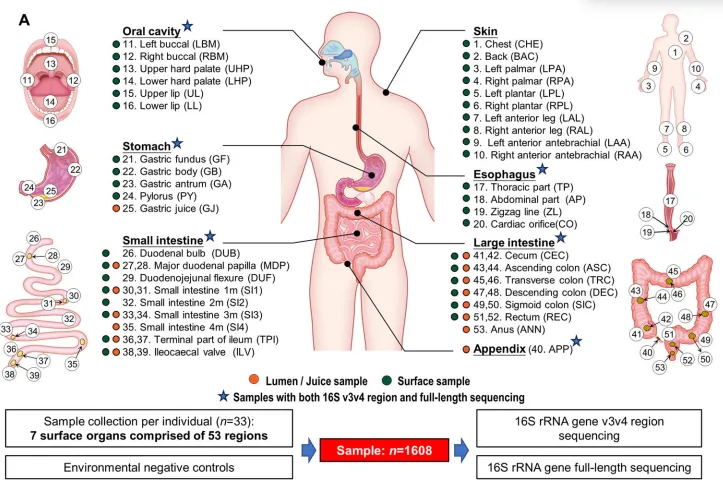
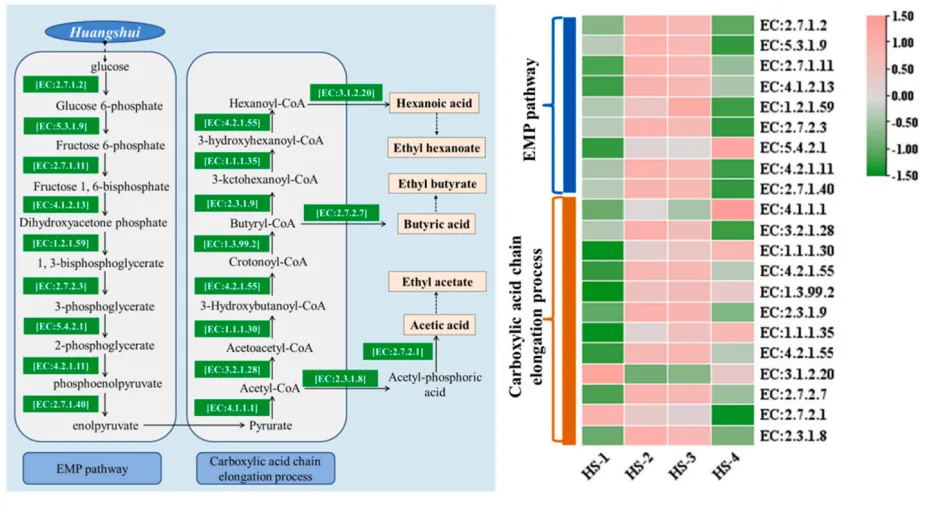
黄水(HS)是固态发酵白酒的副产品,因其丰富的微生物和营养资源而受到更多的关注。本研究揭示了不同HS样品中微生物群落与风味物质的相关性。乳酸杆菌、甲烷杆菌、甲烷杆菌、甲烷菌和甲烷肉杆菌是所有样品的优势属。LEfSe表明,33种风味化合物主要包括己酸、丁酸、乙酸和己酸乙酯,在样品间差异显著,且这些化合物与瘤胃球菌科、拟杆菌科和产甲烷古菌呈正相关(RDA)。此外,PICRUSt2观察了编码酶的丰富基因参与糖酵解途径和羧酸链伸长过程,参与了己酸和丁酸的形成。研究结果为HS中微生物群及其风味代谢提供了科学依据,为合理有效利用HS微生物功能奠定了理论基础。03.器官组织微生态题目:Defining the biogeographical map and potential bacterial translocation of microbiome in human ‘surface organs’
期刊:Nature Communications
影响因子:IF=16.6
发表时间:2024年1月
样本类型:消化系统表面器官(腔和黏膜)和皮肤组织
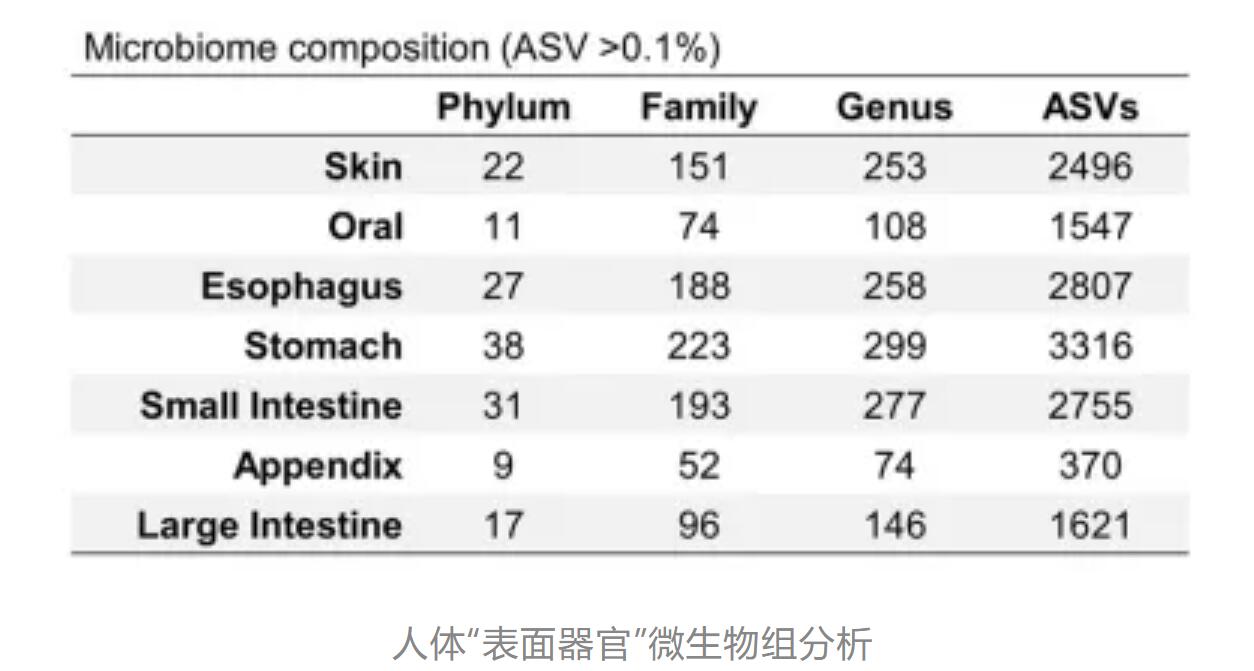
该研究旨在分析去世个体内的器官间和器官内的微生物组。研究从7个表面器官(口腔、食道、胃、小肠、阑尾、大肠和皮肤;53个部位的1608份样本。n=33名受试者)并进行微生物组分析。该研究破译了不同器官内部位的微生物变化,并确定特征微生物以及其功能特征、每个部位特有的相互作用。揭示了胃、小肠和大肠粘膜腔样本之间存在显著的微生物异质性,建立了消化道中微生物的器官间关系。研究发现与胃、阑尾、小肠或大肠相比,皮肤、口腔和食管的α多样性显著较高,胃的细菌多样性最低,推测其低pH限制了细菌的生长。拟杆菌属狄氏副拟杆菌主要富集在小肠、阑尾和大肠中;卟啉单胞菌属、普雷沃氏菌属、链球菌属和奈瑟菌属富集在口腔中;螺旋菌属在口腔和阑尾中富集;而葡萄球菌属和棒状杆菌属是皮肤中的优势属。