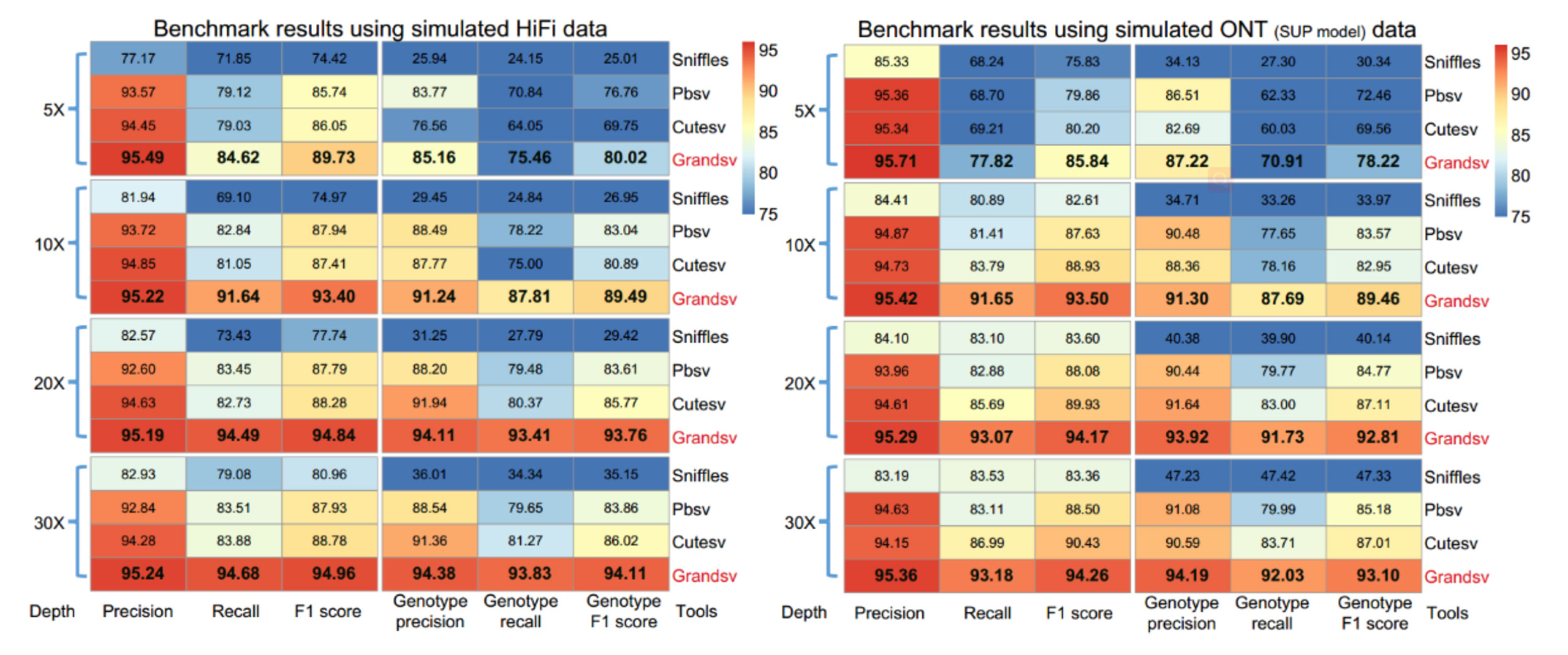
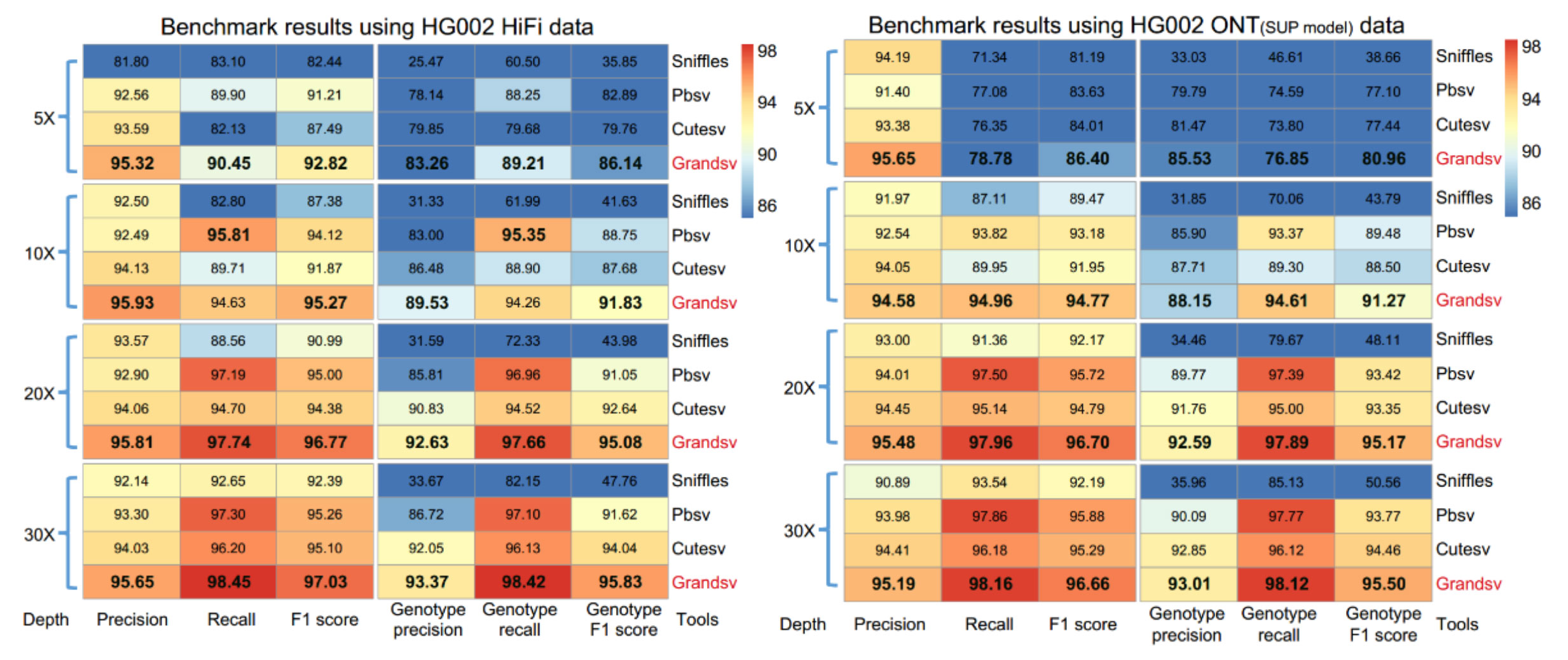
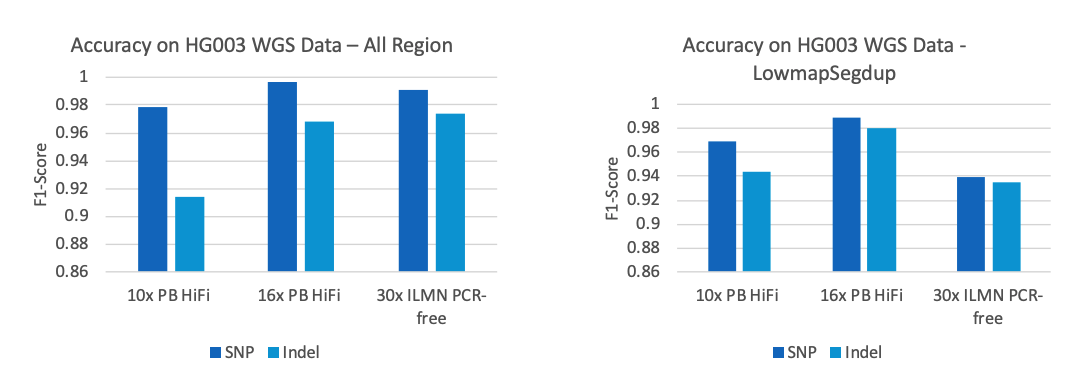
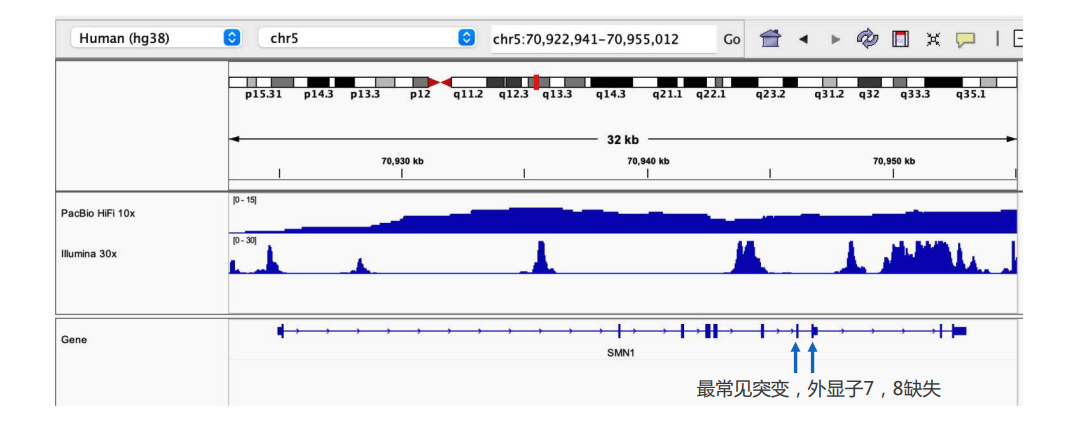
2013年3月11日,希望组(未来组)开始提供三代测序服务,成为中国首家三代测序服务公司。在不断探索与进化的过程中,公司首席生信技术官胡江团队自主研发了三代测序基因组组装领域著名的组装软件NextDenovo,该软件在极大减少计算资源和运行时间的前提下,仍然能够组装出高质量基因组,具有高纠错、高效率、高准确度的优势。今天,在我们正式提供三代测序服务第十周年,我们向全球公开该软件的源代码,并同时预印发布NextDenovo软件科学论文《An efficient error correction and accurate assembly tool for noisy long reads 》,为整个行业的健康、快速、高质量发展做出自己的贡献。
2013年3月11日,希望组(未来组)开始提供三代测序服务,成为中国首家三代测序服务公司。在不断探索与进化的过程中,公司首席生信技术官胡江团队自主研发了三代测序基因组组装领域著名的组装软件NextDenovo,该软件在极大减少计算资源和运行时间的前提下,仍然能够组装出高质量基因组,具有高纠错、高效率、高准确度的优势。今天,在我们正式提供三代测序服务第十周年,我们向全球公开该软件的源代码,并同时预印发布NextDenovo软件科学论文《An efficient error correction and accurate assembly tool for noisy long reads 》,为整个行业的健康、快速、高质量发展做出自己的贡献。
目前,该软件在论文发表之前已经累计下载11000余次,助力发表文章约500篇,受到国内外众多专家学者的一致认可和好评。

为了让大家更多的了解NextDenovo的一些背景,我们专访了希望组首席生信技术官胡江,内容如下:

1.请问当初你研发这个软件的原因是什么?
大约在2018年初,全世界组装软件做PacBio或Illumina数据的比较多,适用于Nanopore测序(ONT)数据的组装软件很少,因当时ONT测序的错误率非常高,要真正使用这些数据进行组装,要么极其消耗计算资源,要么组装效果非常差,导致我们大量ONT测序的de novo组装项目积压、交付困难,为了解决这个全球性的技术难题,我们专门立项开发NextDenovo。
2.你觉得NextDenovo的优势在哪里?
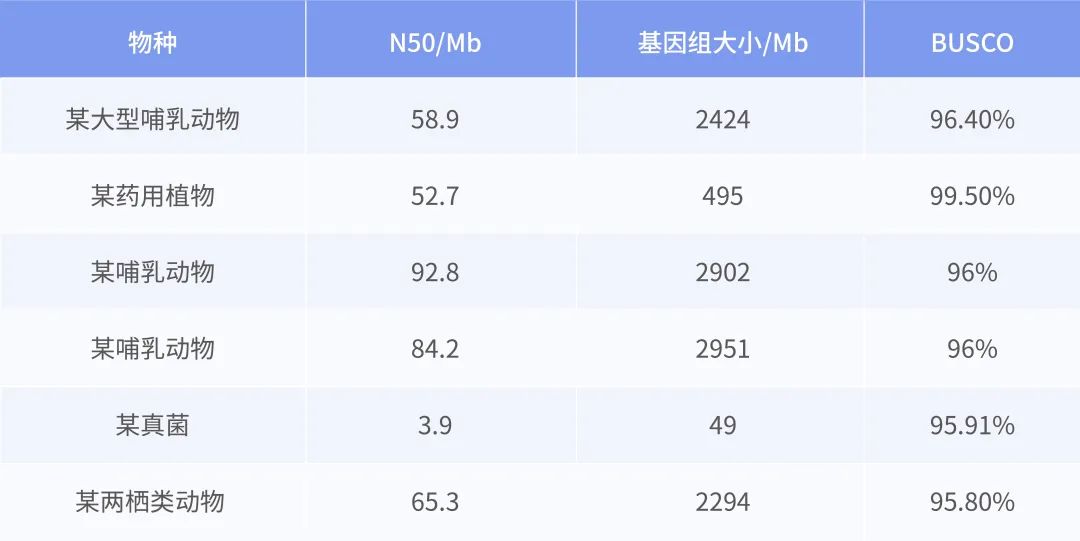
要说优势的话,可能在某些物种的组装完整度上,比如说像我们做的玉米,我们能组装到60M,现在行业里绝大部分组装软件可能只能装到20M,这个对比还是很明显的。另外一个优势就是我们可以从算法上从底层上去调整、修改,可以大范围的去解决在组装过程中每个物种所特有的个性化问题,这个大部分其他商业公司做不了的,因为大部分其他商业公司都只是使用别人开发的软件,底层的东西没法改变,但是我们可以在遇到困难的时候随时修改。
3.请问你为什么选择公开源代码?软件还会不会进行持续更新,NextDenovo和NextDenovo2有何区别?
公开一方面是为了进一步为科研领域做一点分内贡献,另一方面也是很多用户的需求。NextDenovo是针对PacBio CLR或者ONT这种高错误率的数据开发的,组装出来的结果是一个嵌合体,同时也无法直接组装出近T2T水平。NextDenovo2是针对ONT新数据即长又准的特点开发的,主要是用于组装多套基因组(分型),另外可以用于直接组装T2T水平的基因组。
4.如今科技发展迅速,你觉得未来有没有其他软件超越NextDenovo?对此你怎么看?
对于组装软件来说,每一个软件都有自己的特点,不存在哪个软件绝对好,大部分软件在某些数据或者某些物种上表现优于其他软件,但是在另外一些数据或者物种上表现就会差一些。同时,在内存消耗或者运行时间,组装结果准确度上来说,每个软件都有各自的优势,用户可以基于自己的需求选择合适的软件。作为一个组装软件开发者来说,我们的工作就是基于技术的更新,不断更新自己的软件,同时积极解决用户的问题,帮助用户快速了解、运行软件。我们可能会一直存在的一个优势就是,我们有大量的项目训练,会使NextDenovo软件继续一直的不停升级、迭代,从而大概率持续保持竞争力。
5.给我们一些想学习软件开发的人的一些建议吧?
首先就是去把我们常用的软件的文献看懂,还有一些基础的算法,比如比对算法,都有很多详细的计算一定要看懂。因为生信的算法相对来说还是比较简单的,因为都是学生物的人写的,所以不像谷歌、百度这些公司的写的那么复杂。还有就是要不断地要花时间去研究,沉下心来,总是会有收获的。
6.最后,你能介绍一下NextDenovo软件名字的来由吗?
我们开发这个软件的时候,借鉴了华大基因当年开发二代测序软件取名——SOAPdenovo,我们想,我们是中国第二家真正开发组装软件的公司,我们又是在三代测序新技术上开发的,我们干脆就叫NextDenovo吧,就是Next-Generation Denovo的意思,我们的初心是做下一代的最前沿的组装算法和工具。

西安交通大学 叶凯教授
在基因组领域,常用和好用的软件往往是欧美顶尖实验室开发的,这导致我国在样本资源丰富、数据质量并不落后的情况下,总是落后半拍。NextDenovo的开源发布,为我国基因组研究提供了关键技术支撑,为世界基因组研究贡献中国智慧。希望组作为一家企业,发布自主研发的计算方案,却不限制商用,为领域树立了一个标杆,开启了我国基因组领域企业、科研单位开放合作、交叉创新的新篇章。

中山医院眼科 肖传乐教授
NextDenovo是一套非常优秀的序列校正和组装软件。该软件的校正算法详细设计了针对重复区域的校正方法,相比Canu和NECAT相比,该软件在重复区域的校正方面表现出色。因此,该软件特别适合用于校正含有重复区域的超长读长序列,校正后序列的精度可以高达97%-99%,高精度的重复区域校正使得后续的端到端组装变得容易和简单。高精度超长读长校正数据与HIFI数据能够充分发挥其超长和高精度的优势,从而显著提高了端到端基因组组装的成功率,这也是希望组公司成功组装端到端基因组案例最多的原因之一。此外,各种基因组组装软件性能在不同基因组中表现都不一样,其主要原因是组装软件开发者没有足够的数据对软件进行反复提升,而NextDenovo源于希望组公司,可以很好克服这个问题。NextDenovo经历了各种复杂基因组组装难题的提升,并成功应用于许多超复杂和巨大的基因组组装。这些宝贵的经验将被写入NextDenovo软件算法中,值得我们科研人员学习和借鉴。”

昆明动物所 吴东东研究员
三代基因组测序技术飞速发展,相关研究领域也迎来新的一波助力。NextDenovo软件在三代测序数据方面表现出高纠错、高效率、高准确度的优势,尤其针对价格相对便宜的Nanopore 三代测序数据。软件自从公开后,在行业内引起广泛关注和使用,不乏肺鱼、南极磷虾等超大基因组的组装。 相信NextDenovo系列软件必将助推三代测序技术在人类队列泛基因组、精准医学、肿瘤基因组、遗传疾病诊断、农业基因组辅助育种、保护基因组学等等方方面面的应用。

希望组 CEO汪德鹏
10年前,我们学习Michael Schatz、Evan Eichler、Michael Snyder、Au Kin Fai、Jason Chin等行业领导者文章和报告,在全世界学术开放、合作的环境中成长;今天,10年之后,也应该我们为全世界学术界贡献我们自己的研发成果,为全球学术开放和合作添砖加瓦。
本次NextDenovo公开源代码后,我们将继续研发它的升级版本NextDenovo2,NextDenovo2将主要瞄准三代测序T2T基因组组装。NextDenovo开源之后,将不再限制基因组的大小,也不再限制基于NextDenovo的商业使用,但是,如果需要全方位的技术支持,希望组将继续为全球客户提供技术支持服务。
使用NextDenovo软件组装的基因组已发表文献精选:
01南极磷虾基因组
文章:The enormous repetitive Antarctic krill genome reveals environmental adaptations and population insights.
发表期刊:Cell
02非洲肺鱼基因组
文章:African lungfish genome sheds light on the vertebrate water-to-land transition.
发表期刊:Cell
03苏铁基因组
文章:The Cycas genome and the early evolution of seed plants.
发表期刊:Nature Plants
04燕麦基因组
文章:Reference genome assemblies reveal the origin and evolution of allohexaploid oat.
发表期刊:Nature Genetics
05中华绒螯蟹基因组
文章:“Omics” data unveil early molecular response underlying limb regeneration in the Chinese mitten crab, Eriocheir sinensis.
发表期刊:Science Advances
06樱花基因组
文章:Genome assembly, resequencing and genome-wide association analyses provide novel insights into the origin, evolution and flower colour variations of flowering cherry.
发表期刊:The Plant Journal
07红花槭基因组
文章:The chromosome-scale genome provides insights into pigmentation in Acer rubrum.
发表期刊:Plant Physiology and Biochemistry
08水稻基因组
文章:The telomere-to-telomere gap-free genome of four rice parents reveals SV and PAV patterns in hybrid rice breeding.
发表期刊:Plant Biotechnology Journal
09白菜基因组
文章:A near-complete genome assembly of Brassica rapa provides new insights into the evolution of centromeres.
发表期刊:Plant Biotechnology Journal
10西瓜基因组
文章:A telomere-to-telomere gap-free reference genome of watermelon and its mutation library provide important resources for gene discovery and breeding.
发表期刊:Molecular Plant

 2013年3月11日,希望组(未来组)开始提供三代测序服务,成为中国首家三代测序服务公司。在不断探索与进化的过程中,公司首席生信技术官胡江团队自主研发了三代测序基因组组装领域著名的组装软件NextDenovo,该软件在极大减少计算资源和运行时间的前提下,仍然能够组装出高质量基因组,具有高纠错、高效率、高准确度的优势。今天,在我们正式提供三代测序服务第十周年,我们向全球公开该软件的源代码,并同时预印发布NextDenovo软件科学论文《An efficient error correction and accurate assembly tool for noisy long reads 》,为整个行业的健康、快速、高质量发展做出自己的贡献。
2013年3月11日,希望组(未来组)开始提供三代测序服务,成为中国首家三代测序服务公司。在不断探索与进化的过程中,公司首席生信技术官胡江团队自主研发了三代测序基因组组装领域著名的组装软件NextDenovo,该软件在极大减少计算资源和运行时间的前提下,仍然能够组装出高质量基因组,具有高纠错、高效率、高准确度的优势。今天,在我们正式提供三代测序服务第十周年,我们向全球公开该软件的源代码,并同时预印发布NextDenovo软件科学论文《An efficient error correction and accurate assembly tool for noisy long reads 》,为整个行业的健康、快速、高质量发展做出自己的贡献。






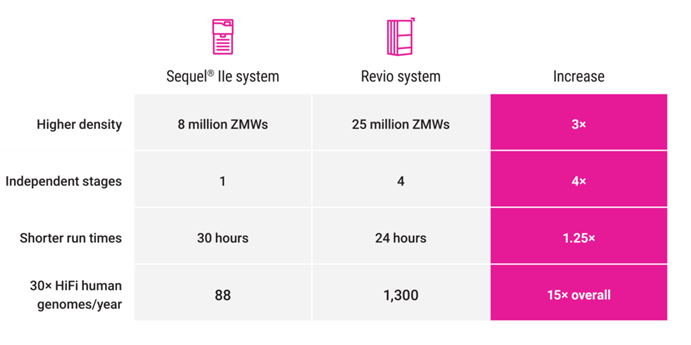
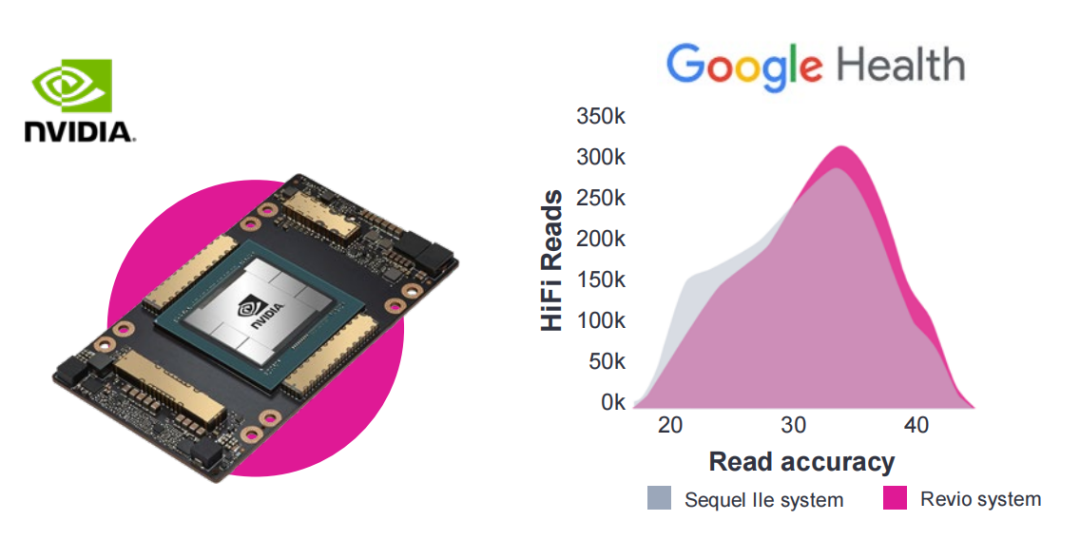





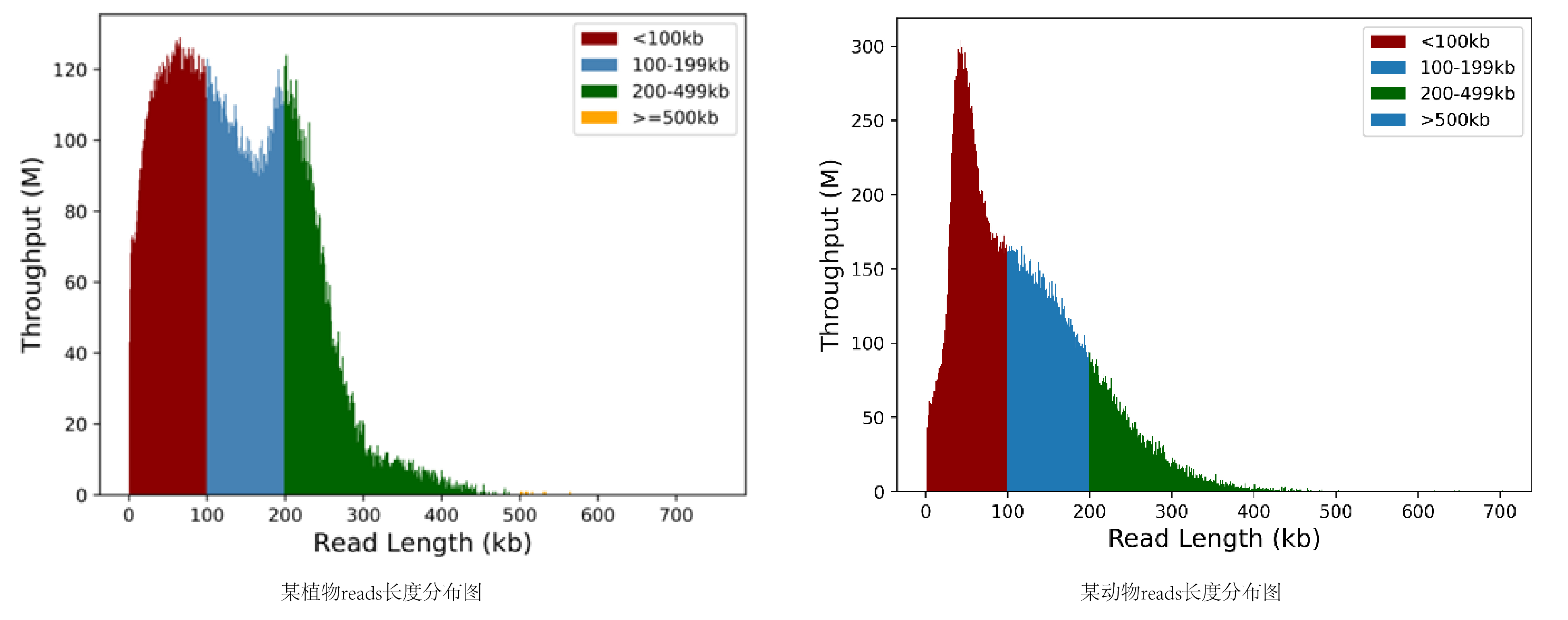
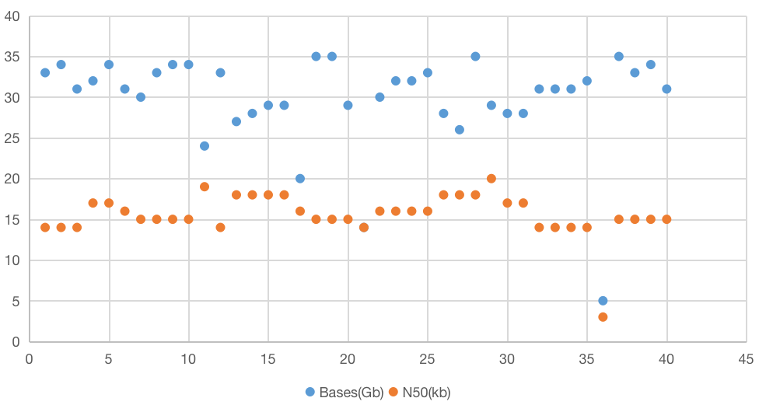 HiFi数据量和长度统计
HiFi数据量和长度统计