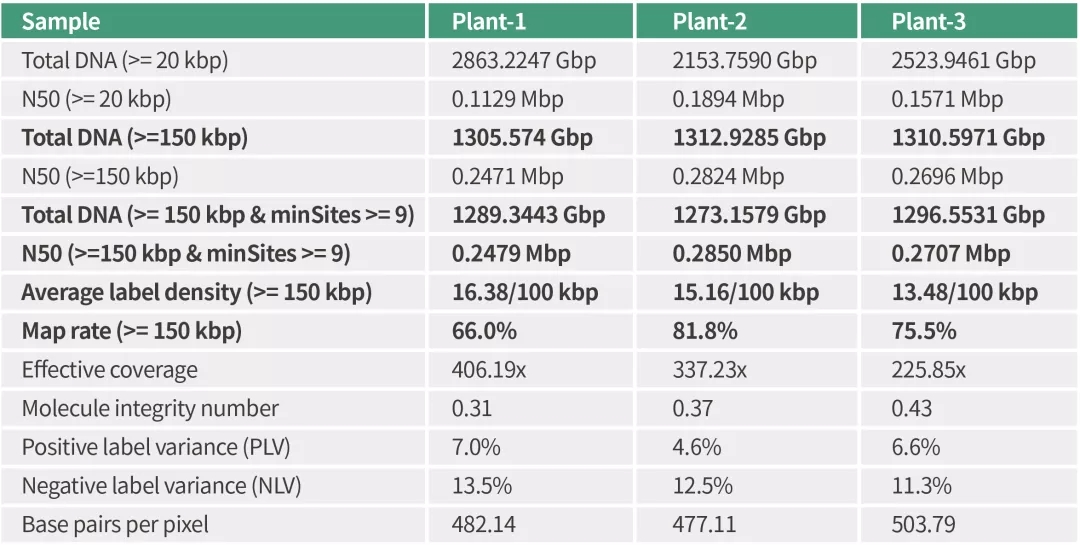
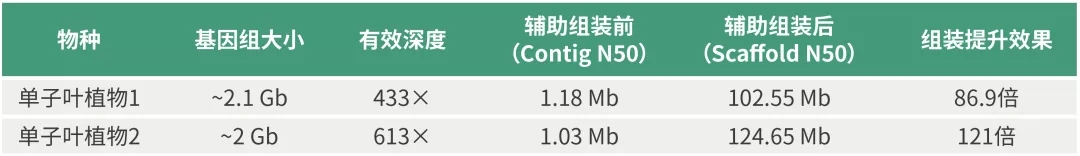
继攻克大型基因组组装难题后,希望组科技服务开发出自动化Hi-C纠错挂载流程,不仅能纠正组装序列的错误,还解决了大型基因组Hi-C挂载错误多,难以人工校正的问题,实现大型基因组从草图到染色体水平组装的全流程技术突破。
目前,利用Hi-C技术将基因组草图序列高精度地定位到染色体,并确定其在染色体上的顺序和方向,构建出染色体水平基因组,已经成为动植物基因组de novo组装的标准配置。然而,某些具有大型基因组的物种很难获得染色体水平的基因组,一方面,大型基因组草图组装难度大,已经发表的大型基因组,组装指标普遍较低,Contig N50多在Kb级别;另一方面,碎片化的大型基因组草图,导致Hi-C挂载率普遍不高组内错误较多,并且几乎无法通过人工方式进行调整。缺少染色体级别的基因组序列,严重阻碍了大型基因组染色体进化、比较基因组及三维基因组研究工作。
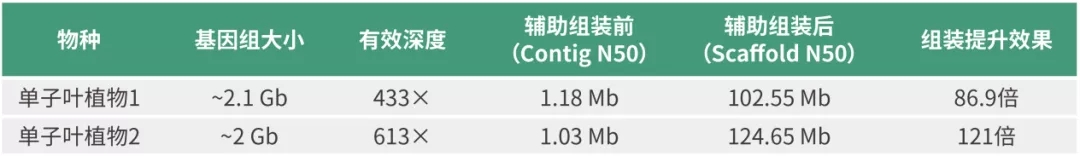
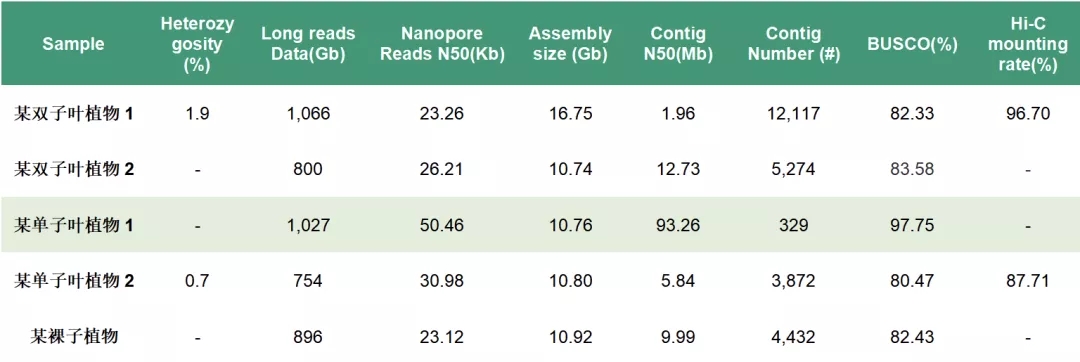
针对大型基因组组装问题,希望组基于NextDenovo自主组装算法结合纳米孔测序的超长读长优势,已完成了多个10Gb以上大型基因组的测序组装工作,获得的大型基因组的Contig N50均在Mb水平,基因组完整性也较高,为实现染色体水平的组装奠定坚实基础。
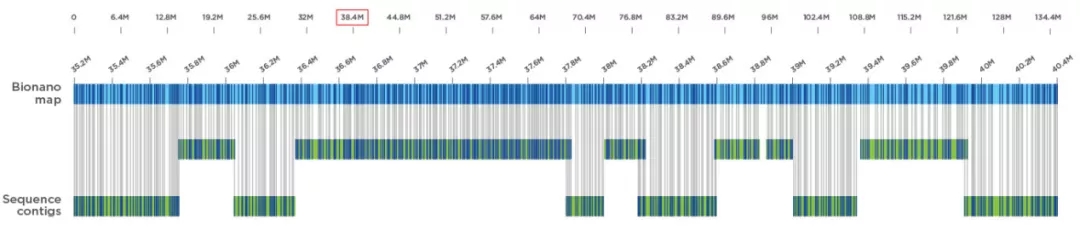
基于高质量的基因组草图,大型基因组的Hi-C挂载率提升明显(95%以上),但是热图显示组内挂载错误较多(图1)。使用传统的软件或人工可以对比较小的基因组进行Hi-C热图校正,但是大型基因组几乎无法采用这种方法。

图1 某大型基因组Hi-C初始挂载热图(左)和自动化纠错、校正后的Hi-C热图(右)
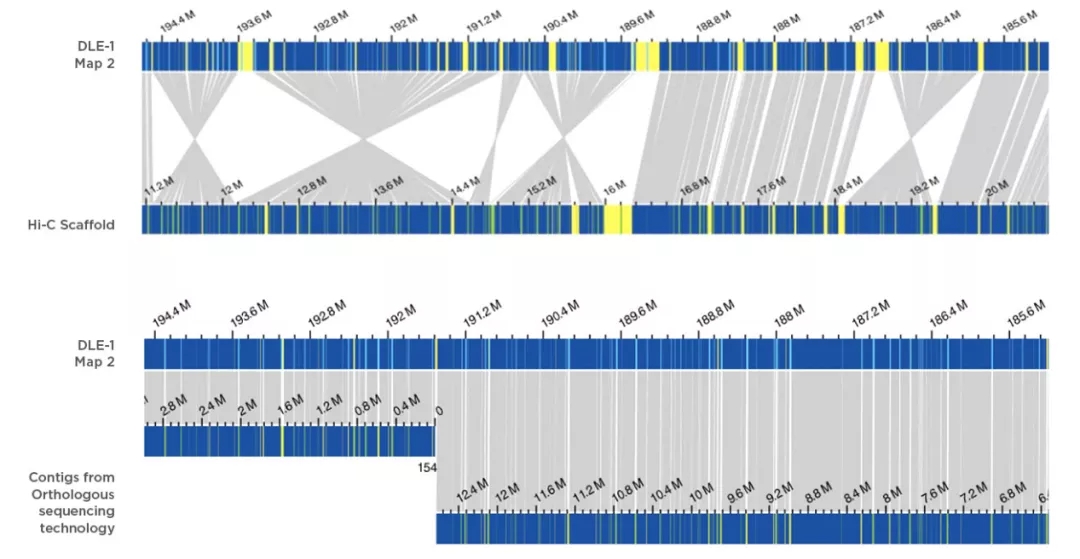
为此希望组开发出自动化Hi-C纠错挂载流程,可以进行Hi-C互作热图绘制展示和组装结果调整,从图形上快速校正已有组装的错误。某大型基因组经过自动化Hi-C纠错、校正以后组内错误得到了极大改善,热图中邻近的序列间(对角线位置)交互强度高,而非邻近的序列之间(非对角线位置)的交互信号强度弱,在对角线以外区域没有明显的噪音(较强交互强度),证明基因组组装效果较好(图1)。
大型基因组的测序和组装仍是世界性难题,希望组通过自主研发NextDenovo组装算法、攻克纳米孔超长测序瓶颈、开发自动化Hi-C纠错挂载流程,最终实现大型基因组从草图到染色体水平组装全流程的技术突破。这是希望组在基因组组装领域持续深耕多年的技术实力的集中体现。
对大型基因组组装感兴趣?
请联系希望组当地科技顾问,或发送邮件至sales-support@grandomics.com。
希望组科技服务,为您的科研之路保驾护航!