Genome Biology 项目文章| 范衡宇教授团队揭示ZAR1/2调控卵母细胞母源mRNA动态变化的新机制

2025年5月9日,浙江大学生命科学研究院范衡宇教授团队在Genome Biology杂志上发表了题为“ZAR1 and ZAR2 orchestrate the dynamics of maternal mRNA polyadenylation during mouse oocyte development”的研究论文。该研究联合Smart-seq2、Total RNA-seq、PAIso-seq2和LACE-seq四种近年来开发的、适用于低起始量样本的转录组检测技术,从mRNA稳定性和聚腺苷酸化状态两个维度重新探讨了卵母细胞在减数分裂过程中的转录本动态变化,并深入分析了ZAR1在母源转录组调控中的作用机制。希望组为本研究提供了PB转录组测序服务。
研究背景
在减数分裂期间,卵母细胞基因组会长时间处于转录沉默状态,直到合子基因组激活(ZGA)才重新启动。这一阶段,母源转录组的动态变化和稳态对母源-合子转换(MZT)至关重要。然而越来越多证据表明Smart-seq2(一种低输入量建库技术)检测到的母源转录组动态可能不够准确。其原因是该技术依赖oligo-d(T)引物捕获母源mRNA的poly(A)尾可能会引入偏差。
结果与分析
在生发泡破裂(GVBD)后的减数分裂过程中,母源基因组转录保持沉默,多年来研究者观察到母源转录组逐渐减少的现象(图1)。这一时期发生降解的转录本对应的基因被称为O-decay基因。通过Smart-seq2和Smart-seq3测序结果显示,与GV期相比,MII期卵母细胞中有许多基因下调,而在差异基因表达中却存在许多上调基因(图1),这些结果显示通过Smart-seq2定义的O-decay基因可能存在偏差。为探究这一问题,该研究采用PAIso-seq2分析技术检测减数分裂过程中多聚腺苷酸化状态的变化,发现GV到MII期卵母细胞中,大部分转录本poly(A)尾明显缩短,这可能导致Smart-seq2无法准确检测短poly(A)尾的mRNA。
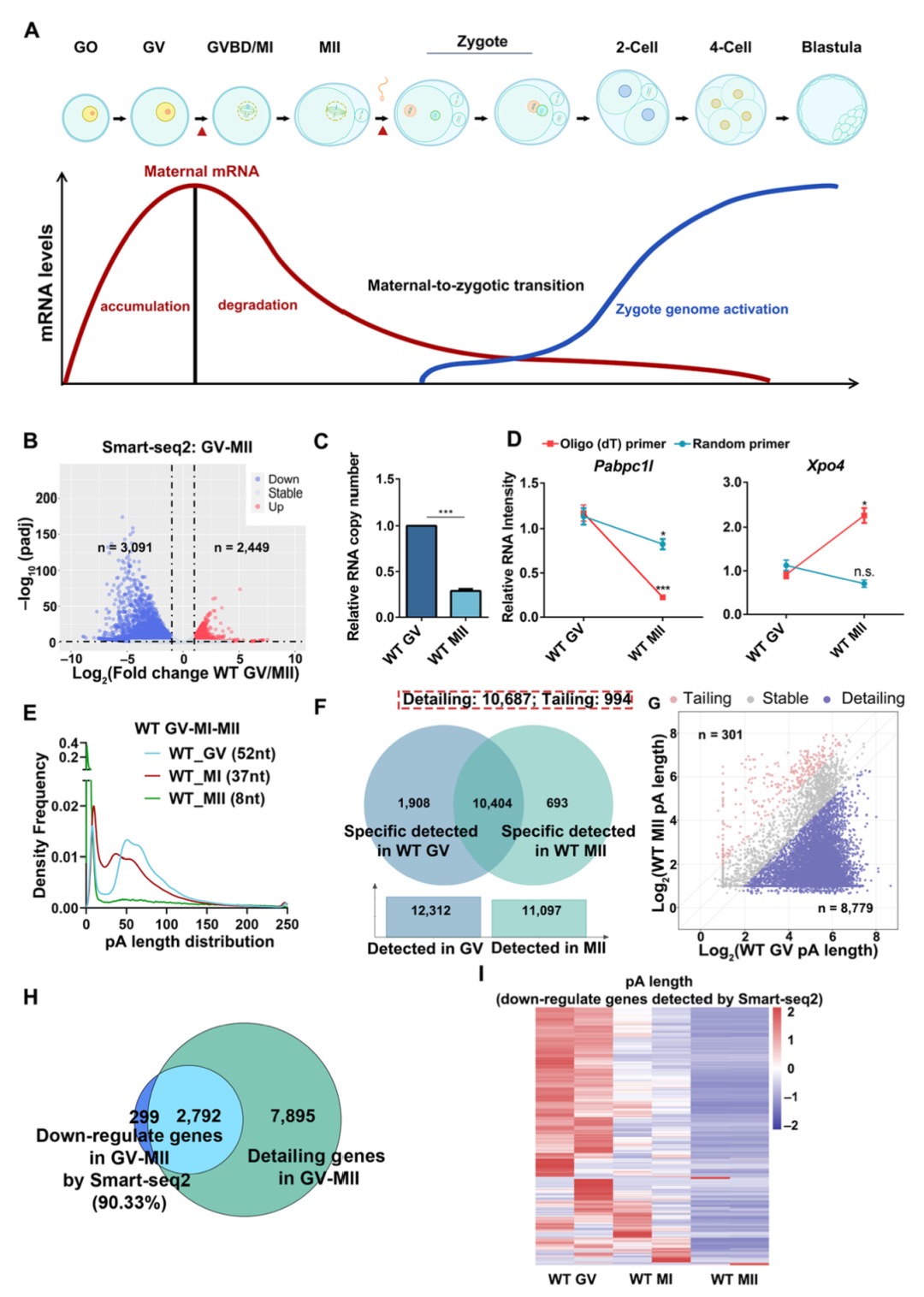
图1 Smart-seq2和PAIso-seq2检测母源mRNA动态变化
为规避Smart-seq2的系统偏差,该研究使用Total RNA-seq数据进行分析,结果显示Total RNA-seq检测到的母源转录组在减数分裂过程中的整体下调幅度显著低于Smart-seq2的结果,且鉴定的O-decay基因数量明显更少(图2)。对Total RNA-seq鉴定的O-decay基因进行了深入分析,发现O-decay基因的转录本主要在GV期卵母细胞中发生多聚腺苷酸化和翻译,在完成生物学功能后被有序降解。因此研究推测在减数分裂过程中,母源转录组经历的多聚腺苷酸化/去腺苷酸化修饰可能比实际降解更为显著。
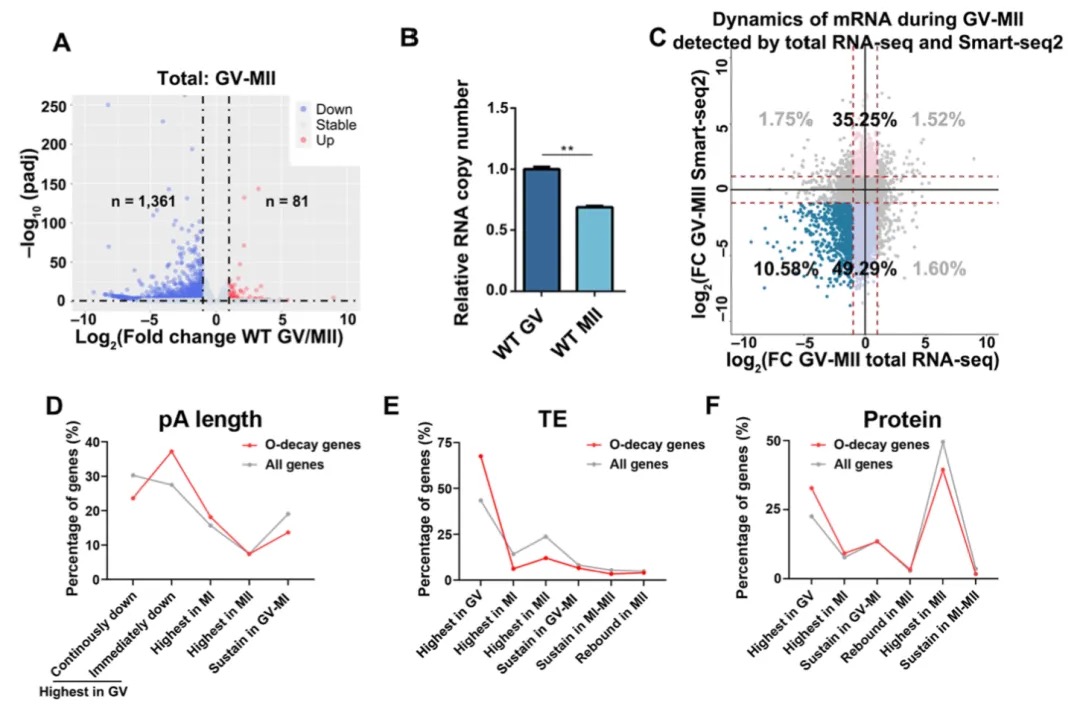
图2 Total RNA-seq检测母源mRNA动态变化
早期研究推测ZAR1促进减数分裂中母源mRNA的降解,而近期研究发现ZAR1参与线粒体相关核糖核蛋白域(MARDO)的组装。为阐明这一矛盾,研究采用总RNA-seq技术对Zar1/2-/-卵母细胞进行差异表达基因分析,结果检测到GV期母源mRNA显著下调,且Zar1/2-/-GV期下调基因中61.76%与正常GV→MII期应下调或持稳的基因相关,说明Zar1/2–在GV期通过稳定mRNA维持其表达。
ZAR1作为RNA结合蛋白(RBP),通过其C端CxxC锌指结构域与母源转录本结合。基于这一分子特征,该研究深入探究了ZAR1的调控机制。LACE-seq定位到其靶向转录本,共鉴定8,000余个潜在靶标,其中Zar1/2-/-GV期下调基因中54.8%为ZAR1靶标。此外发现3’UTR结合基因的稳定性显著高于CDS结合基因,提示ZAR1通过3’UTR结合维持mRNA稳态(图3)。
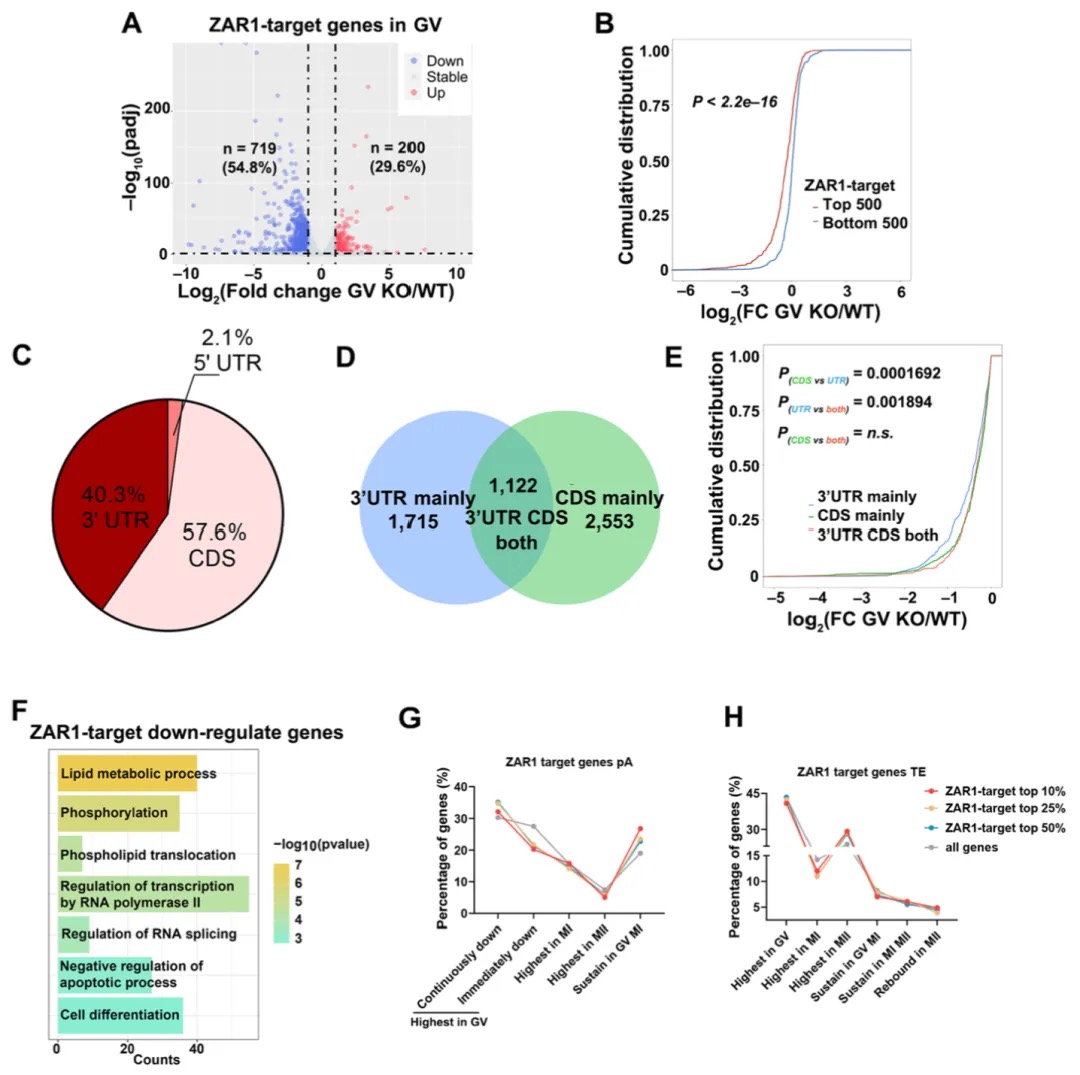
图3 ZAR1通过与3‘UTR区域结合来稳定母体转录本
鉴于Zar1/2-/-卵母细胞中母源转录本多聚腺苷酸化显著异常,研究进一步分析了差异多聚腺苷酸化基因(DPGs)与ZAR1靶标的关系,分析发现ZAR1本身并不直接调控多聚腺苷酸化,ZAR1更倾向于影响mRNA稳定性,而非直接影响poly(A)尾长。研究通过IP-MS技术发现ZAR1与PABP家族、IGF2BP2等RNA稳定因子互作,提示ZAR1可能与其他蛋白质相互作用以调节母源转录组的稳态(图4)。
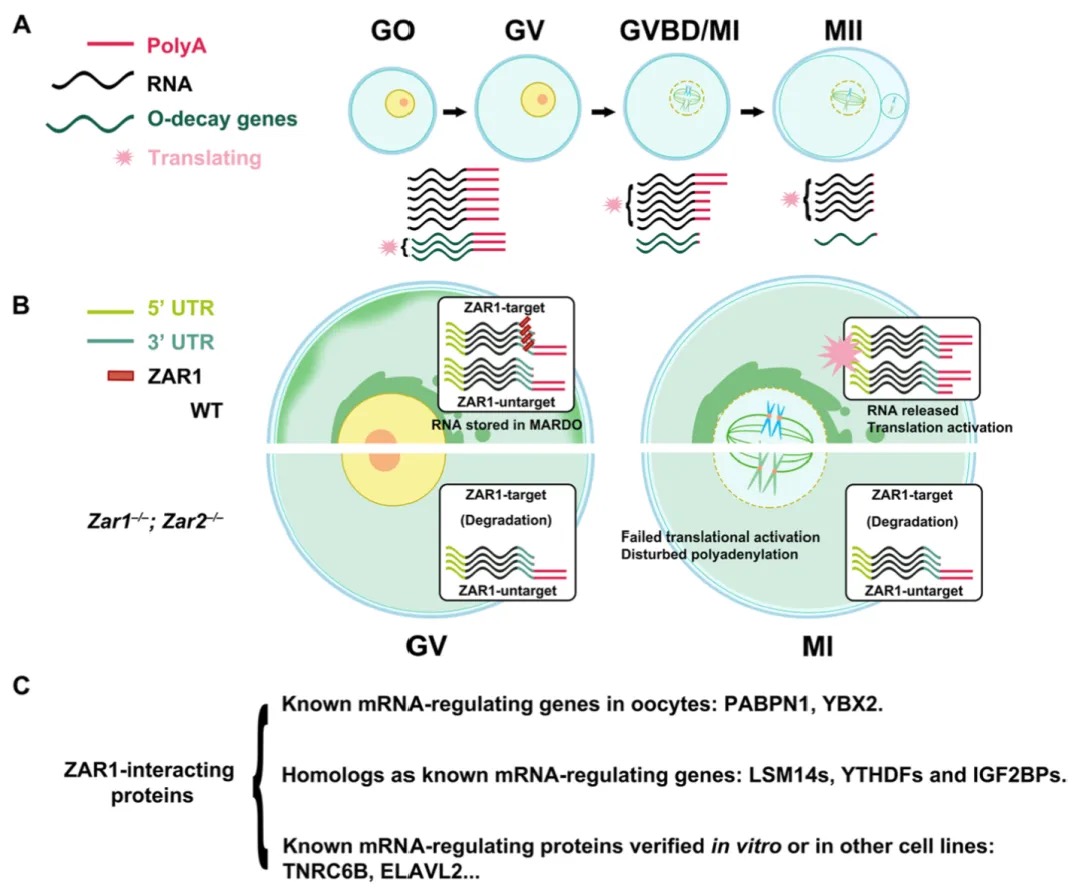
图4 ZAR1在卵母细胞发育过程中调控mRNA多聚腺苷酸化的工作模型
Zar1/2-/-卵母细胞在MII期染色质压缩维持失败,出现纺锤体错位和类原核结构等现象,伴随关键母源基因(如Lsm14b、Ccnb1)表达异常,表明ZAR1在稳定这些基因中起作用,从而有助于维持卵母细胞处于MII期。
文章链接:https://doi.org/10.1186/s13059-025-03593-8



