Nature项目文章|重磅!韩斌院士团队绘制全球首个”野生-栽培稻精细泛基因组图谱”,破解水稻驯化之谜!
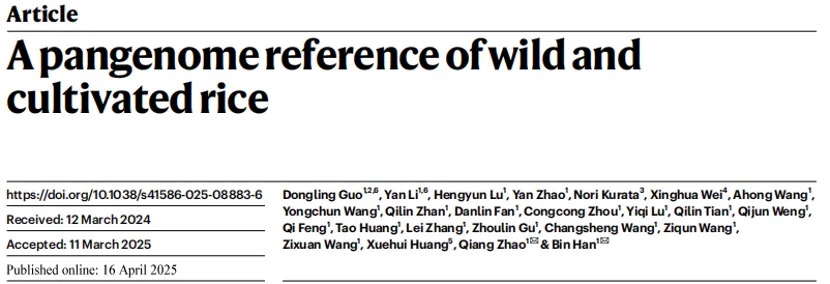
此外,该研究结合Hi-C数据获得30个代表性物种染色体级别组装,与参考基因组共线性验证显示高度一致性,根据端粒重复序列,在每个基因组中平均检测到19个端粒。为更全面表征序列多样性,研究从133个HiFi基因组中捕获到替代组装(a-contig)序列。这些替代组装序列包含了主组装(p-contigs)中缺失的杂合信息。值得注意的是,栽培稻中替代组装序列的规模显著小于野生稻,反映了前者较低的杂合率。这些发现为理解驯化过程中基因组稳定性演变提供了新视角。也强调了在育种中利用野生稻杂合基因组资源的重要性。
由于长期自然选择,野生稻对生物和非生物胁迫具有高度抗性,是天然的抗性等位基因库。因此,通过对野生稻与栽培稻中抗病基因类似物(RGAs)进行分析,研究发现野生稻含有1,710个抗性基因类似物(RGAs),显著高于栽培稻的1,652个。进一步通过共线性分析发现野生稻中具有638个特有RGA位点,其中包括一个野生稻RLK基因LOC_Os07g35680,该基因近期被报道为OsMADS26介导的稻瘟病负调控因子。另外在野生稻中鉴定出多个携带稻瘟病抗性基因Pi5的材料。这些结果表明,野生稻蕴含丰富的抗病资源,可作为培育高抗水稻品种的供体材料。
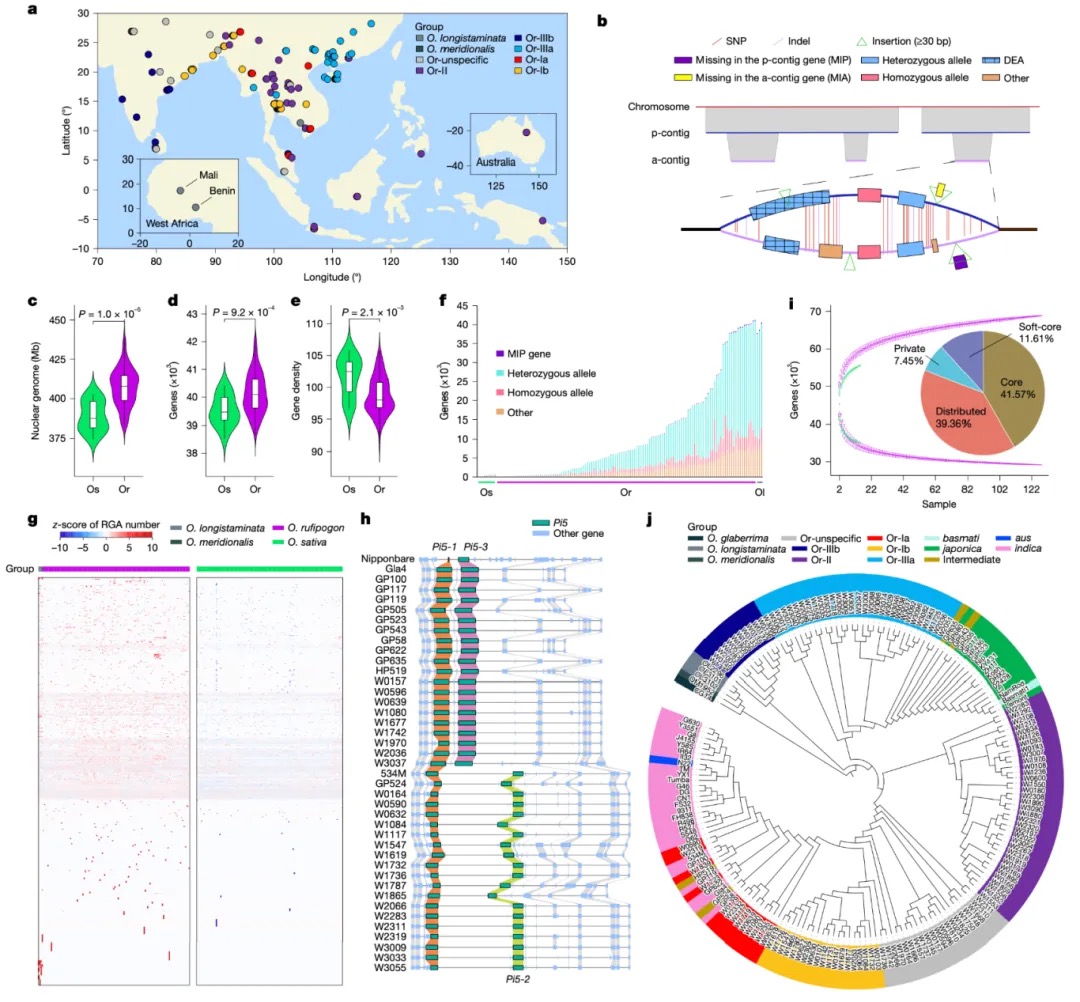
图1 149个代表性野生和栽培水稻品种及泛基因组构建分析
通过对本研究所有样本及已发表33个栽培稻泛基因组中28份亚洲栽培稻的TE注释分析,发现野生稻平均TE含量(53.23%)略高于栽培稻(52.32%),这解释了野生稻基因密度较低的现象。Or-IIIa群体的TE含量显著高于其他群体,而粳稻的TE含量最低。转座元件(TE)进化分析表明,Gypsy家族在Or-IIIa群体中约2.5万年前经历显著扩张,相较粳稻基因组,17个关键Gypsy家族贡献了25.85Mb序列差异。在Or-IIIa中鉴定出约1,000个与扩张Gypsy家族相邻的基因,这些基因显著富集于碳水化合物和萜类代谢和细胞表面信号转导通路,暗示其在环境适应和抗病过程中发挥积极作用(图2)。
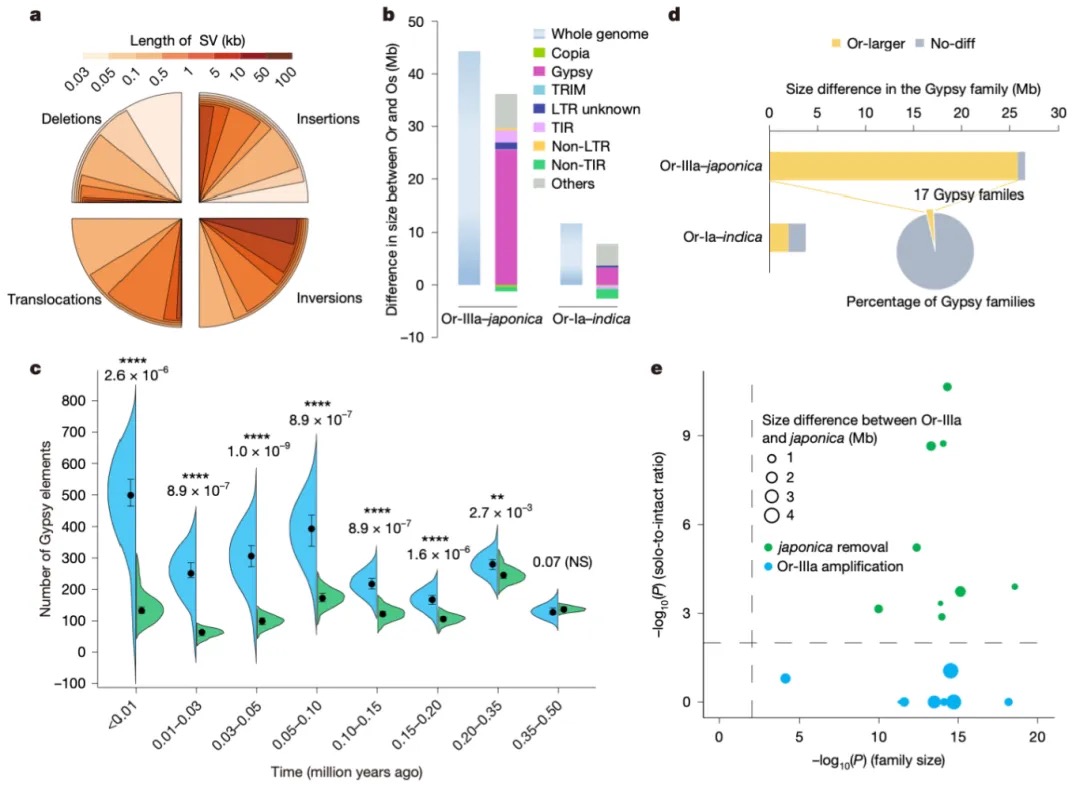
图2 野生和栽培水稻的基因组变异和TE组成分析
图3 野生稻与栽培稻的种群结构及基因渗入分析
亚洲栽培稻的驯化模式一直存在单次驯化与多次独立驯化的学术争议。研究根据选择性清除分析鉴定出12.35Mb的驯化区域,成功捕获了水稻驯化早期关键基因,包括Bh4、PROG1、sh4、FZP等11个已知驯化基因。这11个关键驯化基因的单倍型分析为早期驯化史提供了决定性证据。单倍型网络显示所有栽培稻中这些早期关的驯化基因均与Or-IIIa单倍型直接相关或通过粳稻单倍型间接关联,主要单倍型与最近Or-IIIa单倍型间的变异位点可能是驯化过程中的选择靶点。结合系谱分析将各驯化基因划分为驯化型和祖先型,其品种间分布模式印证了以下驯化路径:粳稻最早由中国南方Or-IIIa群体驯化,向东南亚扩散后与当地Or-Ia和Or-Ib杂交形成籼稻和
aus稻,南亚栽培稻频繁杂交产生intro-indica(籼稻×aus稻),intro-indica与粳稻杂交形成basmati稻(图4)。这些结果强有力支持亚洲栽培稻单次驯化假说。
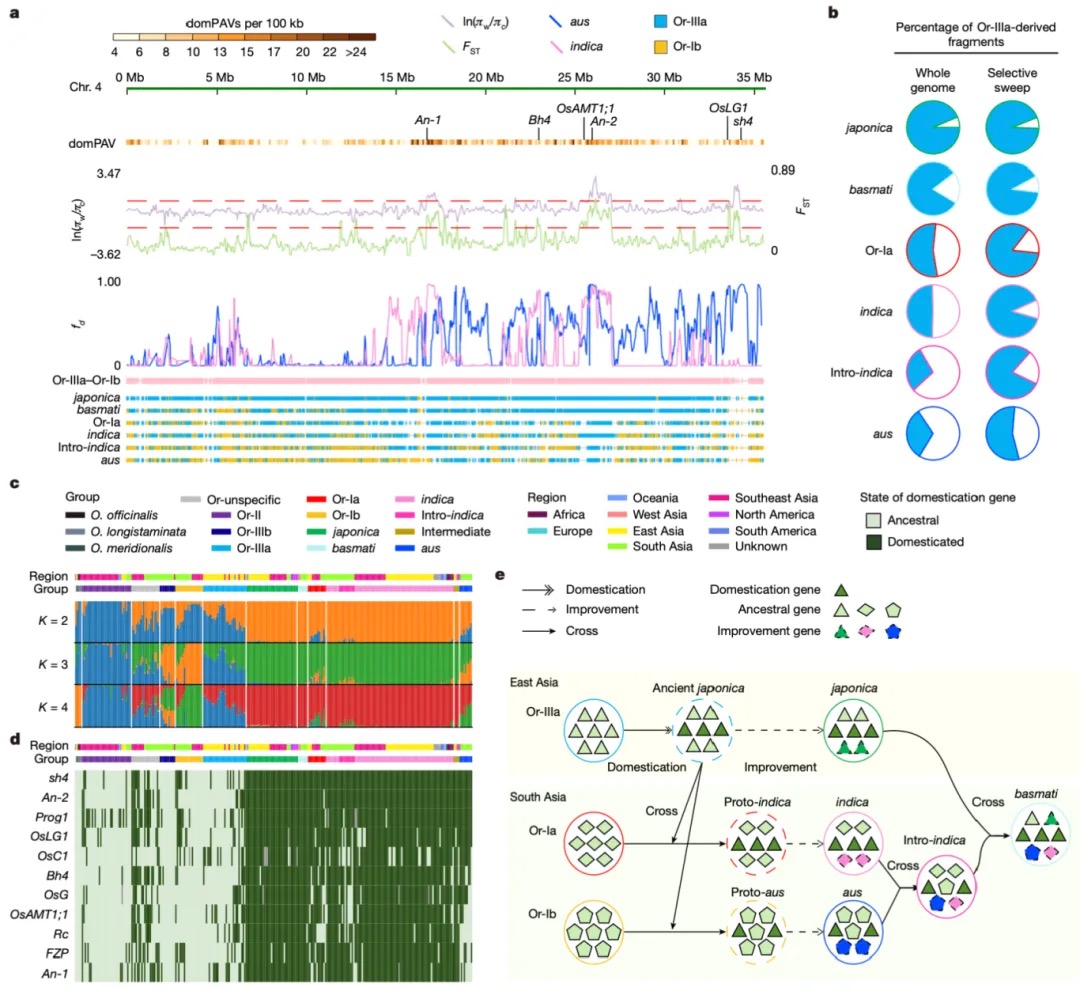
图4 亚洲栽培水稻进化路线
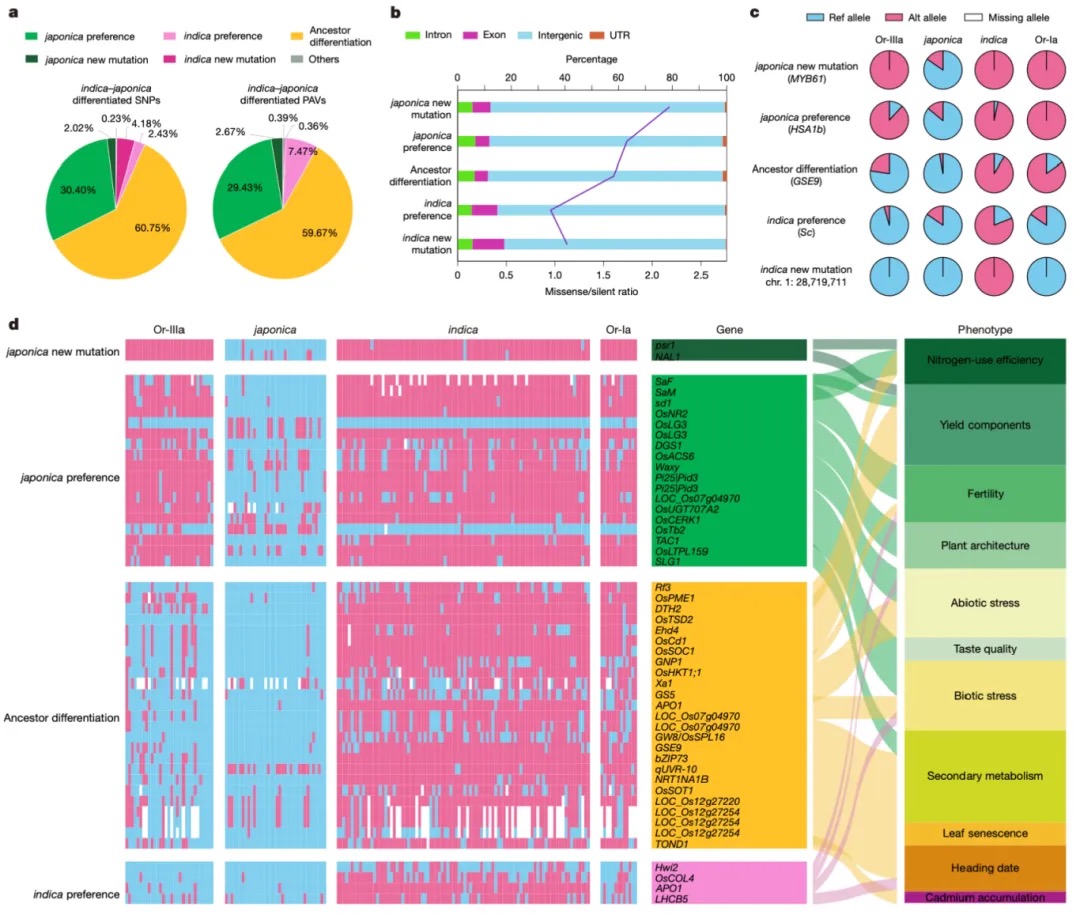
图5 籼稻和粳稻之间的遗传差异
中国科学院分子植物科学卓越创新中心韩斌院士和赵强研究员为本文共同通讯作者,博士研究生郭东灵和高级工程师李艳为本文共同第一作者。上海师范大学黄学教授也为该研究提供了大力支持。该研究受到国家自然科学基金、中国科学院先导专项和国家农业农村部重点研发项目的资助。
文章链接:
https://doi.org/10.1038/s41586-025-08883-6





