希望组NextDenovo助力破译迄今最大的2个动物基因组:南极磷虾(48G)和肺鱼(40G)
目前已知发表的最大的两个基因组: 南极磷虾(48G)和肺鱼(40G)的基因组组装都是由NextDenovo参与协助完成的。NextDenovo软件是由希望组自主研发的三代测序基因组组装工具,在极大减少计算资源和运行时间的情况下,仍然能够组装出高质量基因组,具有高纠错、高效组装、高准确度的优势,已帮助众多科研人员进行基因组的组装以及文章的发表。
(一)The enormous repetitive Antarctic krill genome reveals environmental adaptations and population insights
磷虾是磷虾属的软体甲壳类动物,是所有海洋生态系统的重要组成部分。南极磷虾(Euphausia superba)的生物量为3-5亿吨,是地球上最大的野生动物物种。磷虾基因组估计为42–48Gb,其庞大的基因组规模和复杂性阻碍了它的组装,并阻碍了对南极磷虾适应性遗传基础的研究。2023年3月2日,国际顶级期刊Cell上发表题为“The enormous repetitive Antarctic krill genome reveals environmental adaptations and population insights”的研究论文,揭示了南极磷虾适应南大洋的基因组基础,并为未来的南极研究提供了宝贵的资源。武汉希望组为本研究提供基因组组装服务,武汉希望组首席生信技术官胡江为共同作者。

发表期刊:Cell (IF:66.85)
研究对象:南极磷虾
主要测序技术:Hi-C、PacBio
主要完成单位:中国水产科学研究院黄海水产研究所、青岛华大基因研究院、德国阿尔弗雷德•魏格纳研究所、澳大利亚联邦科学与工业研究组织等机构
希望组贡献:提供NextDenovo组装技术支持
部分研究结果
01染色体水平基因组组装和评估
研究者利用PacBio、Hi-C结合短读长对南极磷虾(图1A)进行测序,使用NextDenovo v2.30 (https://github.com/Nextomics/NextDenovo)组装了48.01Gb的基因组,这是迄今为止报道的最大的动物基因组组装。它比墨西哥蝾螈大约大50%,比两种肺鱼大20%-30%。与120个已经组装的无脊椎动物基因组相比,该组装具有更长的contig N50(178.99kb)(图1B),scaffold N50更是达到了1.08Gb。南极磷虾基因组中的重复DNA异常丰富,使得基因组组装特别具有挑战性。研究发现,基因组组装中含有很大比例的串联重复(TRs)(25.77%),因为TRs很难组装,特别是对于长度大于50bp和高丰度的TRs(图1C)。南极磷虾基因组的重复区密度高于墨西哥蝾螈、肺鱼和两种孔雀石甲壳类动物(图1D)。该基因组组装结果表明,巨大的南极磷虾基因组可以归因于重复序列扩增。72.15%的基因组序列被鉴定为重复序列,在附加重复注释后达到92.45%,略高于报道的澳大利亚肺鱼(90.00%)(图1E)。南极磷虾、凡纳滨对虾和弗吉尼亚磷虾之间的DNA/CMC- EnSpm系统发育树显示,南极磷虾中没有显著扩张的特定分支(图1F)。
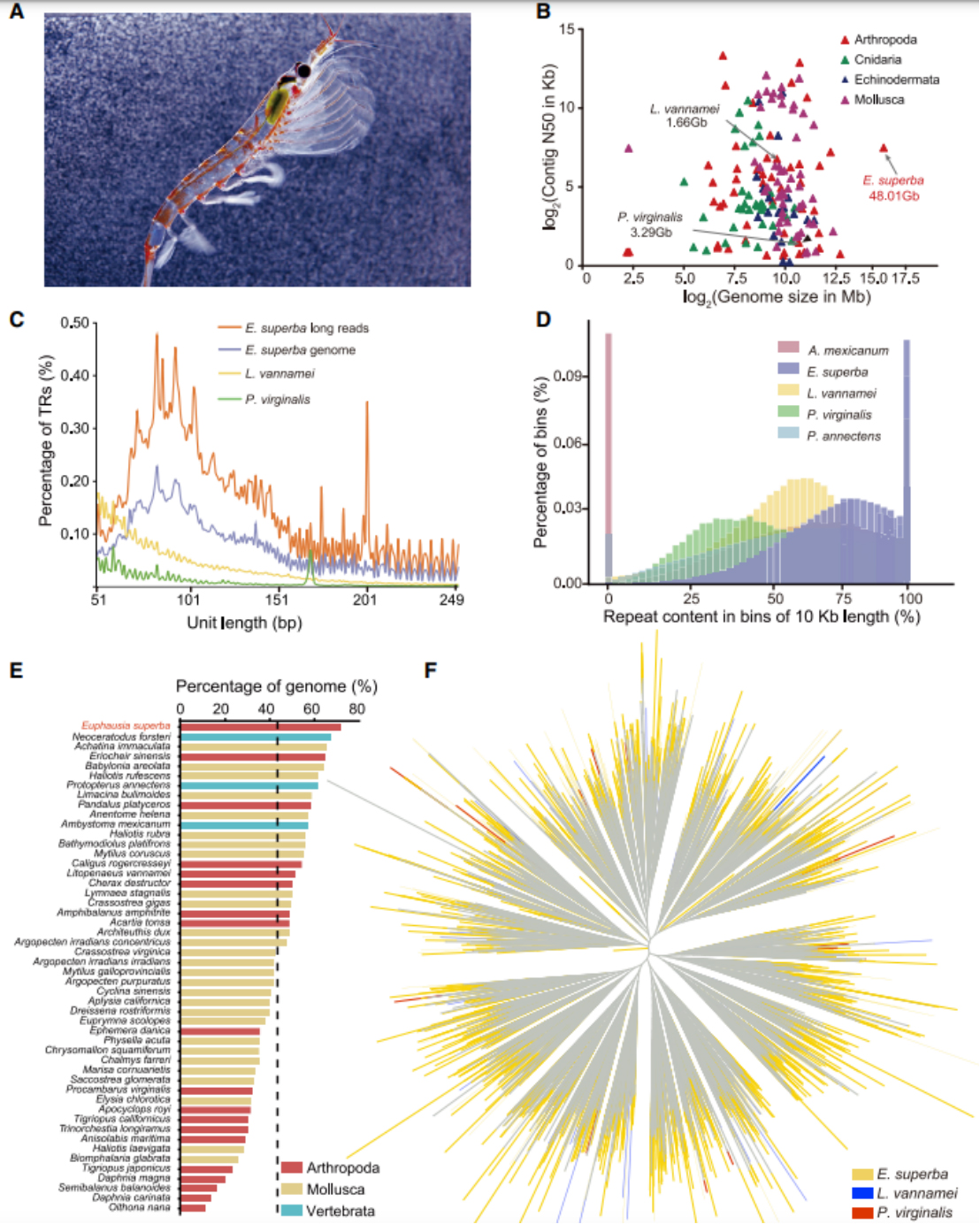
图1 南极磷虾基因组图谱及其重复序列特征
02南极磷虾环境适应的基因组基础
南极磷虾与其他真核生物一样,能够产生自我维持的昼夜节律(反馈回路)。这些包括主要的时钟抑制剂PER、TIM和CRY2以及直接调节CLK和CYC表达的三个关键昼夜节律转录因子VRI、PDP1和REV-ERB。该发现提供了磷虾生物钟的分子结构模型,证实了双反馈回路机制可能存在。进一步评估了生物节律反馈回路中基因表达的季节性差异,揭示了四个昼夜节律基因(CLK、CRY1、NEMO和PDP1)在夏季和冬季之间的差异表达。CLK、CRY1和PDP1在夏季上调,而NEMO在冬季上调(图2A)。研究者在南极磷虾基因组中发现了25个显著扩增的基因家族(图2B)。12个直接参与蜕皮周期(6个家族)和能量代谢(6个家族)(图2C)。这些家族中的大多数基因都有表达,表明额外的基因拷贝具有功能(图2D)。编码卵黄蛋白(VTG)是无脊椎动物中一种重要的蛋黄蛋白,在能量需求旺盛的产卵季节提供营养库,包括CYSC、PFK和PKLR在内的其他能量代谢相关基因在夏季也表现出上调(图2F),PNLIPRP2的两个同源基因之一(一种消化脂肪酶基因)在冬季上调,此外,促进蜕皮和生长的基因(JHE、JHE-like CXE和CHT10)在食物供应量高的夏季上调,而抑制蜕皮的基因(JHAMT和CASP2)在冬季上调(图2F)。
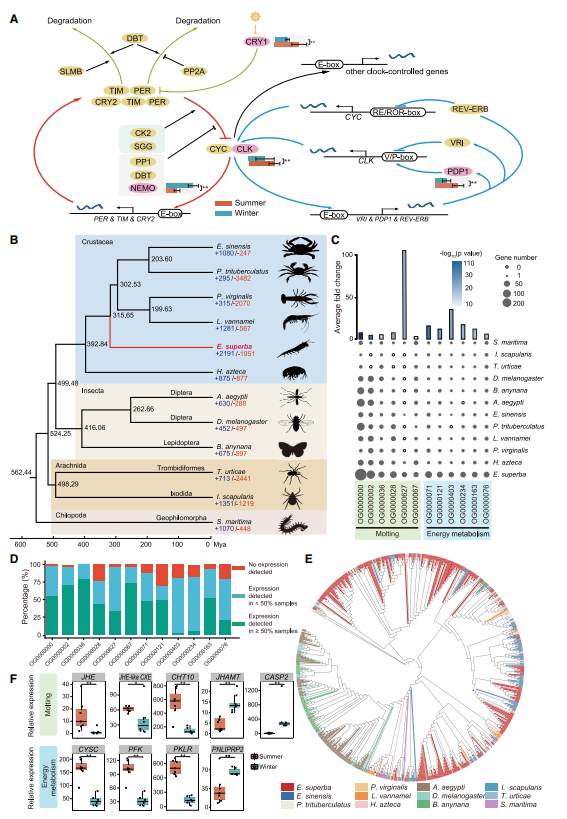
图2 适应南极海洋环境的潜在基因组变化
该研究的主要技术亮点是组装有史以来最大的动物基因组,基因组中超丰富的TR DNA加剧了这一技术挑战,成为主要的生物学发现之一。该发现揭示了南极磷虾适应南大洋的基因组基础,并为未来的南极研究提供了宝贵的资源。
(二)African lungfish genome sheds light on the vertebrate water-to-land transition
肺鱼是现存最接近四足动物的近亲,并保留了由水生向陆生过渡相关的祖先特征。现存的6种肺鱼,有4种生活在非洲,1种生活在南美,还有1种生活在澳大利亚。2个不同的研究团队分别以非洲肺鱼和澳洲肺鱼为研究对象在国际顶级期刊Cell和Nature上发表了研究成果。肺鱼基因组是迄今为止报道的最大的动物基因组(约40Gb),基因组中大量的重复序列(>60%)进一步增加组装的难度,希望组凭借领先的ONT Ultra long测序和自主开发的NextDenovo基因组组装技术分别助力两研究团队完成了高水平的基因组组装,其中,为非洲肺鱼文章提供了Nanopore测序和NextDenovo、NextPolish软件的使用,使得该超大基因组的BUSCO评估达到95%以上,武汉希望组生物科技有限公司胡江为本文的共同第一作者。

图3 非洲肺鱼
发表期刊:Cell (IF:66.85)
研究对象:非洲肺鱼
主要测序技术:Nanopore1D、BioNano和Hi-C
主要完成单位:西北工业大学生态与环境学院、中国科学院水生生物研究所淡水生态与生物技术国家重点实验室、中国科学院昆明动物研究所遗传资源与进化国家重点实验室等
第一作者:王堃、王俊、朱成龙、杨连东,任彦栋、阮珏、范广益、胡江(希望组)
希望组贡献:提供基因组测序和NextDenovo、NextPolish软件及组装技术支持
部分研究结果
01非洲肺鱼染色体基因组组装、重复序列与进化分析
研究团队利用Nanopore Ultra long、BioNano和Hi-C测序,采用NextDenovo + wtdbg2 + NextPolish策略组装,最终获得约40.05 Gb的基因组,Contig N50达到1.60 Mb;结合BioNano和Hi-C数据对基因组构建Scaffold和辅助染色体挂载,最终得到17条染色体,Scaffold N50 2.81 Gb,染色体挂载率达到99%以上。BUSCO评估显示该基因组包含了95%以上的脊椎动物完整基因。非洲肺鱼基因组如此巨大主要是由TEs的扩张引起的,非洲肺鱼基因组的61.7%(24.7 Gb)被注释为重复序列。研究团队通过分析Kimura distance估算了TE历史扩张活动,结果表明TEs,特别是反转录转座子,在过去7000万年中一直活跃。基于基因组组装和注释结果,通过对8种脊椎动物的5149个单拷贝基因进行系统发育重建,证实非洲肺鱼是与四足动物最近的姐妹谱系,非洲肺鱼和四足动物的分化时间可追溯到泥盆纪伊始,估算为419 MA。
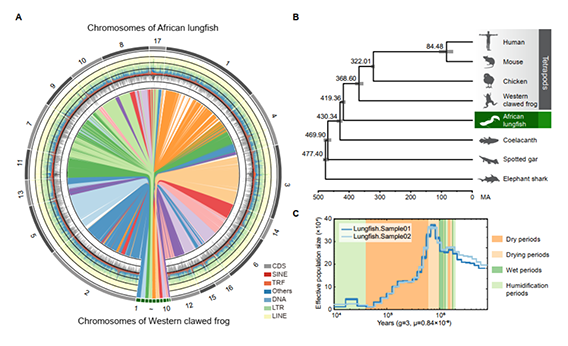
图4 非洲肺鱼染色体水平基因组组装和进化史
02 基因改变增强了呼吸能力
肺呼吸能力的进化可能经历了三个步骤:第一步是硬骨鱼的共同祖先已具备了最初级的呼吸空气的能力(已有文献支持),本研究中检测到所有硬骨鱼中存在Sftpb同样也证实这一观点。第二步是通过诸如Sftpc的出现和邻近Foxp1的保守非编码元件(CNEs)等基因创新,肉鳍鱼类的共同祖先获得了增强空气呼吸的能力。第三步可能是进一步的基因创新,包括Sftpa、Sftpd的出现以及Foxp2附近保守非编码元件(CNEs)的出现,为四足动物进化出呼吸系统提供了最后的关键基础。
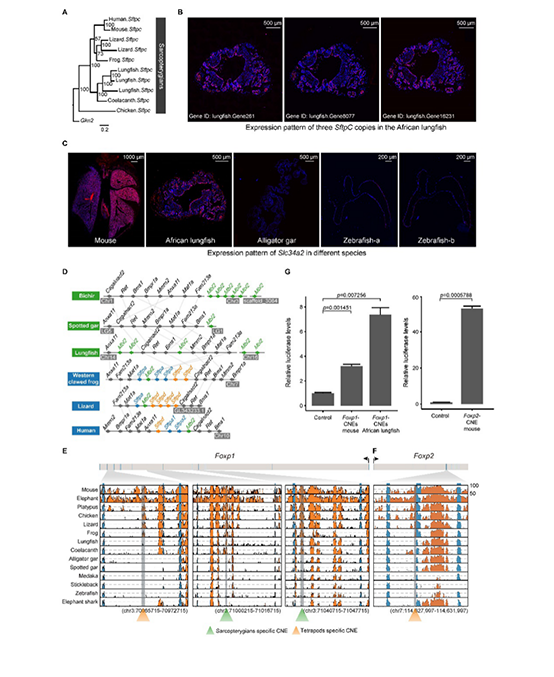
图5 肉鳍鱼类肺呼吸功能的演变
希望组作为三代测序行业的引领者,拥有完备的三代测序平台,强大的生物信息团队,拥有自主研发且在基因组组装领域被广泛应用的NextDenovo系列算法。已为众多科研院所提供优质的测序及分析服务,积累了丰富的项目经验。
欢迎拨打电话153 8703 7487
或联系您身边的科技顾问,
或发邮件至inquiry@grandomics.com咨询!





发表评论
想参加讨论吗?请尽情讨论吧!