Next系列软件应用 | NextDenovo软件脱颖而出,助力家蚕T2T基因组组装

研究思路
研究结果
1.ONT基因组组装
为了评估基因组组装的准确性,作者使用Inspector计算了结构错误和小规模错误的数量。其中NextDenovo的小规模错误数量最少,结构错误数量略低于wtdbg2(图2)。Wtdbg2具有最高的小规模错误数和最低的结构错误数。NECAT的结构误差最多,小尺度误差次之。
总之,对于ONT数据的组装,NextDenovo软件的组装效果最好。
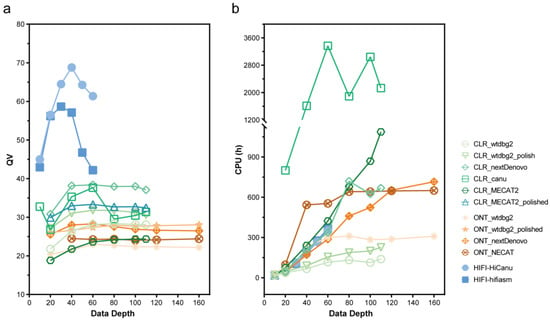
图1 不同数据深度的CLR、ONT、HIFI组装的质量值(QV)评分和计算时间
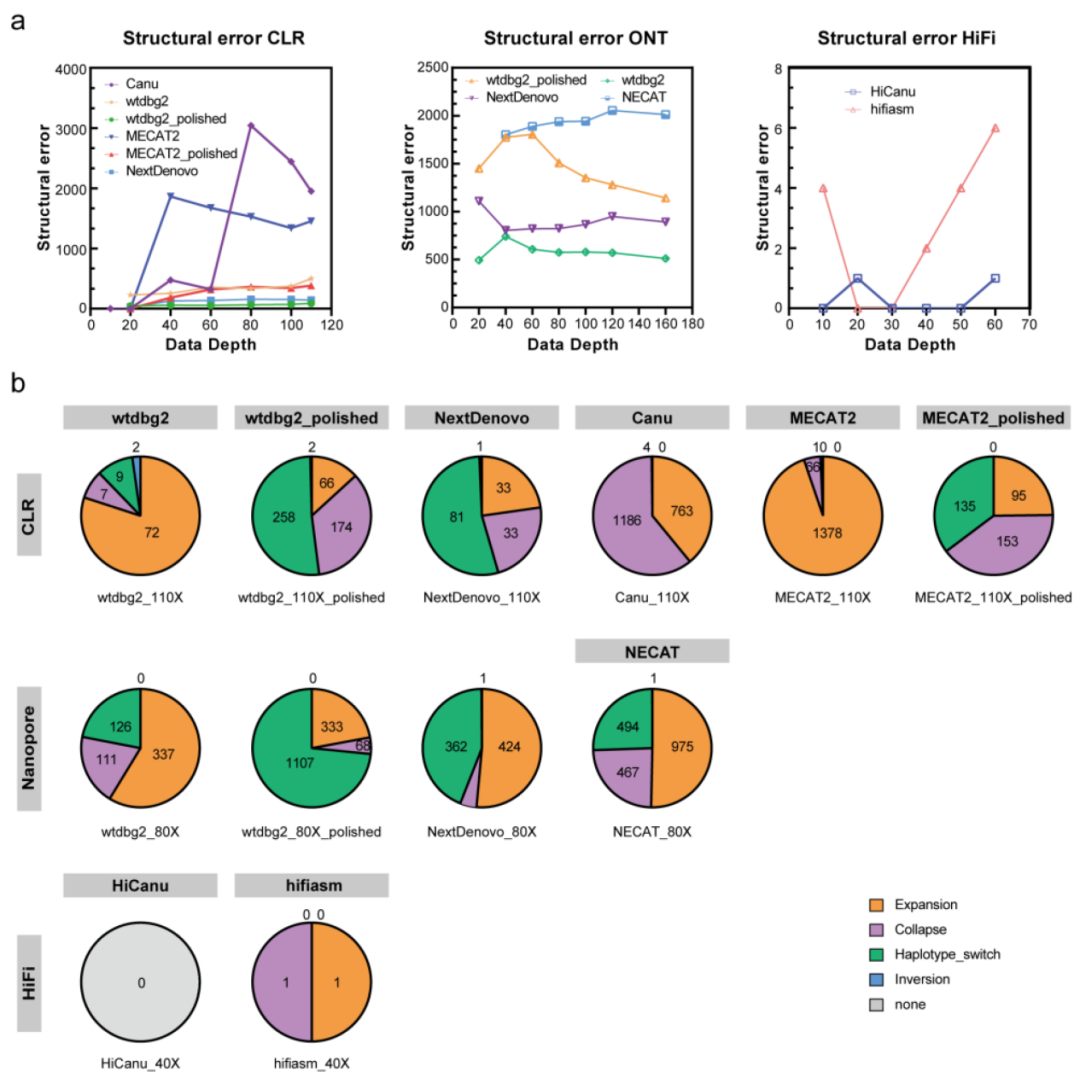
图2 CLR、ONT、HIFI组装的结构错误
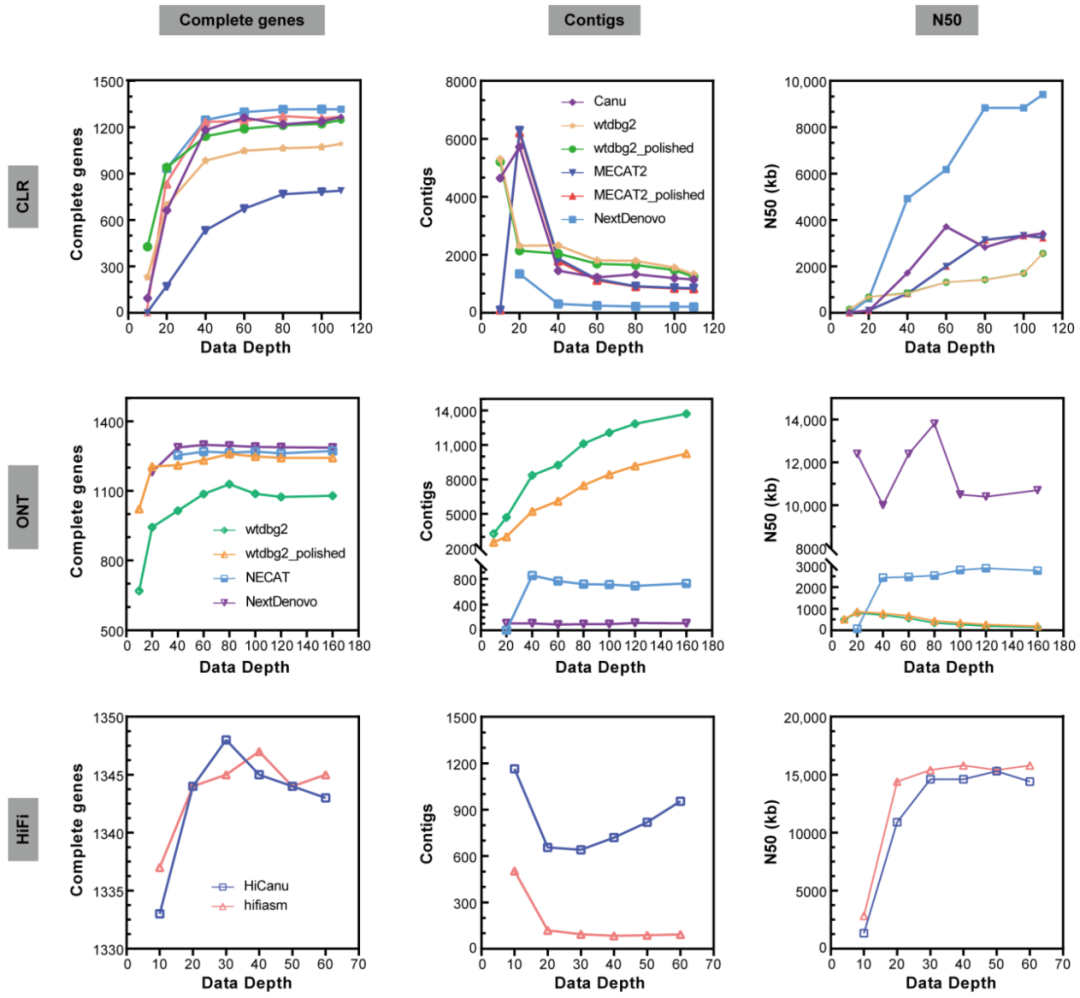
图3 测序深度对基因组组装影响
与CLR和ONT相比,HiFi组装的基因组连续性和完整性明显优于CLR和ONT。HiFi基因组组装的大小、连续性和完整性没有显著差异。最大的差异体现在contig数上,hifiasm组装的contig数目比HiCanu组装的少的多(图3)。与ONT和CLR相比,HiFi组装包含最少的结构误差和小规模误差(图2)。与其他两种测序方法相比,HiFi组装显示出最佳的组装质量、最低的contig、最高的连续性、准确性和完成度。它还需要最少的时间和计算机内存,可以被认为是未来鳞翅目害虫基因组的最佳测序方法。
作者使用3D-DNA在染色体水平上构建基因组,为每种测序方法选择了最佳的基因组组装。使用默认参数,3D-DNA实现了大多数染色体的聚类。然而,仍然存在一些染色体聚类错误和contig易位和反转,这些都是使用Hi-C图识别的。然后,作者设计了基于EagleC的染色体水平基因组组装质量评估标准。这可以快速准确地识别组织错误,并能够以表格的形式报告基因组组装中的错配百分比,以便于纠正这些组装错误(图4c)。根据EagleC的建议,完成了基因组组装的调整,并使用Racon进行了纠错,使用TGS GapCloser进行了补洞。最后,使用五个碱基端粒重复序列(’TTAG’)作为序列查询,鉴定到了50个端粒,并构建了28个假染色体用于家蚕(P50T HiFi)基因组(图4a,c)。根据EagleC的报告,这些差异区域是由几个Mb级组装错误造成的,例如Chr24(图4e)。P50T SilkBase组装中的组装错误也通过5个蚕基因组组装的Chr19平行图得到证实(图4d)。尽管CLR和ONT的基因组组装质量不如HiFi,但在使用EagleC和3D-DNA(基于Hi-C)处理后,两者都完成了非常高的连续和完整的染色体水平基因组组装(图4b)。
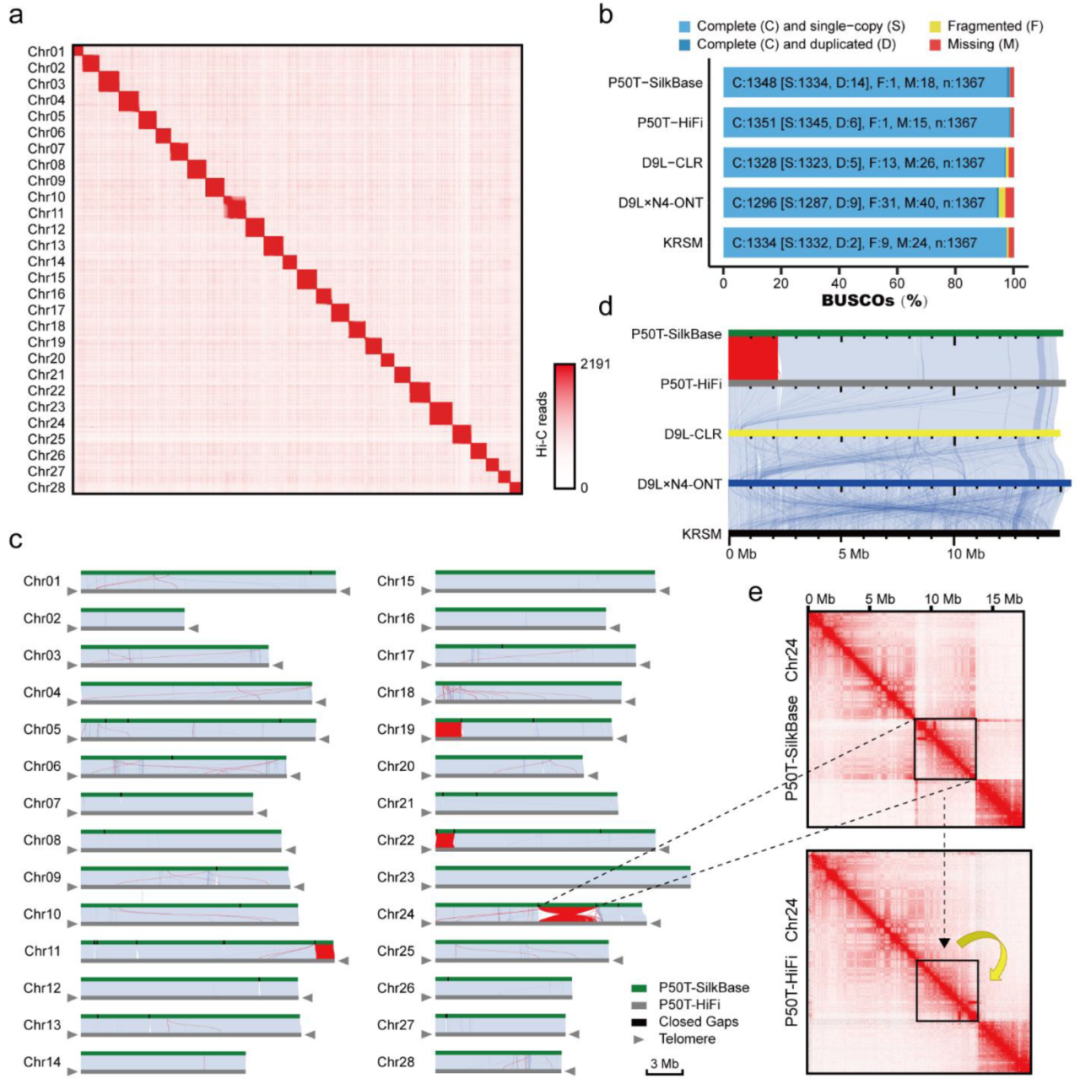
图4 不同家蚕品系染色体水平基因组组装总结






发表评论
想参加讨论吗?请尽情讨论吧!