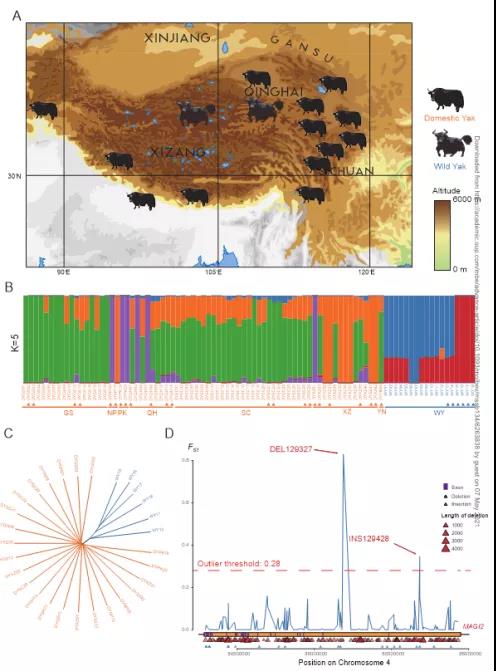
三代测序由于其长读长的特点,可以协助检测以往二代短读长测序未能发现的大的结构变异(Structural Variants,SVs)。结构变异也是研究自然选择和人工选择的重要研究手段。2021年5月3日,兰州大学刘建全团队在Molecular Biology and Evolution杂志在线发表题为“Structural variants selected during yak domestication inferred from long-read whole-genome sequencing”的文章,首先利用三代ONT长读长测序以及Hi-C测序获得了组装高度连续的高质量家养牦牛BosGru3.0的参考基因组,并通过长读长重测序数据分析,对比野生牦牛与家养牦牛的SVs,发现了一些主要与神经系统、行为、免疫和繁殖相关的基因,为高海拔地区动物的驯化提供了新的视角。希望组在本项目研究中提供了三代ONT测序服务。家畜驯化是人类文明史上的主要成就之一。驯化后的动物会有一系列的表型变化,如脑体积减小和驯养性增加等被称之为驯化综合特征。在一些家养物种中,可以通过检测单核苷酸多态性(SNPs)、短的片段插入和缺失(InDels)和拷贝数变异(CNVs)等遗传标记来挖掘潜在的遗传信息。然而,结构变异(包括插入、缺失、复制、倒位或50bp或更长的易位)的作用,由于两种技术限制,并未得到充分的研究。第一个限制,是检测SVs需要能跨越其全长的长读长测序reads。第二个限制是需要具有连续组装的参考基因组,能覆盖基因组中的重复区段。尽管由于单碱基的错误率较高,长读长测序不适合检测单核苷酸变异(SNVs),但它仍是检测大片段SVs的首选方案。在本研究中,利用三代ONT测序以及Hi-C测序,构建了一个高质量的染色体级别组装的BosGru3.0参考基因组,并通过6头野生牦牛以及23头家养牦牛的长读长重测序数据分析,构建覆盖牦牛地理范围内的372220个SVs的SV基因图谱。用于染色体级别组装的BosGru3.0参考基因组,其DNA来源于四川省红原县的一头公牦牛血液。对测序深度约88x的ONT长读长reads进行de novo组装。利用illumina短读长数据对基因组进行polish,并通过Hi-C数据,利用染色体互作强度对基因组数据进行聚类,最终得到一个组装高度连续的BosGru3.0参考基因组,拥有116条contigs,组装成了31条染色体。BosGru3.0的contig N50为44.72Mb,scaffold N50为114.39Mb,远高于其他反刍动物的参考基因组。组装完成后,对重复元件、蛋白质编码基因和非编码元件进行了预测。总共预测了21232个蛋白编码的基因。表1 BosGru3.0与BosGru2.0的基因组组装比较研究者选择了23个不同地点的家养个体和6头野生牦牛进行了全基因组长读长重测序。长读长基因组测序的N50平均长度分别达到了22.59Kb(家养)和21.99Kb(野生),有效深度从8.4x到15.6x(家养),11.4x到21.2x(野生)。最终鉴定出372220个SVs,其中包括328936个缺失,32618个插入,4321个重复,1993个倒位以及4352个易位。根据所有SVs在BosGru3.0上的位置进行了相关注释,发现257155个SVs在基因间区,93582个SVs在内含子区,14964个SVs在外显子区,1811个SVs在UTR区以及有3620个SVs在基因的上游和下游侧翼150bp处。大多数SVs(74.43%)含有重复序列。野生牦牛和家养牦牛基因组的SV序列比较显示,各重复序列占比均无明显差异。为了进一步识别可能参与驯化的SVs,研究者们计算了所有野生和家养牦牛的SVs FST值,发现了3680个SV的FST outliers值大于人为选择的0.28。在这些高FST的 SVs中,有2391个SVs(占所有SVs的0.64%)存在于基因间区,有1288个SV在外显子、内含子或725个基因的侧翼区域。其中,有34个在外显子区域有缺失,有24个导致了ORF的位移(无意义SVs)。随后对725个高FST的SVs的基因功能进行了注释,发现这些基因最显著富集的功能与神经系统发育和人类疾病,长时程抑郁相关。其他GO功能分类与神经系统相关,包括了神经元的分化,神经元的生成等。典型的例子是具有第二高FST的基因,其结构变异位于信号蛋白MAGI2的内含子中。人类MGAI2基因的缺失与癫痫和精神分裂症有关,在攻击性犬种研究中发现其MAGI2附近存在几个CNVs。其他三个高FST SV基因(GAD2、PLCB2和GRIK2)也有文献报道了类似的行为关联。其他携带SVs的基因还涉及到了免疫、解剖学形态结构以及经济性状等方面。例如,NAFI已被证实通过在T细胞中下游靶向的IL-2生长因子来调控有效的免疫调节细胞因子的表达。GSK3B是GSK3A的一个isoform,有研究发现其与猪的脂肪存储能力有关。敲除GSK3A可改善小鼠对葡萄糖的糖耐量,并提高了肝糖原的储存和胰岛素的敏感性。无意义SVs相关的一些基因也有研究表明是参与了智力或大脑的发育,例如PAX2、MAGT1以及SHROOM2和SSBP3。综上所述,SVs已参与牦牛的驯化过程,且其靶向基因优先与神经系统、行为和免疫相关。这些研究的发现为牦牛的驯化和牛的进化提供了新的见解。原文链接:https://doi.org/10.1093/molbev/msab134 
 项目文章 | 基因组研究解析波纹唇鱼进化历史并分析与性别反转相关的视蛋白基因...
项目文章 | 基因组研究解析波纹唇鱼进化历史并分析与性别反转相关的视蛋白基因... 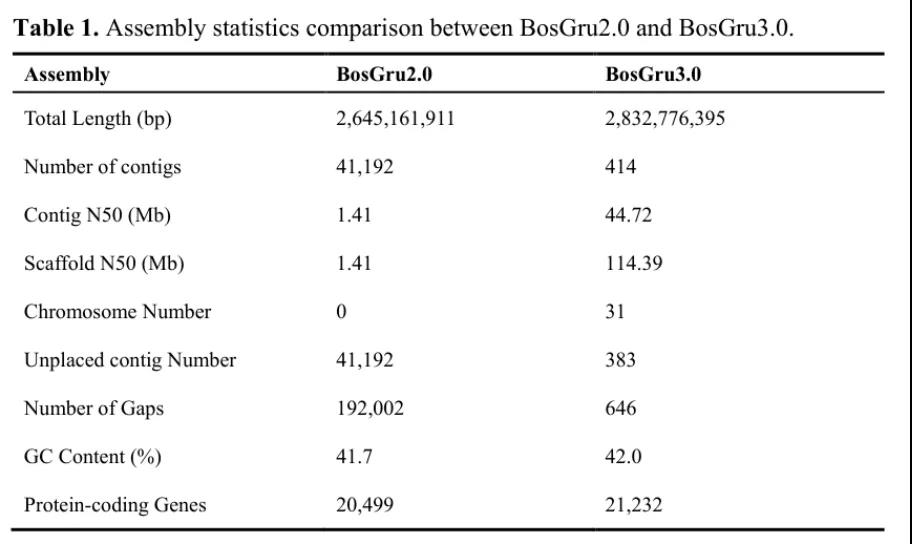



发表评论
想参加讨论吗?请尽情讨论吧!