项目文章丨利用长读长全基因测序检测牦牛驯化过程中选择的结构变异


近日来,喜讯连连!首先是2月3日,中国科学院深海科学与工程研究所何舜平研究员与西北工业大学王堃副教授的联合团队在《Molecular Biology and Evolution》上公布了高质量的剑鱼、旗鱼基因组,解析了它们的恒温机制。接着是3月1日,兰州大学草地农业生态系统国家重点实验室,生态学创新研究院杨勇志研究员团队在《Horticulture Research》上发表了毛榛染色体级别基因组,挖掘了影响榛子油脂合成的潜在基因和基因家族。希望组有幸参与两篇高质量论文的部分测序和组装工作,详细内容如下:
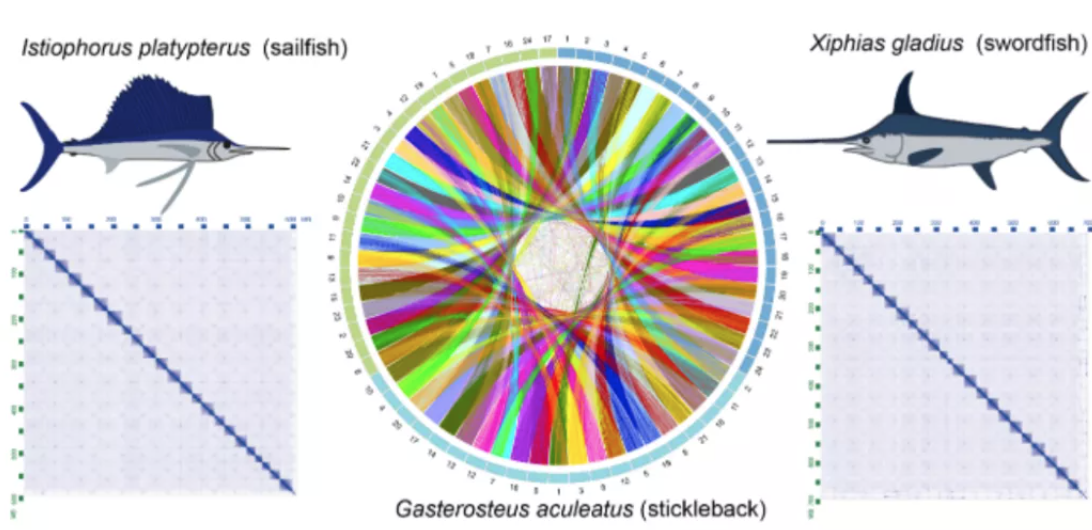
图 sailfish and swordfish
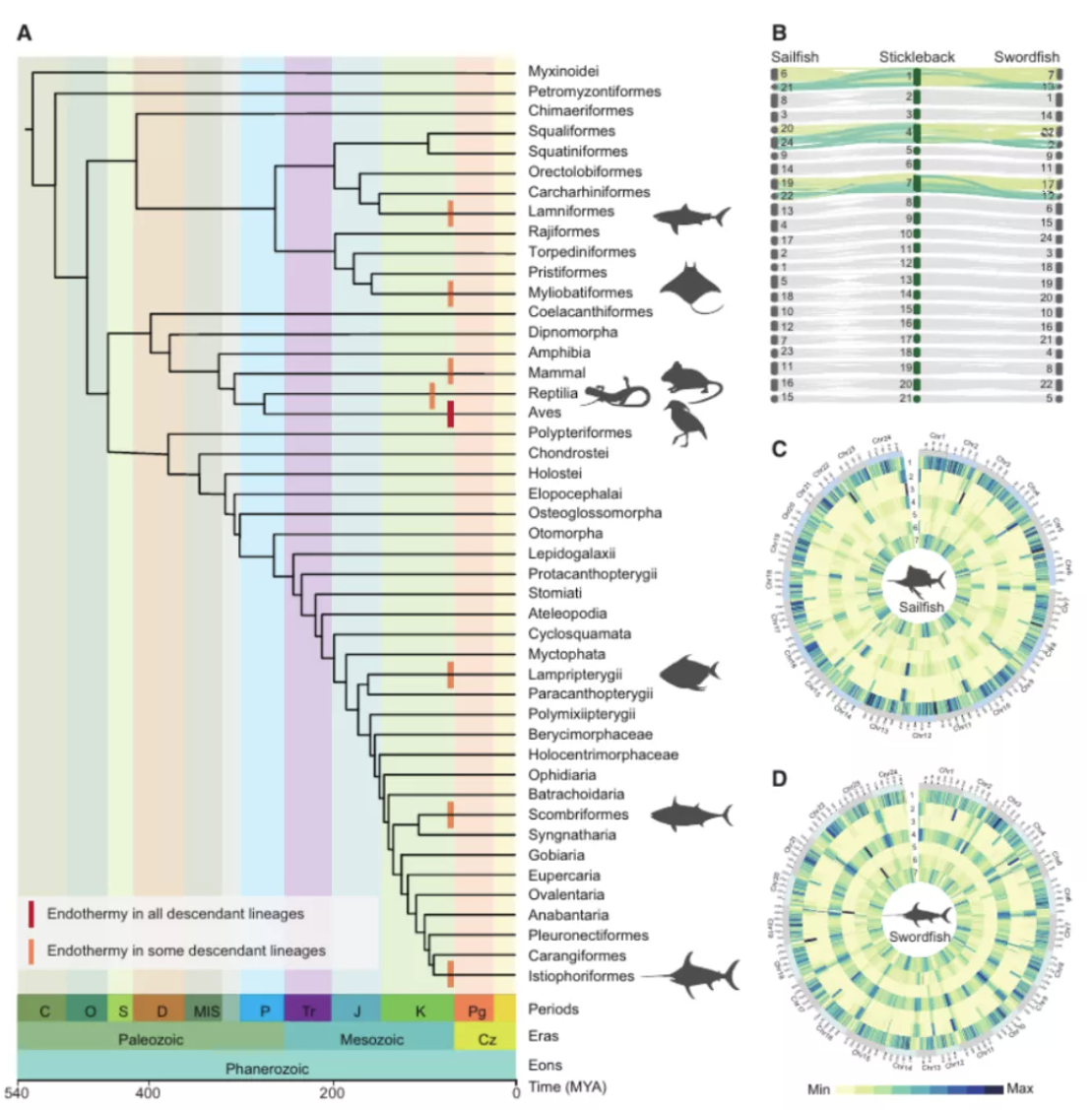
图2脊椎动物的恒温起源和两种真骨鱼的基因组组装
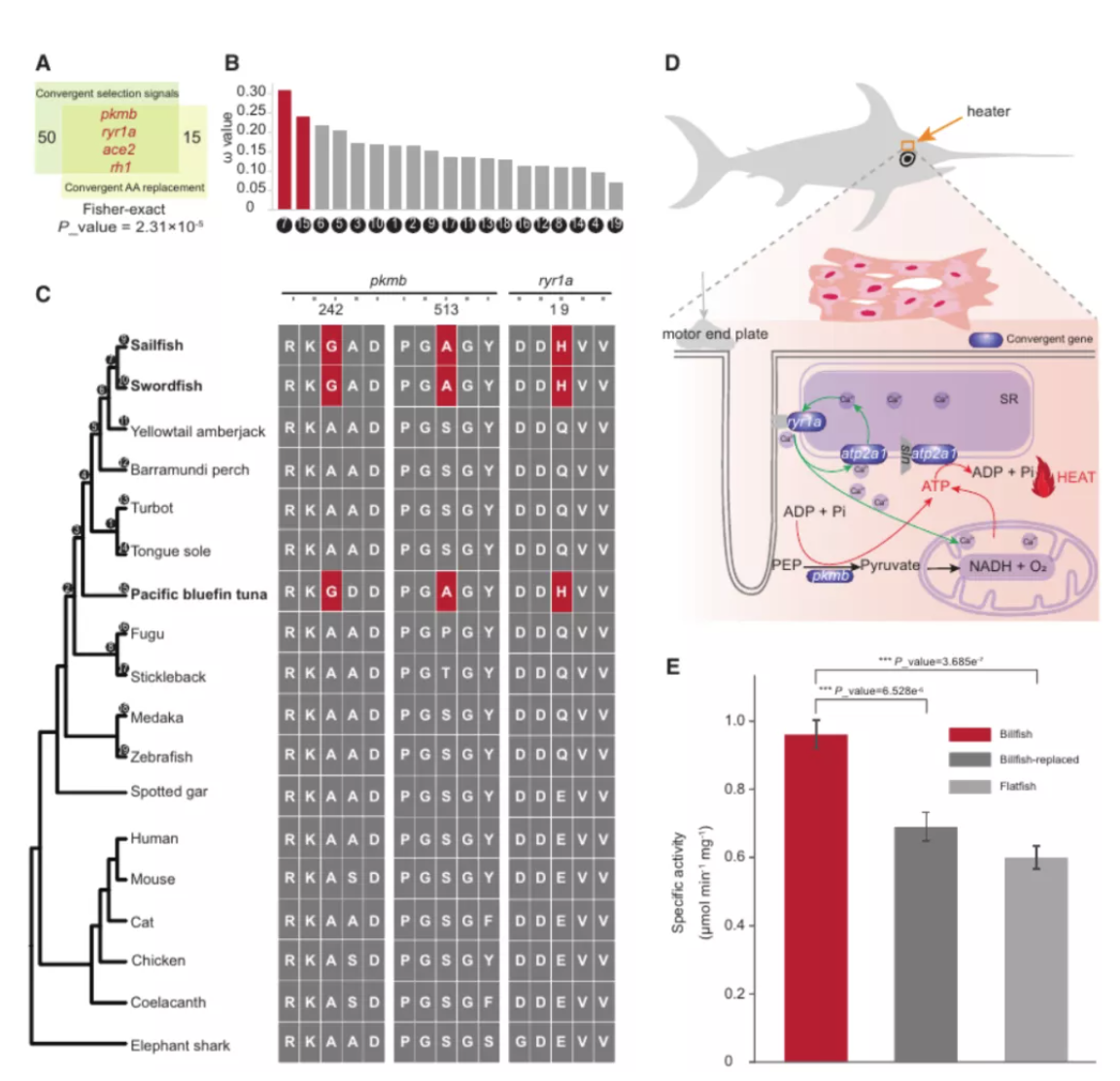
图3 真骨鱼和金枪鱼中产热的趋同进化

图1 毛榛
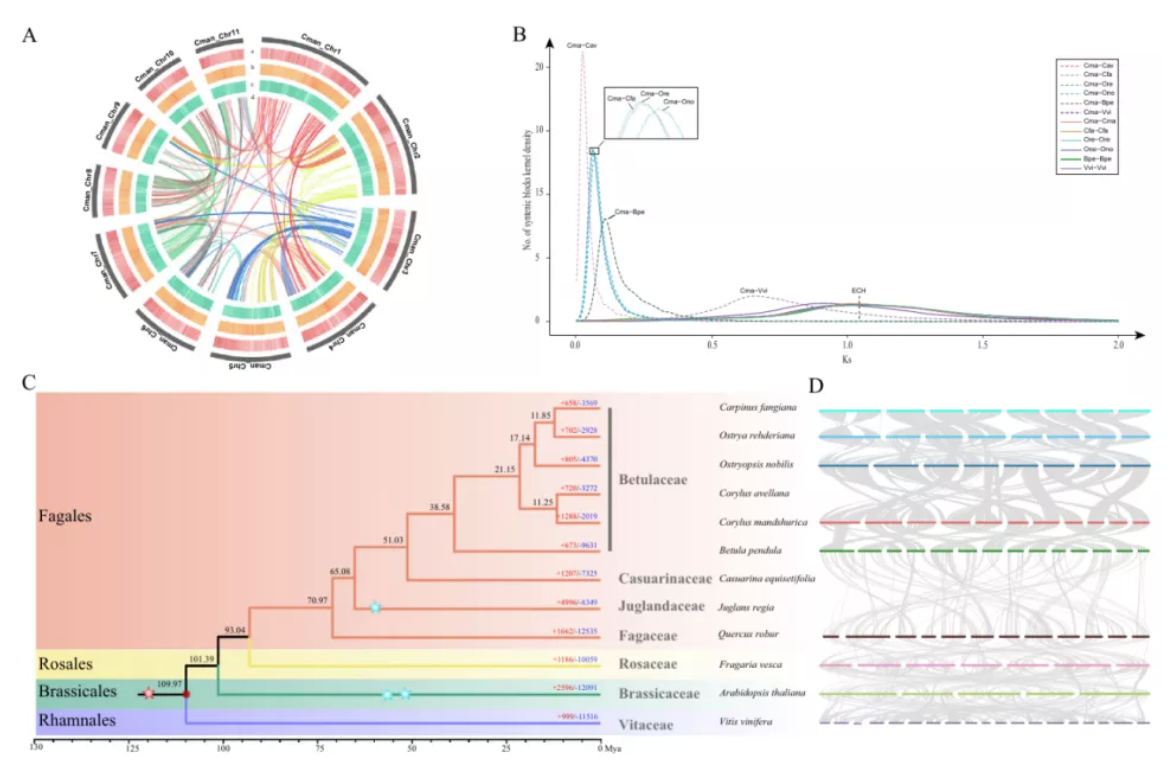
图2 毛榛 C. mandshurica 基因组特征及进化和比较基因组分析
利用比较基因组学的方法,本研究重构了桦树属基因组的进化历程,通过推断桦木科五个基因组之间的同源性,确定了毛榛的进化地位,显示毛榛基因组是由于祖先基因组经历1次染色体相互易位,2次嵌套染色体融合和1次染色体端端相连形成了目前的11对染色体核型。同时推测桦木科物种共同起源于一个有14对染色体的祖先基因组,解析了桦木科物种繁多的原因是祖先基因组发生了多次断裂和融合等事件。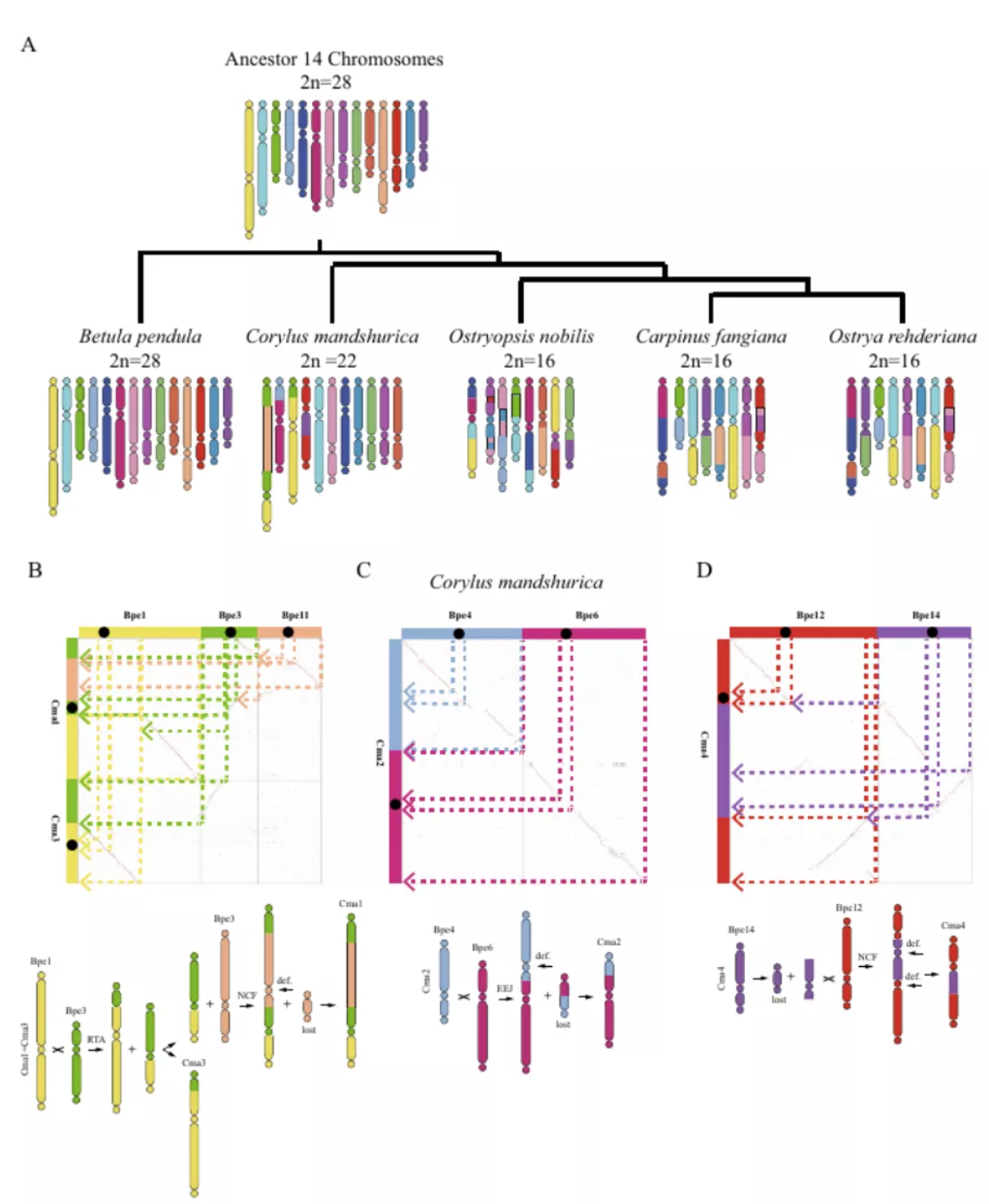
图3 桦木科五个基因组的核型进化图解和祖先核型及进化过程中的染色体融合
本研究共鉴定了96个油酸生物合成的候选基因,其中10个表现出了快速进化和正向选择特性。进化分析探究了影响榛子油脂合成的潜在基因和基因家族,鉴定了764个油脂相关基因和一些于抗逆相关的基因家族,这些抗逆基因家族在毛榛基因组中呈现快速扩张趋势。
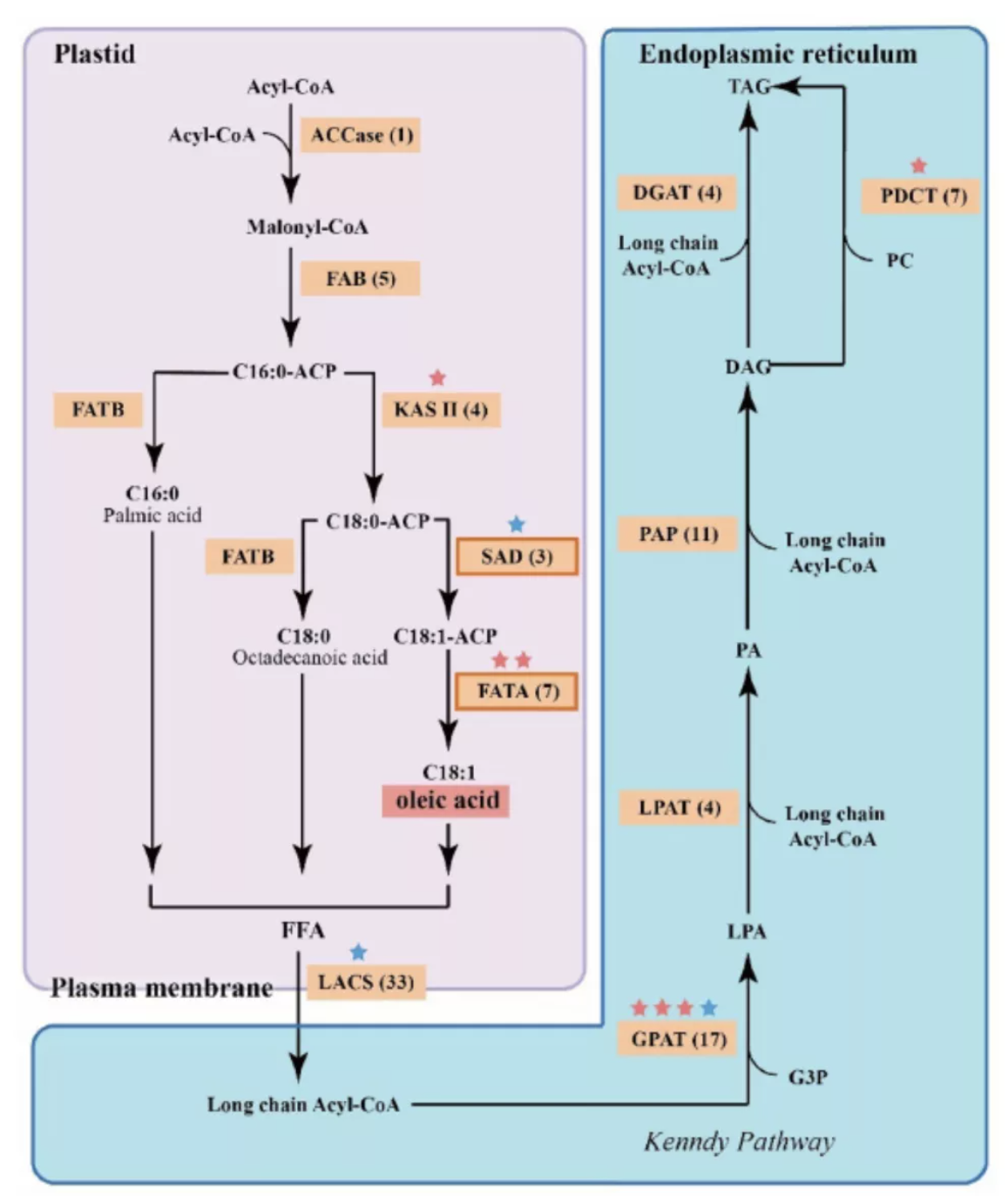
图4 毛榛基因组代谢途径和生物合成相关基因
2021.03.01,Nature Communications杂志在线发表题为“Single-molecule long-read sequencing reveals a conserved intact long RNA profile in sperm”的研究论文,由美国罗切斯特大学李鑫团队与爱荷华大学(现俄亥俄州立大学)区健辉团队合作发表。该研究利用三代测序技术检测了精子细胞中完整的 long RNAs(spiRNAs),在小鼠和人类精子中分别检测到了3440和4100种 spiRNAs。结果显示,这些spiRNAs种类上包含mRNA和long non-coding RNAs,进化上spiRNA在小鼠和人类之间是相对保守的,并且在编码核糖体的mRNAs中显示富集。该研究描述的完整long RNAs图谱为进一步研究其生物成因和功能提供了基础,同时本研究中的策略和自主开发的生物信息分析流程为其它类型样本完整longRNAs鉴定提供了参考。希望组提供了本次研究的部分三代测序服务。

文章题目:Single-molecule long-read sequencing reveals a conserved intact long RNA profile in sperm
发表期刊:Nature Communications
发表时间:2021.03.01
影响因子:12.121
测序技术:Pacbio Iso-Seq、Illumina、Nanopore cDNA全长转录组
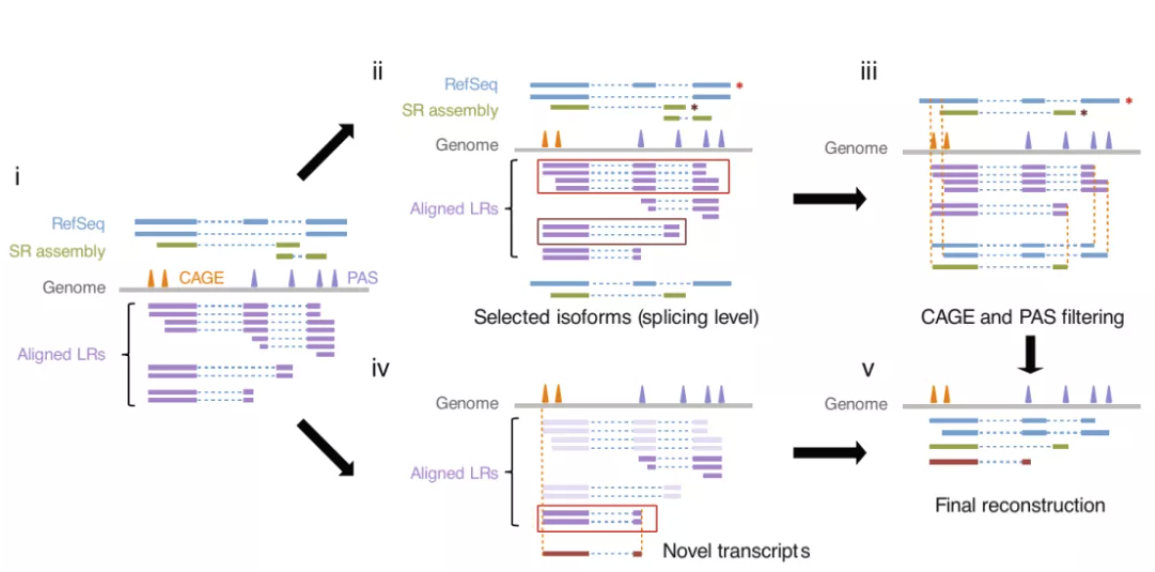
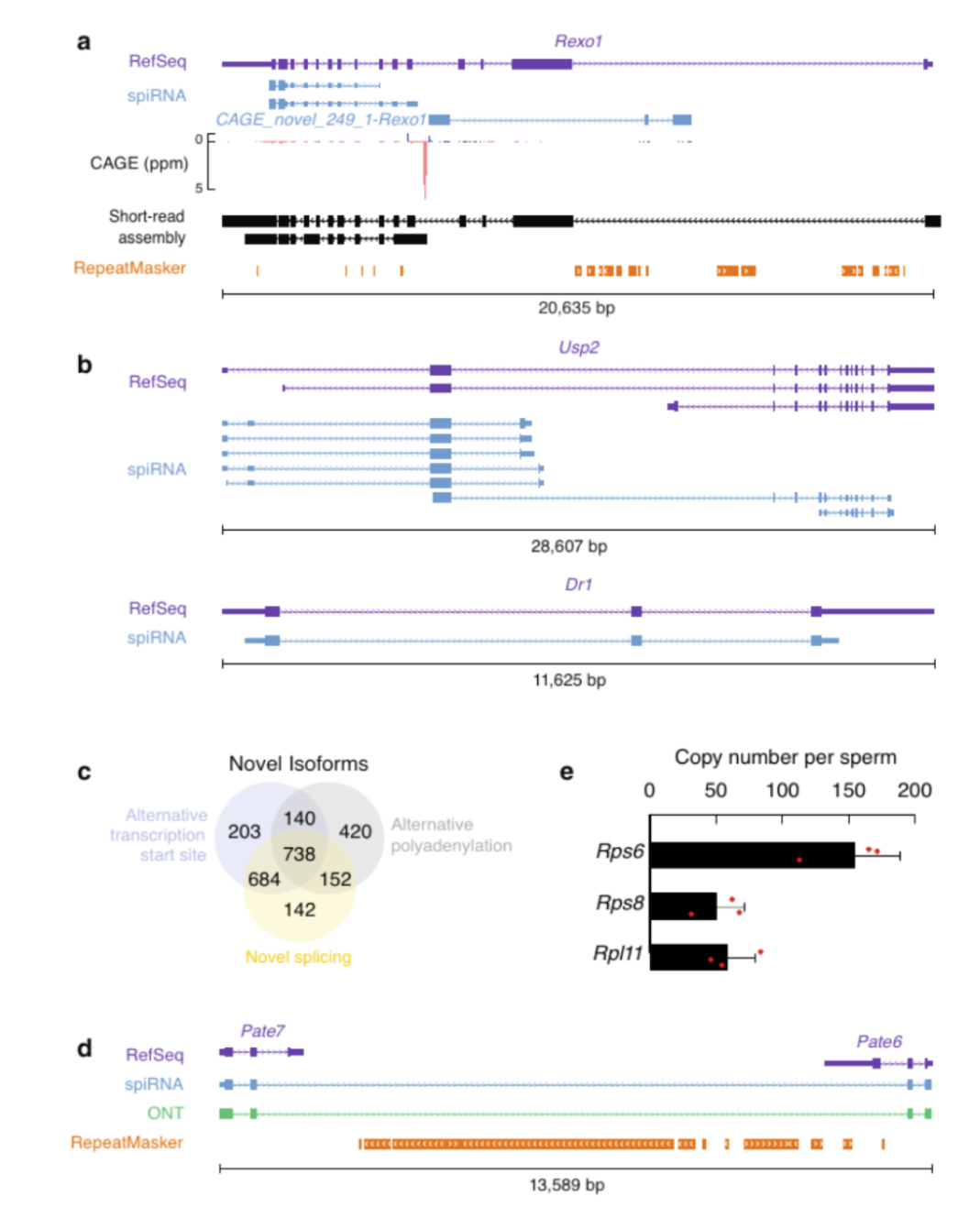
图2:精子中存在完整的long RNA转录本.
2. spiRNAs包含mRNAs和lncRNAs
为了验证spiRNAs在精子发生发生过程中的编码潜力,研究者们结合已有的Ribo-Seq数据库分析后,将小鼠的spiRNAs分成了2343个mRNAs和1097个lncRNAs,RPFs(ribosome protected fragments)在spi-mRNAs的编码区富集(图3a),并且发现在spi-mRNAs上富集的RPFs呈现出了三核苷酸的周期性 (three-nucleotide periodicity)(图3b)。此外该研究还验证了新转录本的潜在编码功能,来自已知位点的共2479个新isoforms中有1538个被注释为mRNAs, RPFs也分布在新的外显子序列中(图3c),这说明spi-mRNAs中的RPFs是可以进行翻译的。而对于来自新位点的198个新转录本,研究者们观察到78个已经注释的mRNAs和120个lncRNAs(图3d,e)之间存在明显差异,这种现象和全转录本中相似(图3a,b)。
 图3 spiRNAs include both mRNAs and lncRNAs
图3 spiRNAs include both mRNAs and lncRNAs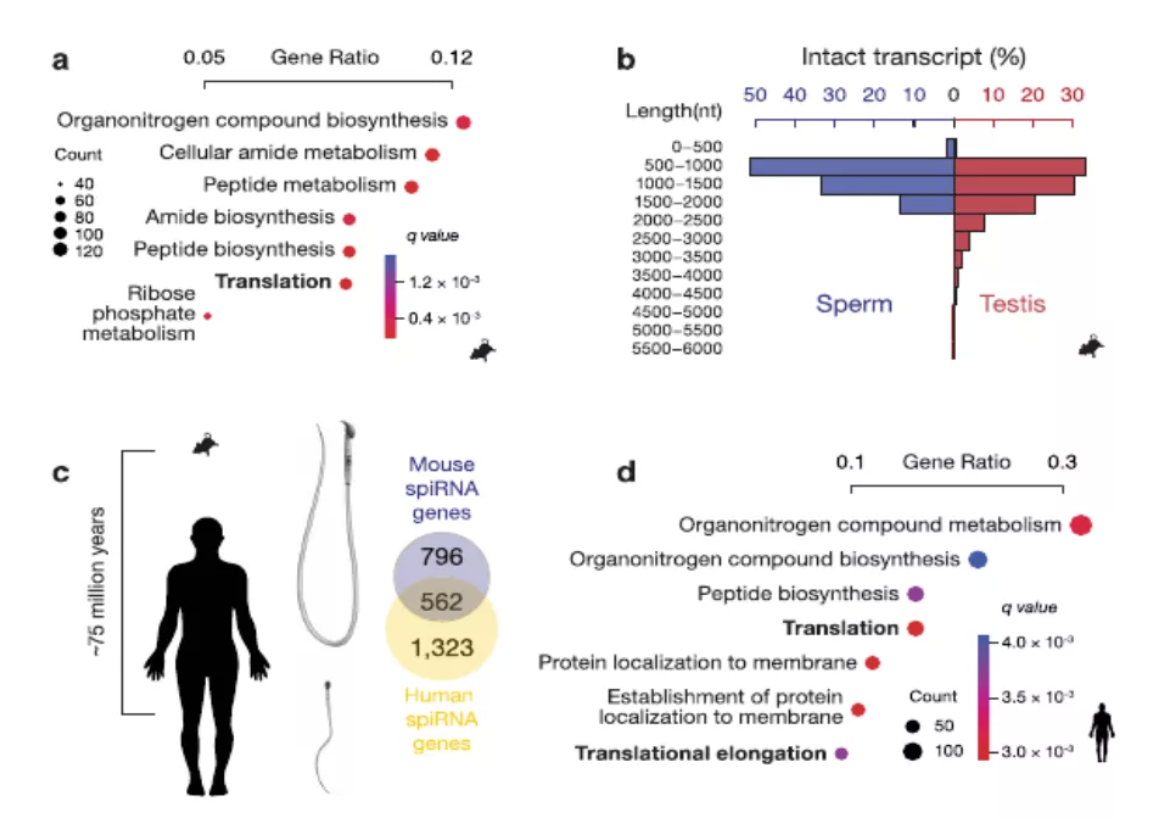
这项研究证明了精子中存在完整的 long RNAs,并在编码核糖体蛋白功能中显示富集,其功能与精巢中的RNAs不同,说明其具有一定的组织特异性。而另外发现的spiRNA在小鼠和人类中具有保守性,说明可能存在一种潜在的保守机制决定着spiRNAs序列库。
总之,该研究结合自助开发的研究策略和生物信息分析流程,揭示精子细胞中的完整RNA图谱,推动了RNA介导的表观遗传学研究,并为该领域进一步的研究提供了宝贵资源。
肺鱼是现存最接近四足动物的近亲,并保留了由水生向陆生过渡相关的祖先特征。现存的6种肺鱼,有4种生活在非洲,1种生活在南美,还有1种生活在澳大利亚。近日,2个不同的研究团队分别以非洲肺鱼和澳洲肺鱼为研究对象在国际顶级期刊Cell和Nature上发表了研究成果。肺鱼基因组是迄今为止报道的最大的动物基因组(约40Gb),基因组中大量的重复序列(>60%)进一步增加组装的难度,希望组凭借领先的ONT Ultra long测序和自主开发的NextDenovo基因组组装技术分别助力两研究团队完成了高水平的基因组组装,其中,为非洲肺鱼文章提供了Nanopore测序和NextDenovo、NextPolish软件的使用,使得该超大基因组的BUSCO评估达到95%以上,武汉希望组生物科技有限公司胡江为本文的共同第一作者;为澳洲肺鱼文章提供了Nanopore Ultra-long测序服务。

图1 非洲肺鱼
发表期刊:Cell (IF:38.637)
研究对象:非洲肺鱼
主要测序技术:Nanopore1D、BioNano和Hi-C
主要完成单位:西北工业大学生态与环境学院、中国科学院水生生物研究所淡水生态与生物技术国家重点实验室、中国科学院昆明动物研究所遗传资源与进化国家重点实验室等
第一作者:王堃、王俊、朱成龙、杨连东,任彦栋、阮珏、范广益、胡江(希望组)
通讯作者:王文、何舜平、邱强、赵文明
希望组贡献:提供基因组测序和NextDenovo、NextPolish软件及组装技术支持
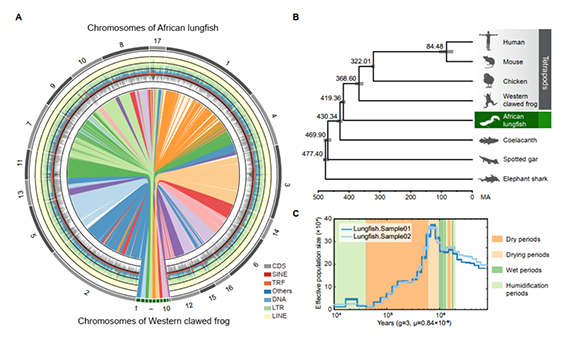
图2 非洲肺鱼染色体水平基因组组装和进化史
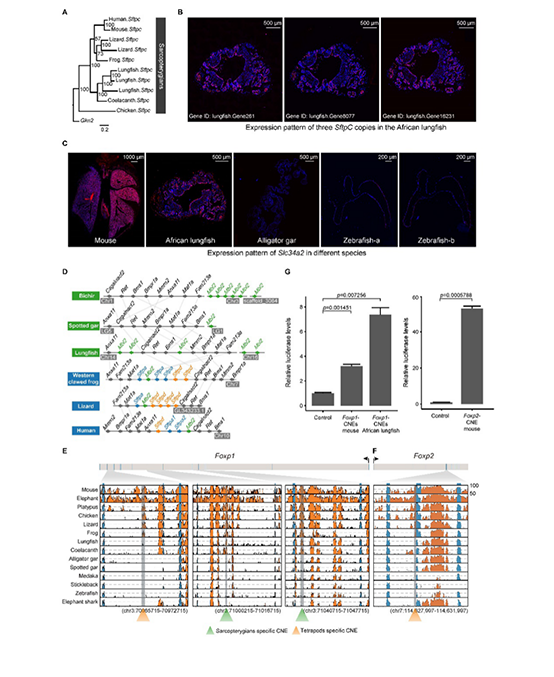
图3 肉鳍鱼类肺呼吸功能的演变
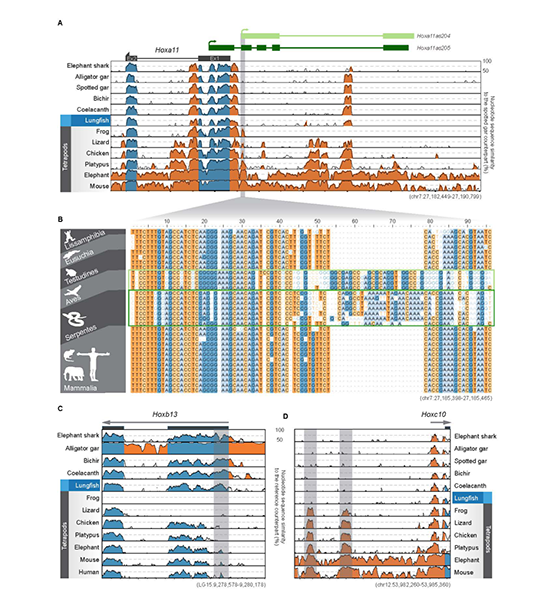
图4 肢起源和与陆地运动相关的基因变化
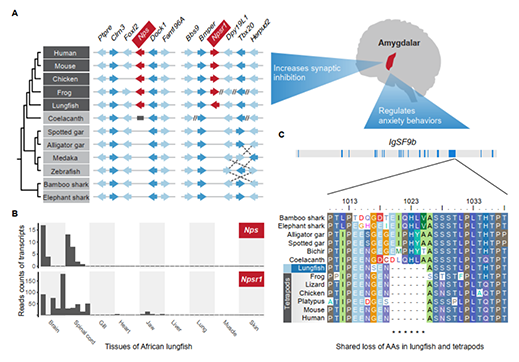
图5 与杏仁核和抗焦虑能力相关的基因改变
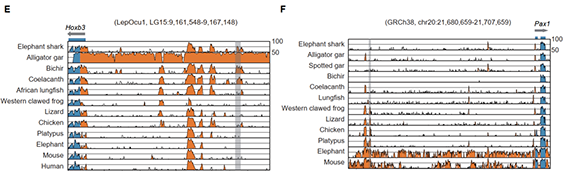
图6 与咽重塑相关的基因变化
研究对象:澳洲肺鱼
主要测序技术:Nanopore1D 、Ultra long和Hi-C
主要完成单位:德国康斯坦茨大学生物系、德国维尔茨堡大学生物中心等
第一作者:Axel Meyer、Siegfried Schloissnig、Paolo Franchini、Kang Du、Joost Woltering
通讯作者:Axel Meyer、Manfred Schartl、Elly M. Tanaka、Thorsten Burmester、Oleg Simakov
希望组贡献:提供NanoporeUltra-long测序服务

图7 澳洲肺鱼
图8 肉叶鳍的预适应调节与hoxd基因调控
总之,这2篇高水平的基因组文章分别以非洲肺鱼、澳洲肺鱼为研究对象,分析了肺鱼类的物种和基因组特点,并从不同视角推演了脊椎动物从水生到陆生进化过程,增加了我们对脊椎动物陆生进化的理解,为鱼类到人类的进化提供了宝贵的资源。
2009年首个单细胞转录组测序技术问世,开启了单细胞组学时代(scRNA-seq)(Tang et al., 2009)。过去十余年间单细胞测序技术的不断发展极大地加速了生物医学领域的相关研究,帮助科研人员克服了稀有生物样本以及生物样本内生异质性等重大挑战,一系列模式生物及人类自身的单细胞转录组图谱也由此诞生。然而目前的单细胞测序技术几乎全都是基于二代测序平台,测序读长短,一般在150bp左右,即使采用双端测序技术,测得的有效读长也不超过500bp。而人类转录组中转录本的长度普遍在1000bp以上,有些转录本长度甚至超过100kb(Piovesan et al., 2016; Frankish et al., 2019),远远超过二代测序方法所能检测的最大读长。
为了解决基于二代测序平台的单细胞转录组测序技术难以获得单个细胞中全长转录本的准确信息这一核心困难, 2020年12月30日,北京大学未来基因诊断高精尖创新中心、生物医学前沿创新中心汤富酬课题组与北京希望组生物科技有限公司合作在Plos Biology上在线发表了题为“Single-cell RNA-seq analysis of mouse preimplantation embryos by third-generation sequencing”的研究论文。该研究的主要突破有:
1)开发了一种基于三代单分子测序平台的高灵敏度单细胞转录组测序方法—SCAN-seq (Single cell amplification and sequencing of full-length RNAs by Nanopore platform),能够在单细胞分辨率直接获取全长转录本序列信息,表现出高灵敏度和高稳健性,在小鼠胚胎干细胞每个单细胞中可以检测到8000多个基因的表达,与之前基于二代测序平台最灵敏的单细胞转录组测序方法不相上下(如图1所示)。
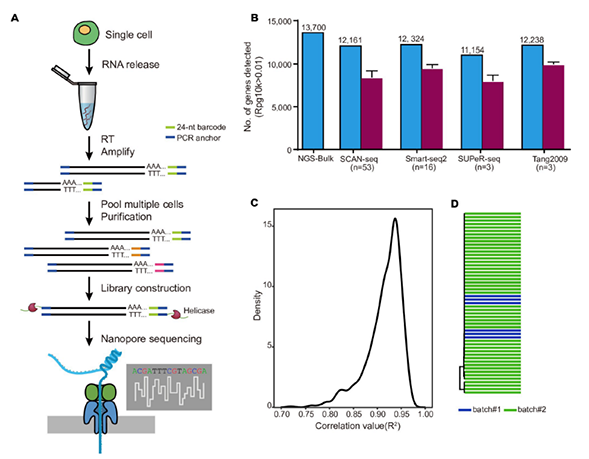
图1 SCAN-seq的流程和评估
2)鉴定出了30000多种全新的转录本。总共只测序了200多个单细胞就在小鼠胚胎干细胞(mESCs)和小鼠植入前胚胎中分别鉴定出6487条和27250种新转录本。相比基于二代测序平台的所有单细胞转录组测序方法,SCAN-seq能够区分新找到的转录本是来自同一已知转录本的新转录本,还是来自不同已知转录本已注释剪接点的重新组合的新转录本(如图2所示)。
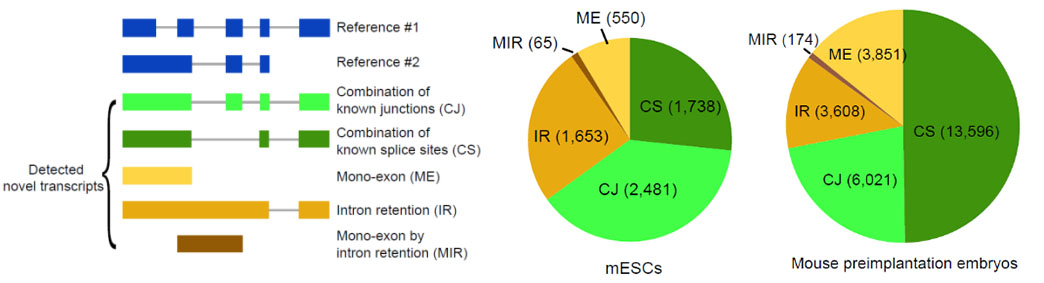
图2 未注释转录本的鉴定
3)首次提出单细胞转录组三代测序数据可以将一个单细胞中的父母源转录本准确区分开、分别进行精准定量分析。SCAN-seq显示出在同一个单细胞中精准识别小鼠品系特异性单核苷酸多态性(SNPs)的能力,平均误差率只有1.8%。利用这一方法,在单细胞分辨率确认了小鼠2-细胞期后的胚胎细胞中父源等位基因的mRNA比例逐渐增加,到囊胚期时每个胚胎细胞中来自母源和父源等位基因的mRNA拷贝数变得相当(如图3所示)。
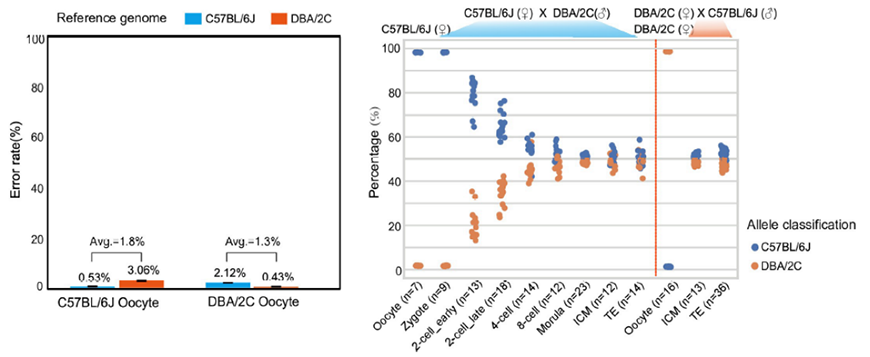
图3 等位基因特异性转录本的分析
该研究开发出的SCAN-seq新方法具有广阔的应用前景,能够克服单细胞转录组二代测序方法的各种局限性,将单细胞组学测序从“二”时代推进到“三”时代:(1)从一般只能测序单细胞中cDNA一端的有限信息,提升到能够测序单细胞中cDNA的全长信息;(2)从单细胞中一个基因的所有不同可变剪接产物(转录本)混合测量无法区分,提升到把单细胞中每个基因的所有不同可变剪接产物(转录本)精准分开;(3)从单细胞中一个基因的父母源表达信息混合在一起无法区分,提升到把单细胞中每个基因的父母源转录本精准分开;(4)从只能在单细胞中检测独特序列基因的转录本信息,提升到同时也能精准检测单细胞中高度重复序列的转录本信息;(5)从“一个基因,一个表型”的精度(one gene, one phenotype;人类基因组中有大约3万个基因),提升到“一种基因可变剪接转录本,一个表型”的精度(one RNA isoform, one phenotype;人类基因组中有大约30万种不同的可变剪接转录本)。总之,单细胞转录组三代单分子测序技术将揭开更多的转录组中“暗物质”的奥秘,给人类生物医学研究带来全新的发展机遇。
生物岛实验室研究员范小英、北京大学生命科学学院博士生廖雨涵和北京希望组生物科技有限公司汤冬硕士、李丕栋硕士为该论文的并列第一作者。北京大学未来基因诊断高精尖创新中心、生物医学前沿创新中心汤富酬教授与北京希望组王洋博士为该论文的共同通讯作者。该研究项目得到了国家自然科学基金委、北京市科技委和北京大学未来基因诊断高精尖创新中心的支持。
希望组作为三代测序的引领者,一直深耕三代测序领域,引进国际先进的PacBio Sequel II、ONT PromethION 48、MGISEQ2000、Bionano Saphyr光学图谱等技术平台,为科学研究和临床检测等提供多平台多水平的测序分析服务。利用单细胞结合三代测序平台,获取全长转录组信息,可为研究“一种基因可变剪接转录本,一个表型”打下夯实的基础。欲详细了解单细胞转录组三代测序服务及更多应用场景,可邮件联系sales-support@grandomics.com或联系希望组当地销售顾问。
参考文献:
Piovesan, A., Caracausi, M., Antonaros, F., Pelleri, M. C., & Vitale, L. (2016). GeneBase 1.1: A tool to summarise data from NCBI Gene datasets and its application to an update of human gene statistics. Database (Oxford), 2016, baw153.
Frankish, A., Diekhans, M., Ferreira, A. M., Johnson, R., Jungreis, I., Loveland, J., et al. (2019). GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 47, D766–D773.
近日,兰州大学草地农业科技学院联合广西畜牧研究所及国际家畜研究所共同合作的象草基因组研究成果以“The elephant grass (Cenchrus purpureus) genome provides insights into anthocyanidin accumulation and fast growth”为题在国际知名期刊《Molecular Ecology Resources》(3年IF=7.15)在线发表。希望组科技服务为本研究提供了Illumina、Nanopore和Hi-C测序服务,承担了基因组的组装及注释任务。该研究首次报道了象草的高质量染色体级别基因组,明确了象草的进化地位,在基因水平解析了紫色品种象草 “紫色”花青素积累的机制,并提出C4光合作用和激素信号转导通路的扩张可能有助于象草快速生长的新见解[1]。

象草(Cenchrus purpureus Schumach)因大象爱采食而得名,是禾本科、黍族多年生大型草本植物,原产于亚洲。象草因其具有生物量大、生长快速、适应性强等特点,被用作重要的饲草作物在全世界热带及亚热带被广泛种植。此外,由于象草在生物能方面的优势也使其潜在的能源草。该研究是对象草研究的重大突破,为象草进化、性状改良和功能基因研究提供了理论基础。

图1 紫色象草
研究团队以紫色象草(Cenchrus purpureus cv. Purple)为材料,K-mer评估显示象草具有较高杂合(1.5%)。利用Illumina、Nanopore、Hi-C测序。采用NextDenovo + SMARTdenovo策略组装获得1.97Gb的基因组, Contig N50 为1.83Mb,最长Contig达到15.1Mb。结合Hi-C数据对基因组辅助染色体挂载及遗传连锁图谱,得到14条染色体,挂在率为96.65%。BUSCO评估结果达 97.8%,预测注释基因65,927个。
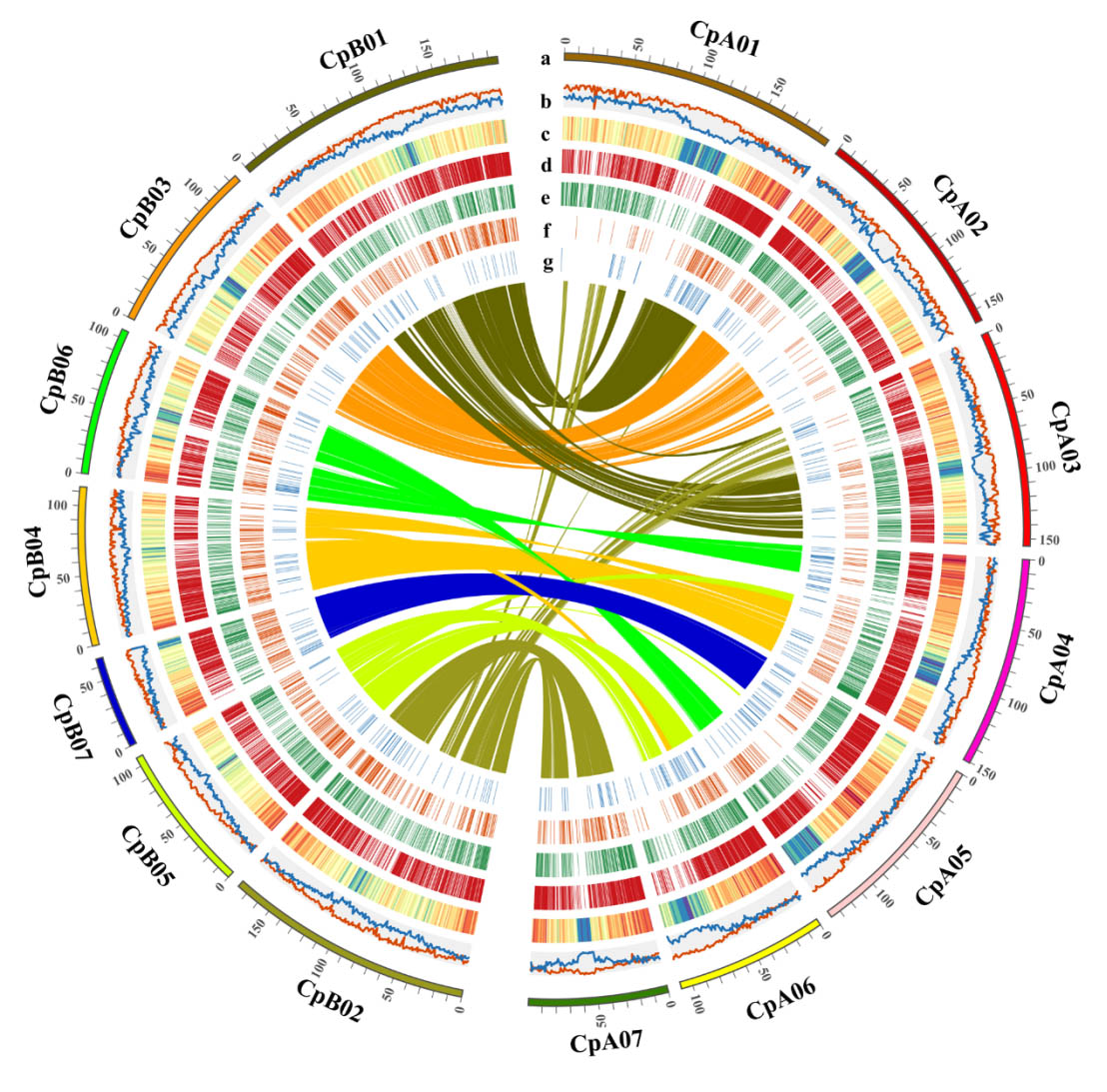
图2 象草亚基因组特征
象草为异源四倍体(2n=4x=28),包含A’和B两个亚基因组。研究表明同属二倍体植物珍珠粟(Cenchrus americanus,2n=2x=14)的A基因组与象草A’基因组具有更高同源性。通过共线性分析研究者成功将象草的A’和B两个亚组区分开来,并利用单拷贝基因分析证明象草A’亚基因组和珍珠粟A基因组具有较近的同源性。象草A’A’BB的异源四倍体基因组大约起源于6.61 (4.11-10.92)MYA,并发生了较大的染色体重组。此外,研究者还利用转录组分析了象草亚基因组显性表达,结果表明其可能行使不同的功能。

图3 紫色象草花青素积累机制
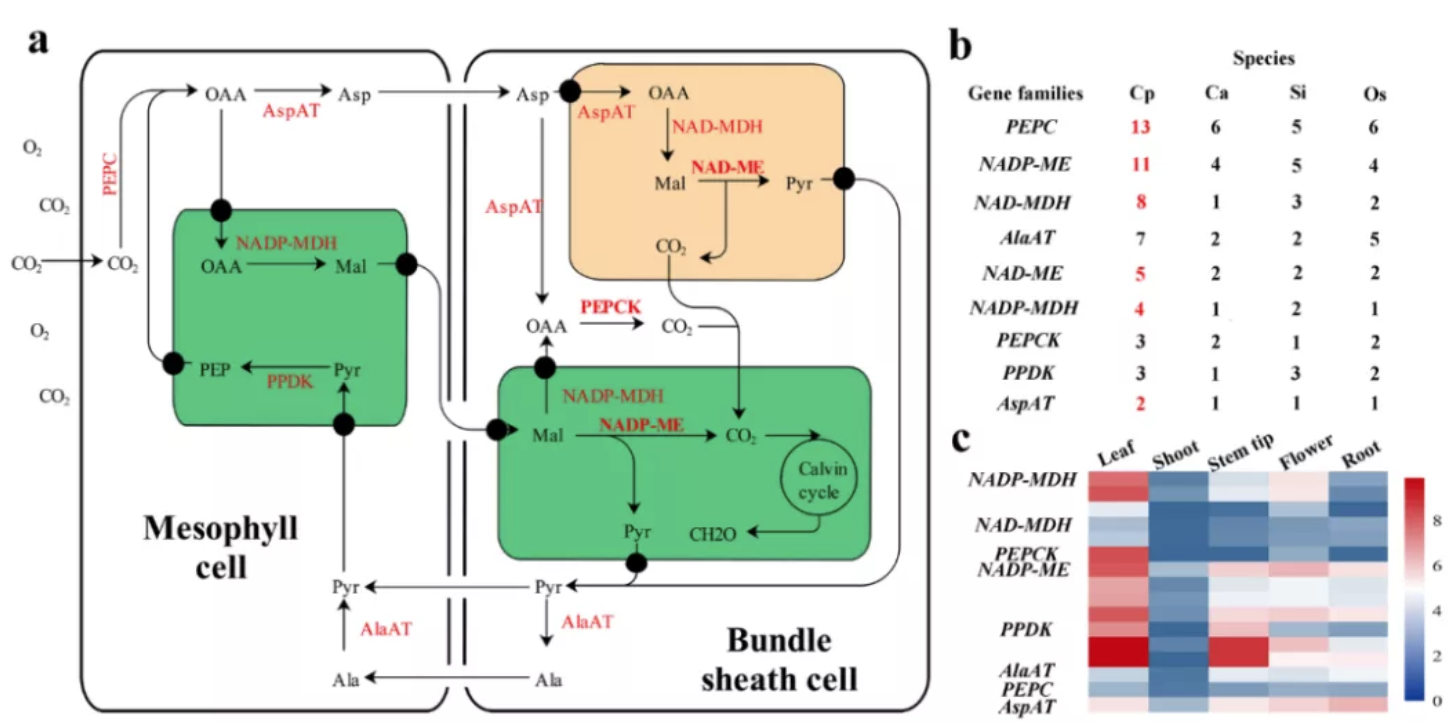
图4 象草C4光合途径
该研究利用报道的高质量的象草基因组、解析了花青素合成及快速生长机制,为象草作为优良饲草和潜在能源草的分子改良育种具有重要意义。此外,对于该属的进化以及其它物种的开发利用提供了重要资源。兰州大学草地农业科技学院张吉宇教授为通讯作者、广西畜牧研究所易显凤研究员、国际家畜研究所Jones Chris博士为共同通讯作者。兰州大学草地农业科技学院博士生闫启为第一作者、团队博士生吴凡、许攀和希望组孙宗毅为共同第一作者。
1. Yan Q, Wu F, Xu P, Sun ZY, Li J, Gao LJ, Lu LY, Chen DD, Muktar M, Jones C, Yi XF, Zhang JY. The elephant grass (Cenchrus purpureus) genome provides insights into anthocyanidin accumulation and fast growth. Mol Ecol Resour 2020, doi:10.1111/1755-0998.13271

1 特征数据统计
三代数据共得到1161468个ROI,其中72.2%是全长非嵌合序列。47%的全长序列唯一比对到基因组,三代测到的转录本平均长度为2487 bp,明显长于基因组上已有注释的转录本平均长度。矫正后,单碱基错误率降至 1.50%( 0.26% insertions, 0.27% deletions and 0.97% mismatches),校正后,BUSCO比对的完整性提升到83%。
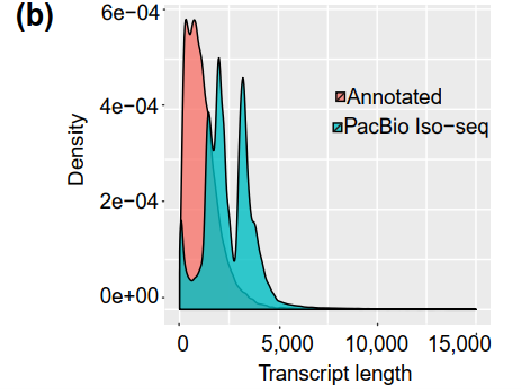
图1 转录本平均长度
2 已有数据横向比较
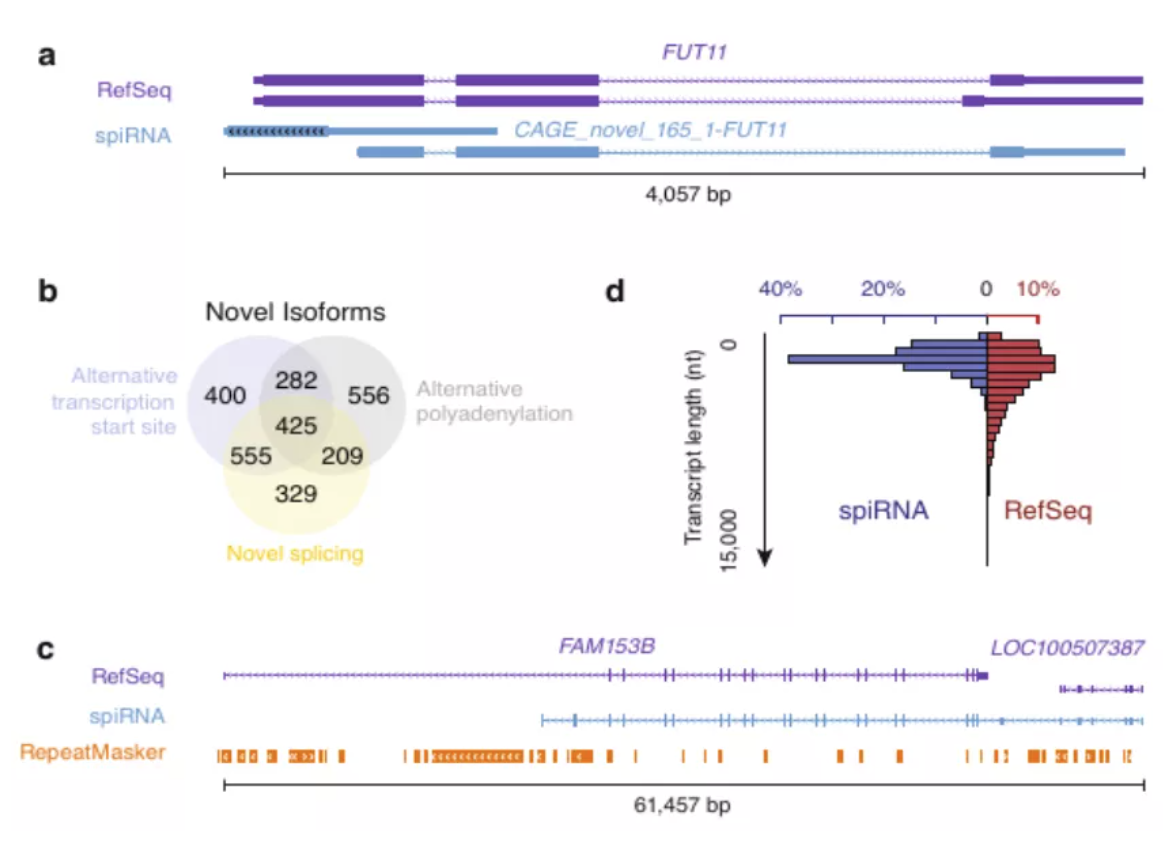
将Ensembl Plants Database中已有的cDNA序列与本次测得序列比较,数据库中的26346个序列与PacBio测得的63714个序列匹配上,且PacBio测到的全长cDNA更长。将非冗余的147698个转录本和之前已经测序的Darmor-bzh进行比较,发现有142476个转录本能够覆盖到37403个基因位点,其中31392个基因位点是多外显子基因。未比对上这个基因组的5222个转录本中,有4947个转录本可以比对到近源物种(拟南芥,白菜,甘蓝),这表明有些转录本可能是栽培种ZS11特有的。
3 可变剪接
共检测到222061个可变剪接事件,来自15068个基因位点,主要是内含子保留(IR),其中128967个转录本是现有基因组上未注释到的。统计显示,20230个多外显子基因有用多个剪接异构体,其中5761个基因能够产生5种以上异构体。比如,BnaC01g03120D在基因组注释上仅有1个转录本,但是PacBio测到了14个不同的剪接异构体。另外发现,可变剪接在An亚基因组中更为普遍。

图2 BnaC01g03120D转录本可视化
4 LncRNA鉴定及验证
鉴定到20个已知lncRNA,529个新lncRNA,平均长度1.7 kb,lncRNA具有明显组织特异性。两个亚基因组中的同源基因分别产生了54和53个lncRNA,结果表明两个亚基因组的贡献是相等的。
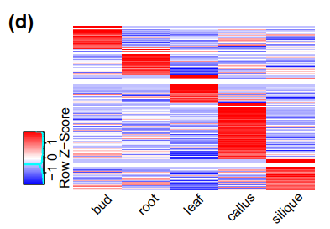
图3 各样品中lncRNA的表达量
5 APA分析
分析poly(A)位点的侧翼序列,发现上游富集尿嘧啶(U)和下游富集腺嘌呤(a)的核苷酸偏好明显。在polyA的上游,我们鉴定到了两个保守的加A信号,AAUAAA和UGUA。从两个亚基因组得同源基因对中分别鉴定到13812和14184个poly(A)位点,3299和3522个APA基因。An亚基因组的同源基因对polyA位点产生的贡献小于Cn亚基因组的同源基因。
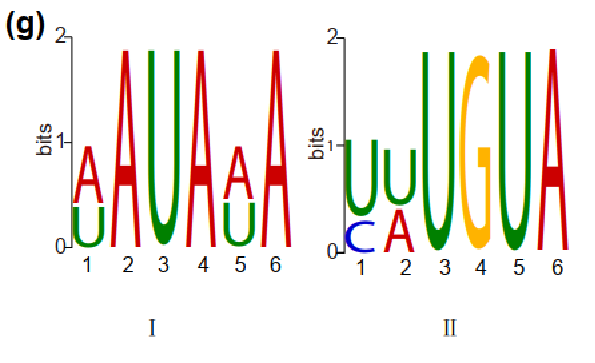
图4 MEME分析转录本中的poly(A)信号
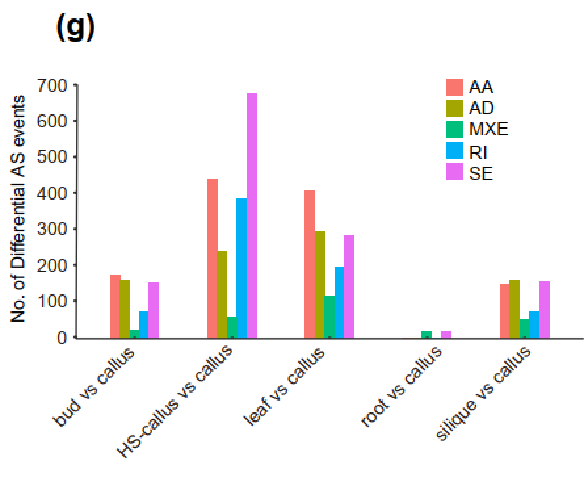
图5 各组间差异AS事件统计

希望组最新引进Sequel II,拥有成熟分析流程,更多方案设计和前沿资讯,欢迎垂询!
西双版纳植物园孙永帅团队在Nature Communications发表了题为Genomic basis of homoploid hybrid speciation within chestnut trees的研究论文,该研究以中国特有的峨眉锥栗研究系统为材料,应用进化生态基因组学研究方法,发现了一个树木杂交物种以及生殖隔离位点的分布式样。
物种形成模型可分为二歧分支式物种形成和杂交物种形成。二歧分支模型中,每个物种只对应一个祖先群体。杂交物种则源自于两个或多个类群。进一步地,杂交成种分为多倍体杂交成种和同倍体杂交物种形成。多倍体物种形成较常见于植物界。而同倍体杂交物种形成类群颇为少见。迄今,有5个认可度较高的同倍体杂交物种形成类群,均分布在美洲。
现存物种及类群间的生殖隔离强度往往高于其祖先群体间的隔离强度。孙永帅团队将这一原理引入到进化生态学与基因组学交叉研究中。即,在与生殖隔离关联的基因组区域上,现存类群间的基因流应低于其祖先群体间的基因流。在生殖隔离位点上,亲本物种的等位基因往往因环境、遗传限制而不能共存。基于这些原理,该团队应用群体基因组学方法鉴定了中华板栗(也称板栗)和锥栗的生殖隔离位点,进而用之检验峨眉锥栗是否起源于板栗和锥栗间杂交。与此前研究报道的5个同倍体杂交成种的实验设计不同,在峨眉锥栗杂交系统中,板栗和茅栗的姐妹种对关系为鉴定板栗和锥栗的生殖隔离位点提供了天然对照和便利(图1)。
该研究首先用多个方法分析峨眉锥栗与板栗、锥栗的遗传差异,为峨眉锥栗的分类地位提供了基因组学证据。然后,采用hhs方法、溯祖模型比较分析等对峨眉锥栗的杂交起源过程进行解析,并估算亲本物种对峨眉锥栗基因组的相对贡献。随后,该研究鉴定了与生殖隔离关联的候选基因组位点。在峨眉锥栗基因组中,仅6个生殖隔离位点来自于板栗。基因功能注释分析发现两个花期关联基因位于本研究鉴定的生殖隔离关联位点上。这些结果表明,亲本物种间生殖隔离位点的重新组合可为新物种形成的重要机制。深入分析发现,候选生殖隔离位点偏集中分布于基因组的低重组区域。研究认为,自然选择和遗传重组间互作塑造了峨眉锥栗基因组的进化过程。
西双版纳植物园植物进化生态学研究组孙永帅博士为研究论文的第一作者和通讯作者。该项研究得到了国家自然科学基金委,中国科学院和云南省的经费支持。
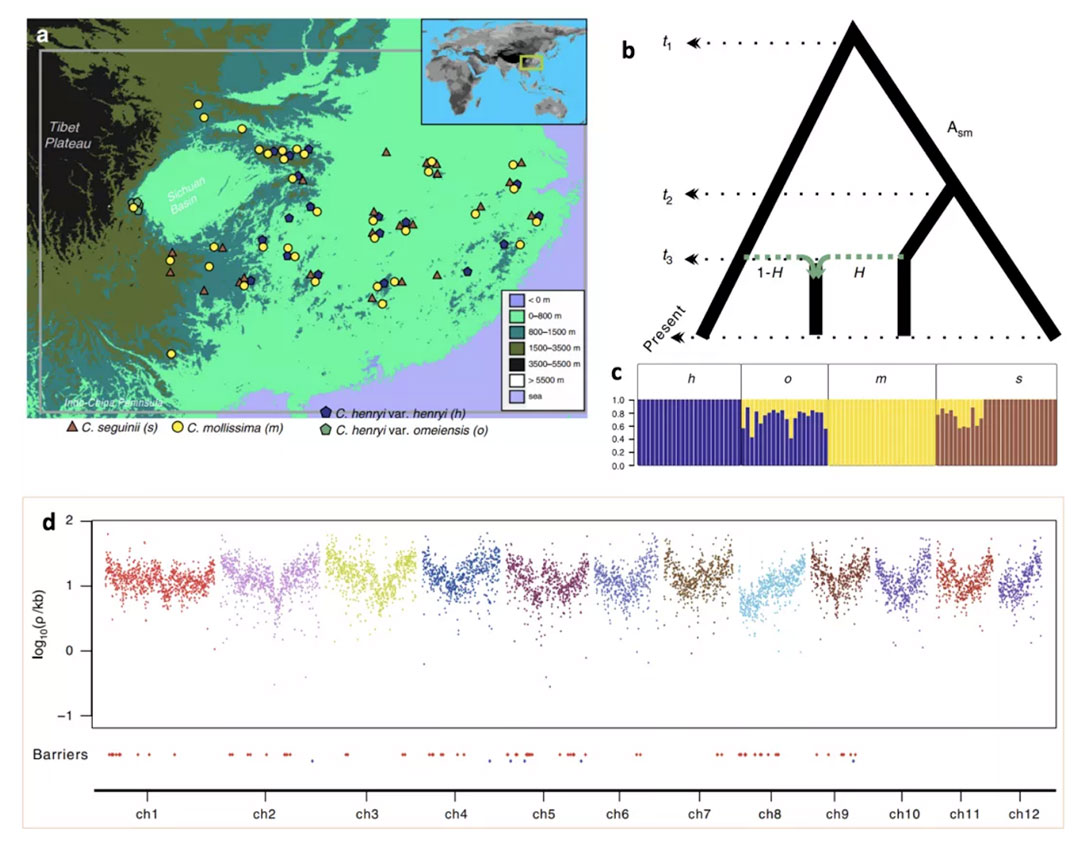
图1. 4个栗属Castanea类群的样品采集地(a)、演化关系(b)、遗传结构(c),以及板栗基因组的重组率分布以及生殖隔离位点的分布式样(d)
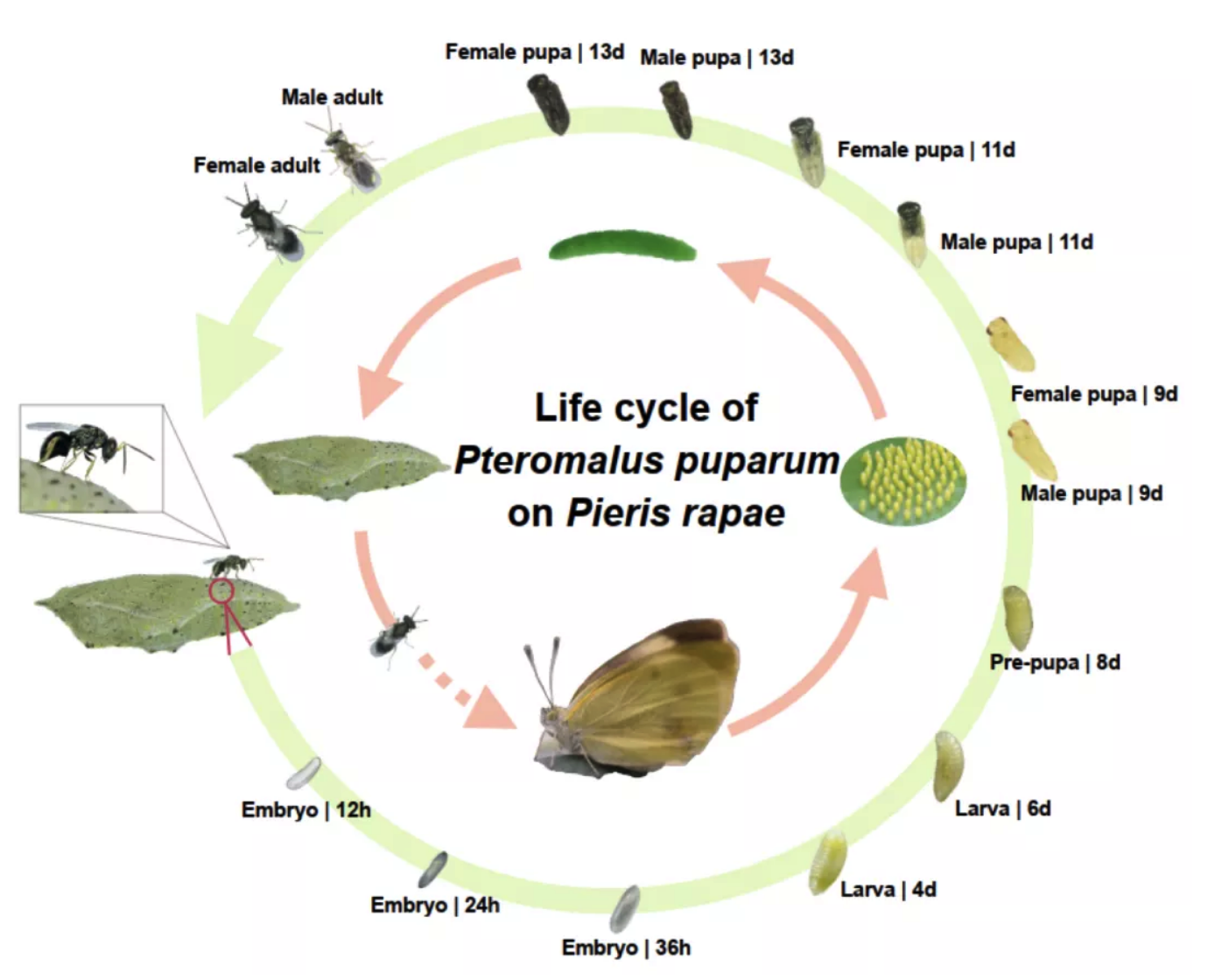
本研究结合短读长、长读长测序和Hi-C技术,生成了高质量染色体水平蝶蛹金小蜂基因组装配。组装的基因组大小为338 Mb,contig N50为38.7 kb,scaffold N50为1.16 Mb,结合Hi-C数据将scaffold组装到5条染色体上,scaffold N50提升至65.8 Mb,其中96%以上的组装碱基位于染色体上。基因组BUSCO评估达98%,表明该装配具有很高的完整性,为后续研究提供了极好的基因组资源。

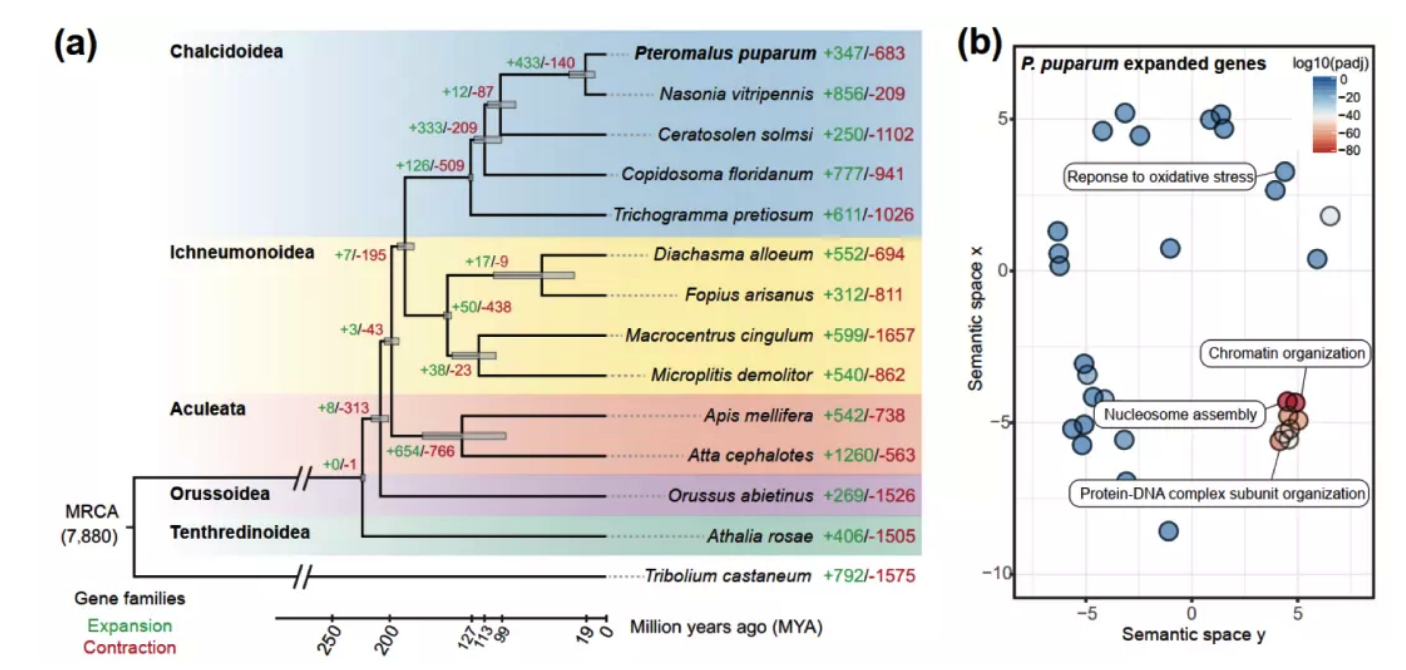
研究者利用蝶蛹金小蜂及其他12中代表性膜翅目昆虫的3399个单拷贝基因构建系统发育树,蝶蛹金小蜂与丽蝇蛹金小蜂进化关系最为接近,在约19 Mya年前发生分化(图3a)。GO分析发现蝶蛹金小蜂基因组中,扩张基因家族富集在核小体装配、染色质组织、蛋白质分解代谢过程、细胞凋亡过程和对氧化应激的响应等通路;几丁质分解代谢过程和脂质代谢过程中显示出显著收缩的基因家族(图3b)。
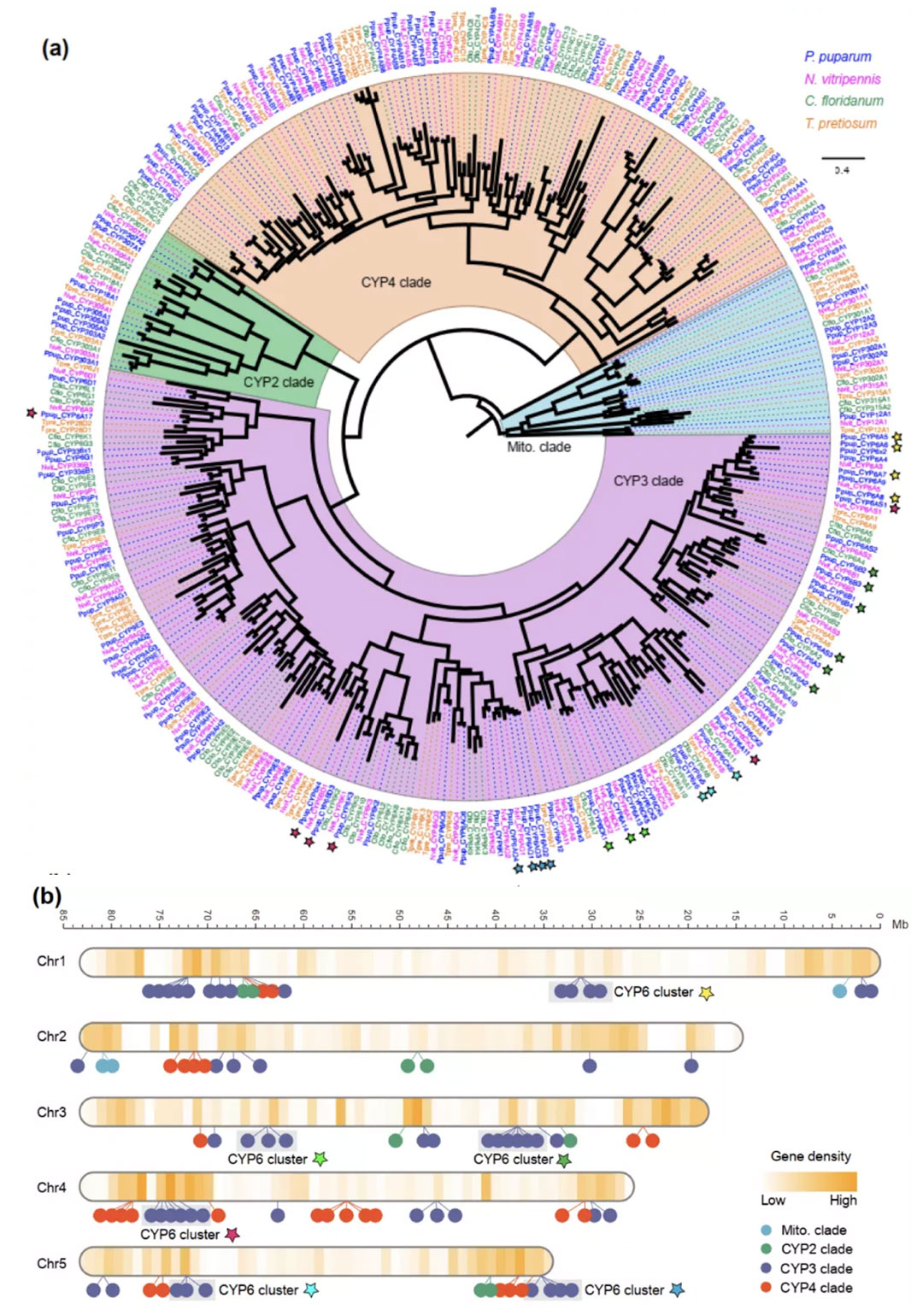
本研究是昆虫高质量基因组组装研究的极好范例,并将为寄生蜂分子生物学、系统进化及生物防治研究提供有价值的资源。
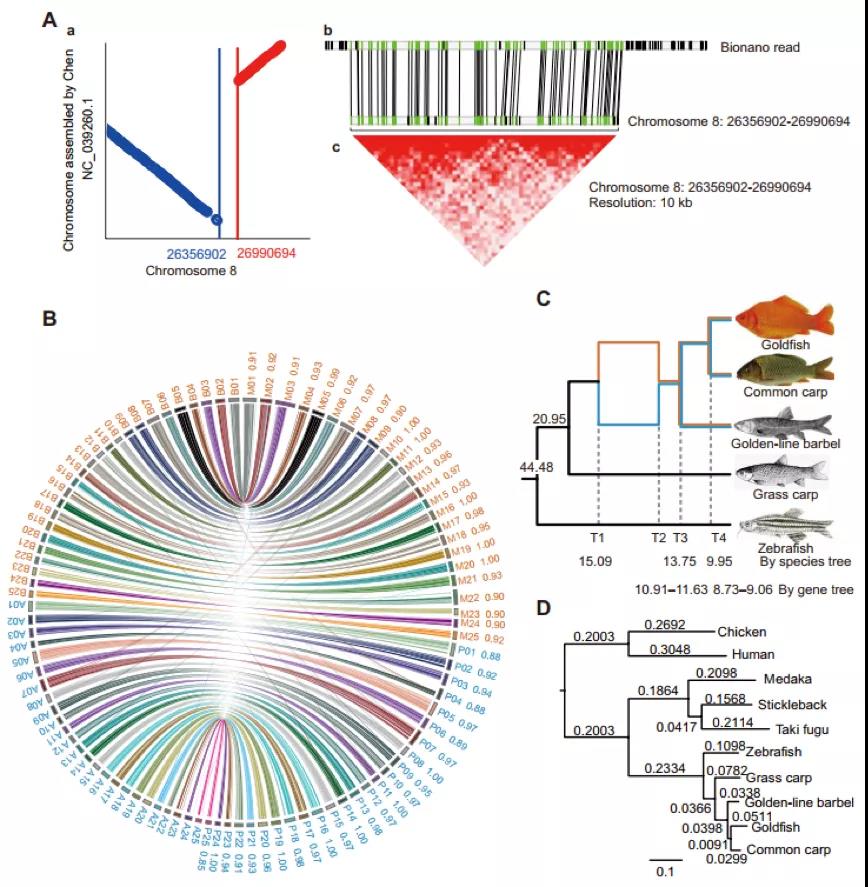
图1 红鲫基因组组装质量比较、共线性及鲤亚科鱼类多倍化演化历史重建。(A)本研究组装的红鲫基因组与前人发表的基因组共线性分析,提示光学图谱和Hi-C数据的辅助组装提升了多倍体基因组序列的连续性和准确度;(B) 红鲫与鲤鱼的亚基因组共线性分析结果;(C) 基于系统发育关系重建鲤亚科基因组的异源多倍化演化历史;(D) 基于单拷贝直系同源基因构建的物种树。