项目文章 | 超长测序 + Nextdenovo助力四个水稻基因组T2T组装
北京大学何航研究员、隆平高科杨远柱研究员与广东省农业科学院水稻研究所周德贵副研究员为论文共同通讯作者,北京大学博士生张宜林、隆平高科傅军、王凯博士、北京大学博士后韩雪为论文共同第一作者。北京大学现代农业研究院邓兴旺教授等对本研究提供了支持。本研究得到国家重点研发计划项目、湖南省科技创新计划等重大项目的支持。希望组提供Nextdenovo组装软件支持。
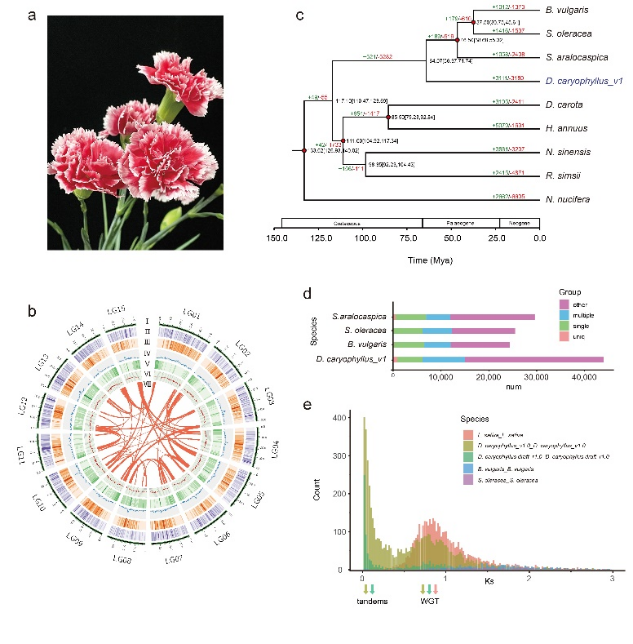
2022年6月24日,北京大学现代农学院邓兴旺实验室何航团队、隆平高科杨远柱团队与广东省农业科学院水稻研究所周德贵副研究员在Plant Biotechnology Journal期刊在线发表了研究论文“The telomere-to-telomere gap-free genome of four rice parents reveals SV and PAV patterns in hybrid rice breeding”,该研究利用HiFi和Ultra-long测序结合的组装策略,完成了湘陵628S、晶4155S、隆科638S与华占4个杂交水稻骨干亲本无缺口参考基因组组装,结合其余六个已发表的杂交水稻亲本基因组,系统分析了结构变异在杂交育种过程中的分布和影响,发现结构变异在F1功能基因及转录杂种优势起着重要作用。该研究展示杂种优势和杂交育种研究已进入无缺口参考基因组水平。
1. 四个两系杂交水稻骨干亲本无缺口参考基因组构建
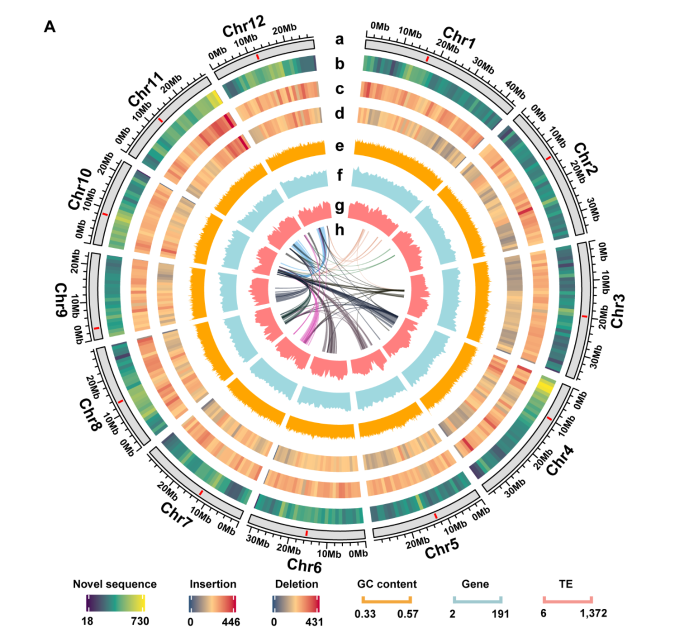
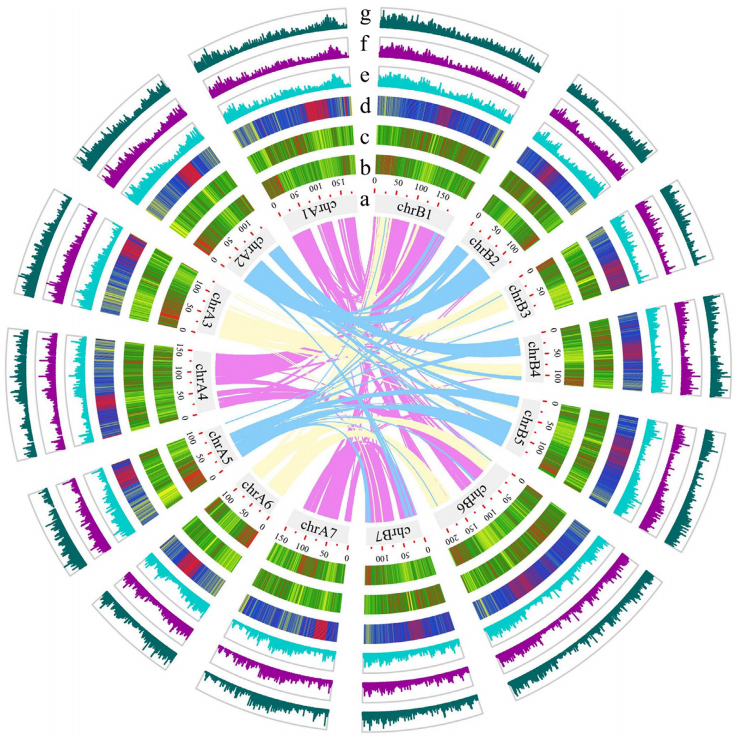
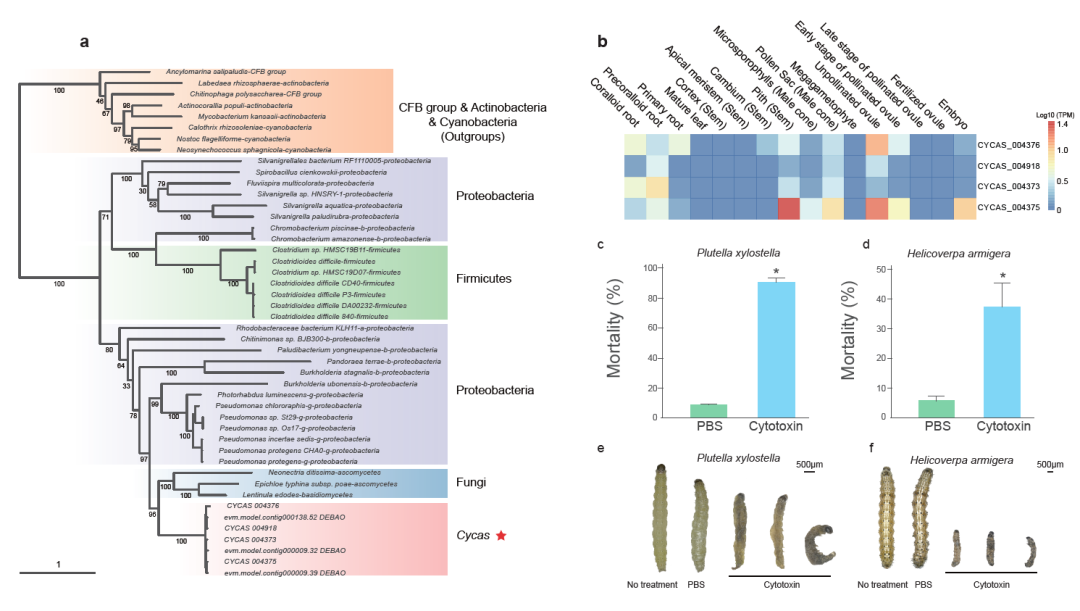
湘陵628S(XL628S)、隆科638S(LK638S)、晶4155S(J4155S)是隆平高科自主培育的骨干两系不育系,华占(HZ)是中国水稻所与广东省农业科学院水稻研究所共同培育的优秀的两系和三系骨干恢复系,与另一个广泛推广的恢复系五山丝苗(WSSM,R534)属于同一家系(图1A),隆平高科利用隆科638S、晶4155S和华占选配的超级稻品种隆两优华占和晶两优华占连续三年蝉联我国杂交水稻品种推广面积前三位,选配的隆两优534、晶两优534连续多年列我国杂交水稻品种推广面积前十位。利用PacBio(HiFi)和Nanopore(Ultra-long)平台进行三代测序后构建基因组,并通过新的组装方法获得无缺口基因组(图1B)。通过对基因组进行准确性、完整性、连续性的评估,二代测序比对率为98.83%~99.66%,覆盖度大于99.97%。BUSCO值为98.7%~98.9%,LAI值为24.02~24.89。每个基因组上都有着丝粒区和20到22个端粒,证明了基因组组装的高质量(图1C)。湘陵628S、隆科638S、晶4155S和华占无缺口参考基因组的构建,为其功能基因组研究和下一步分子设计育种研究奠定了基础。
2.籼稻基因组结构变异在杂交水稻培育中的应用
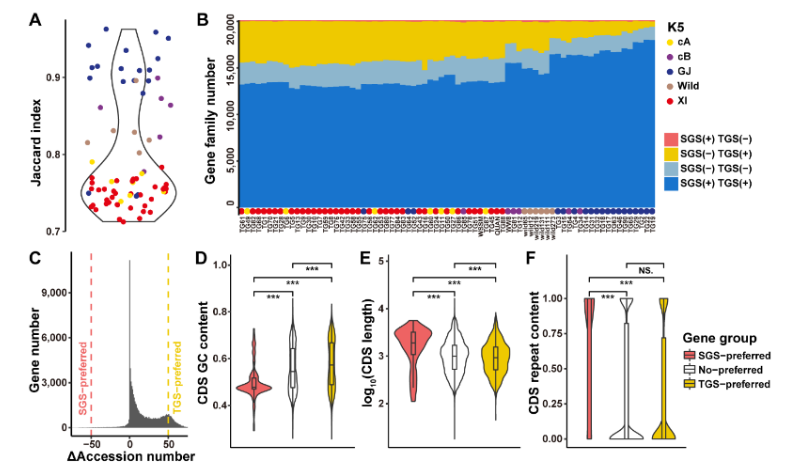
基于对以上四个杂交水稻骨干亲本以及已发表的珍汕97(ZS97)、明恢63(MH63)、蜀恢498(R498)、五山丝苗(WSSM)、Y58S、9311的基因组,与日本晴(MSU)进行基因组比对,鉴定出不同水稻亲本之间的结构变异。在日本晴和10个籼稻基因组之间鉴定出422,858-526,481个INDEL和56,817个非冗余的SV,包括52,943个PAV(图1D)。在10个籼稻基因组中,共有2182个功能基因受到PAV的影响,其中565个为籼粳稻之间PAV,包括OsLBD37,SaM,OsMFT1,OsF3H,其余为籼稻内部PAV基因,而其中70个为某一籼稻基因组特有PAV基因,这部分基因可能是该杂交组合表现优异的重要原因。
本研究收集的10个籼稻骨干亲本具有典型的代表性,包括2010年前栽培面积最大的著名杂交稻组合SY63(ZS97A/MH63);2012-2016年推广面积最大的超级杂交稻品种Y两优1号(Y58S/93-11);2018-2020年中国推广面积前三位的绿色超级杂交稻组合晶两优华占(J4155S/HZ),隆两优华占(LK638S/HZ)和晶两优534(J4155S/WSSM),以及推广面积前十位的隆两优534(LK638S/WSSM)。分别有593-738个双亲互补PAV基因发生在6个组合中,其中只有46个基因被所有组合共同利用,三个时期的杂交组合中利用的互补基因存在显著差异(图1E)。转录水平杂种优势基因也在互补基因中显著富集(图1F,G)。本研究原始数据与基因组及变异相关数据均在国家基因组科学数据中心PRJCA008812 (BioProject)和Github (https://github.com/yilinZhang-bio/Four-rice-gap-free-genome) 公布。
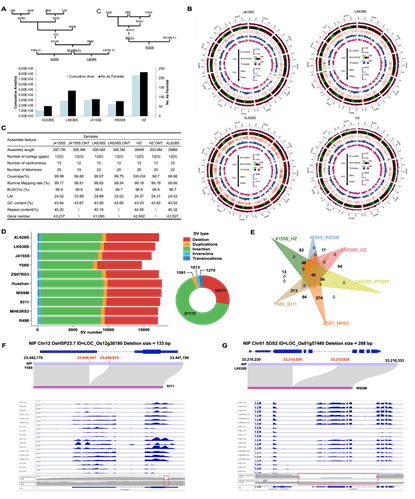 图1四个杂交水稻亲本无缺口基因组及杂交育种中的PAVs
图1四个杂交水稻亲本无缺口基因组及杂交育种中的PAVs






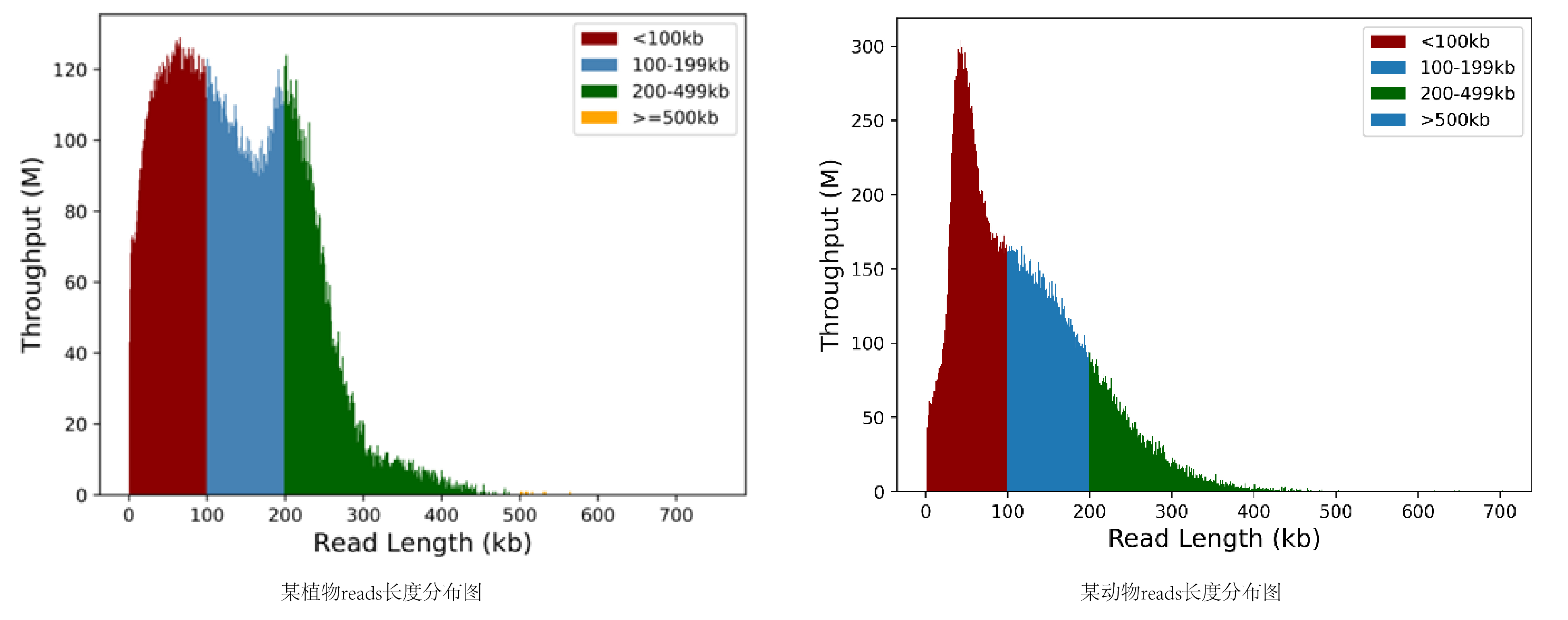
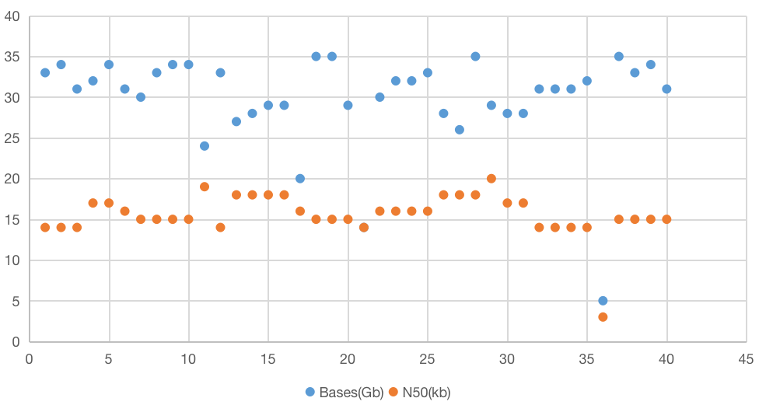 HiFi数据量和长度统计
HiFi数据量和长度统计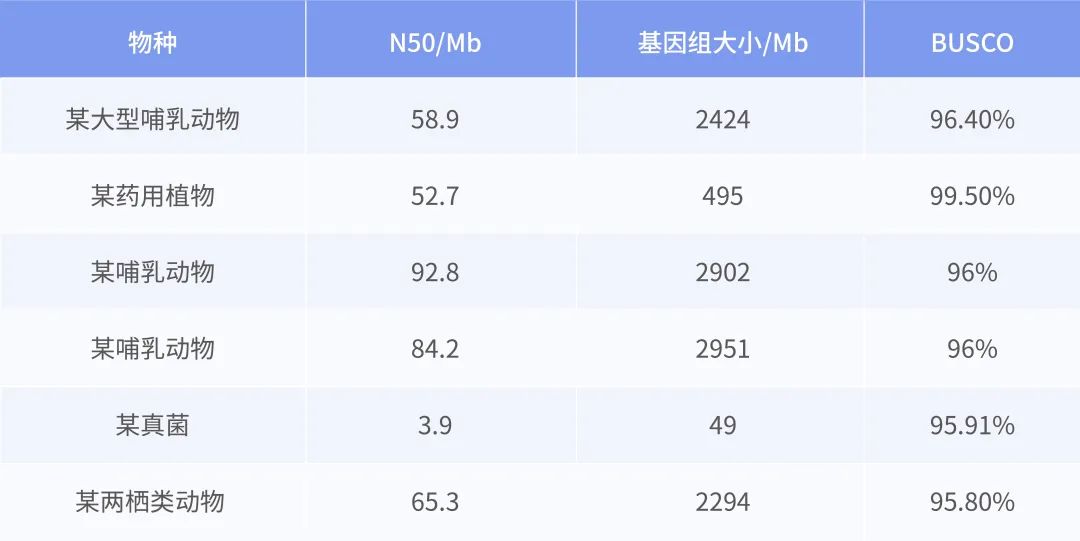




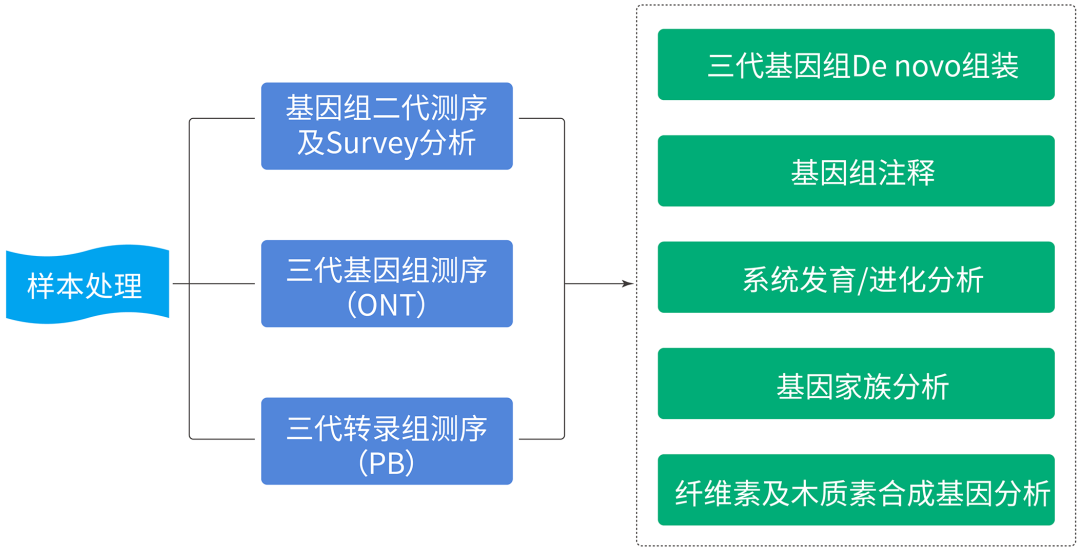
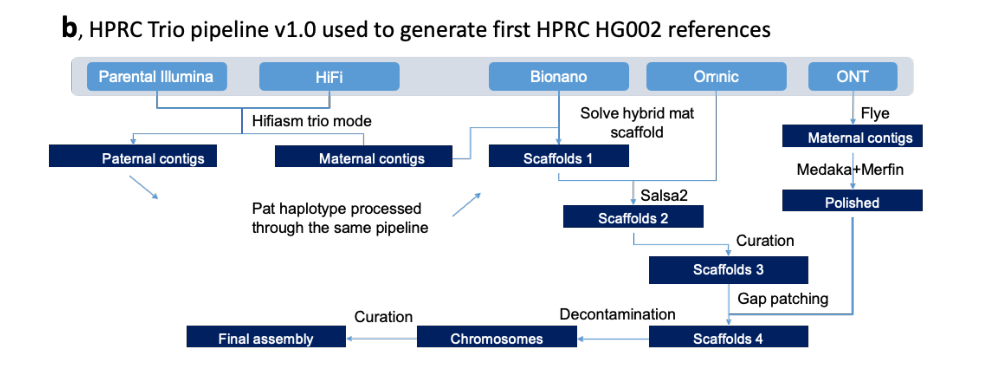 HPRC Trio pipeline v1.0组装流程图
HPRC Trio pipeline v1.0组装流程图