希望组与Bionano合作开展COVID-19感染患者基因组变异研究
/0 评论/在 新闻动态 /通过 zuBionano 将用于鉴定导致冠状病毒疾病易感性的基因变异的研究
第一项研究将在中国武汉进行;欧洲和北美的研究有望紧随其后

*本文基于Bionano Genomics公司官方网站新闻”Bionano Genomics Will Be Used in Research Identifying Gene Variantsthat Contribute to Coronavirus Disease Susceptibility”翻译。
项目文章两连发||三代测序助力药用动物圆点斑芫菁、菲牛蛭基因组草图组装
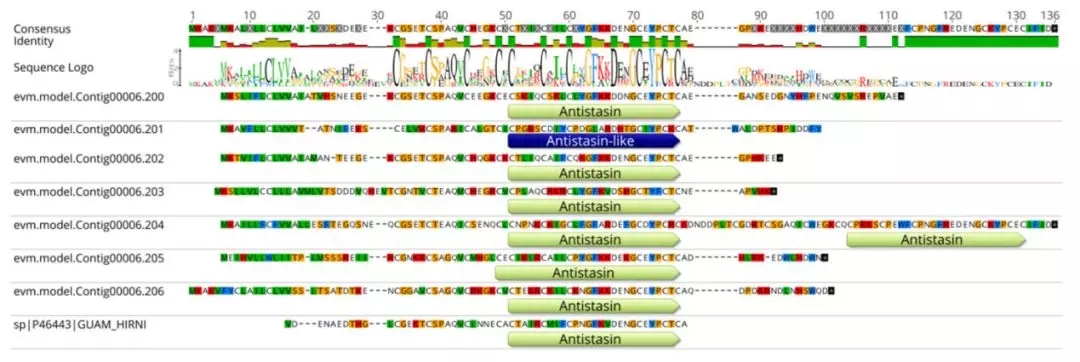
/0 评论/在 科研动态 /通过 zu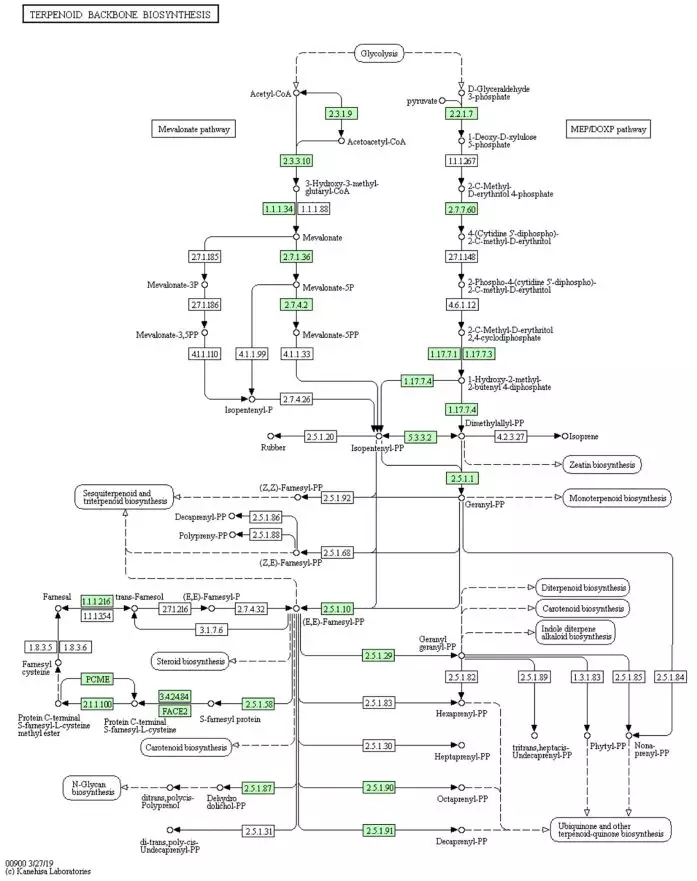
图1 萜类生物合成“KEGG通路图”,绿色方框基因在圆点斑芫菁基因组中发现。
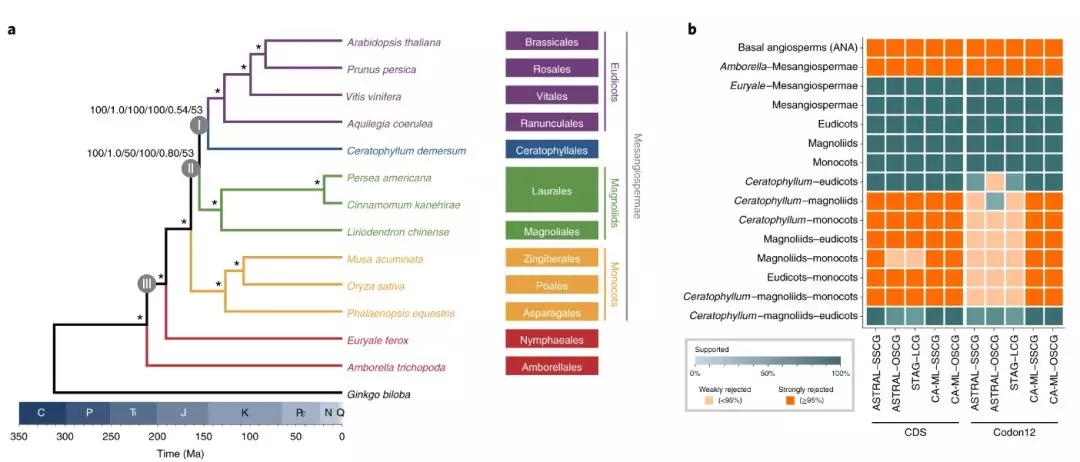
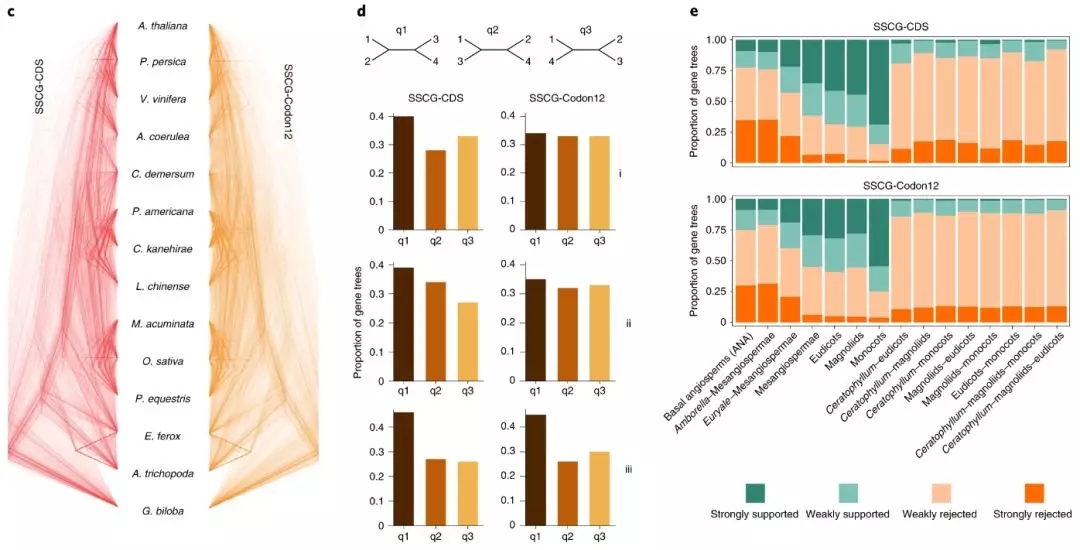
项目文章||芡实与金鱼藻三代基因组揭示早期被子植物演化
/0 评论/在 科研动态 /通过 zu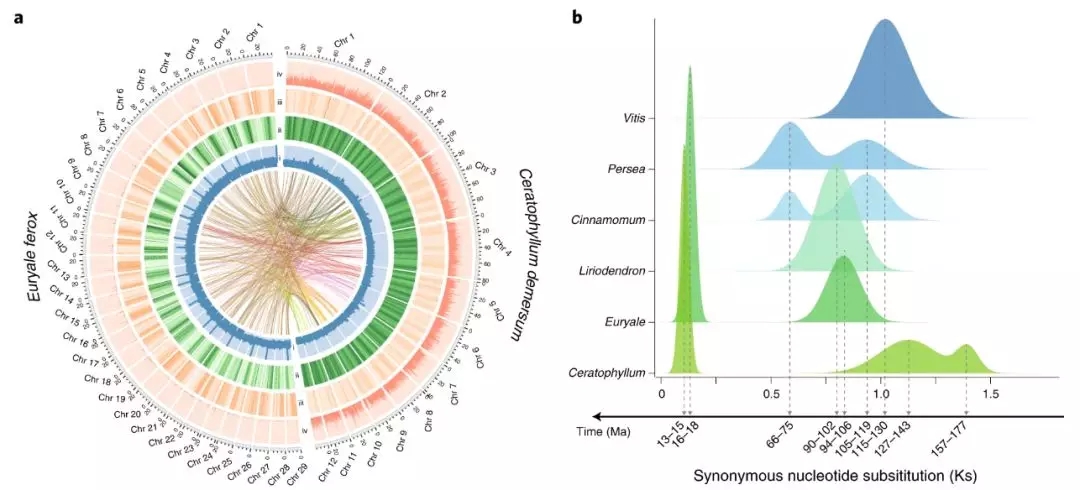
疫情防控利器——希望组全长新型冠状病毒(2019-nCoV)试剂盒研制成功,实时监控病毒演化与变异即将成为现实
/0 评论/在 新闻动态 /通过 zu
3. 开发基于全长基因组的病毒演化和变异检测技术

我们和华为5G和华为云团队一起完成了实时机动的测序数据的上云传输和计算实验,能够确保在没有固定IT基础设施的临时区域或缺少有线网络和本地计算资源的地点能够通过无线网络进行病原体的检测和分析。
希望组武汉测序中心通过ISO9001:2015质量管理体系认证
/0 评论/在 新闻动态 /通过 zu
云 +AI 赋能三代基因测序,希望组荣获华为云年度峰会“选择不凡奖”
/0 评论/在 新闻动态 /通过 zu


希望组2019大事件盘点
/0 评论/在 新闻动态 /通过 zu希望组三代测序助力特发性震颤相关致病基因发现
/0 评论/在 科研动态 /通过 zu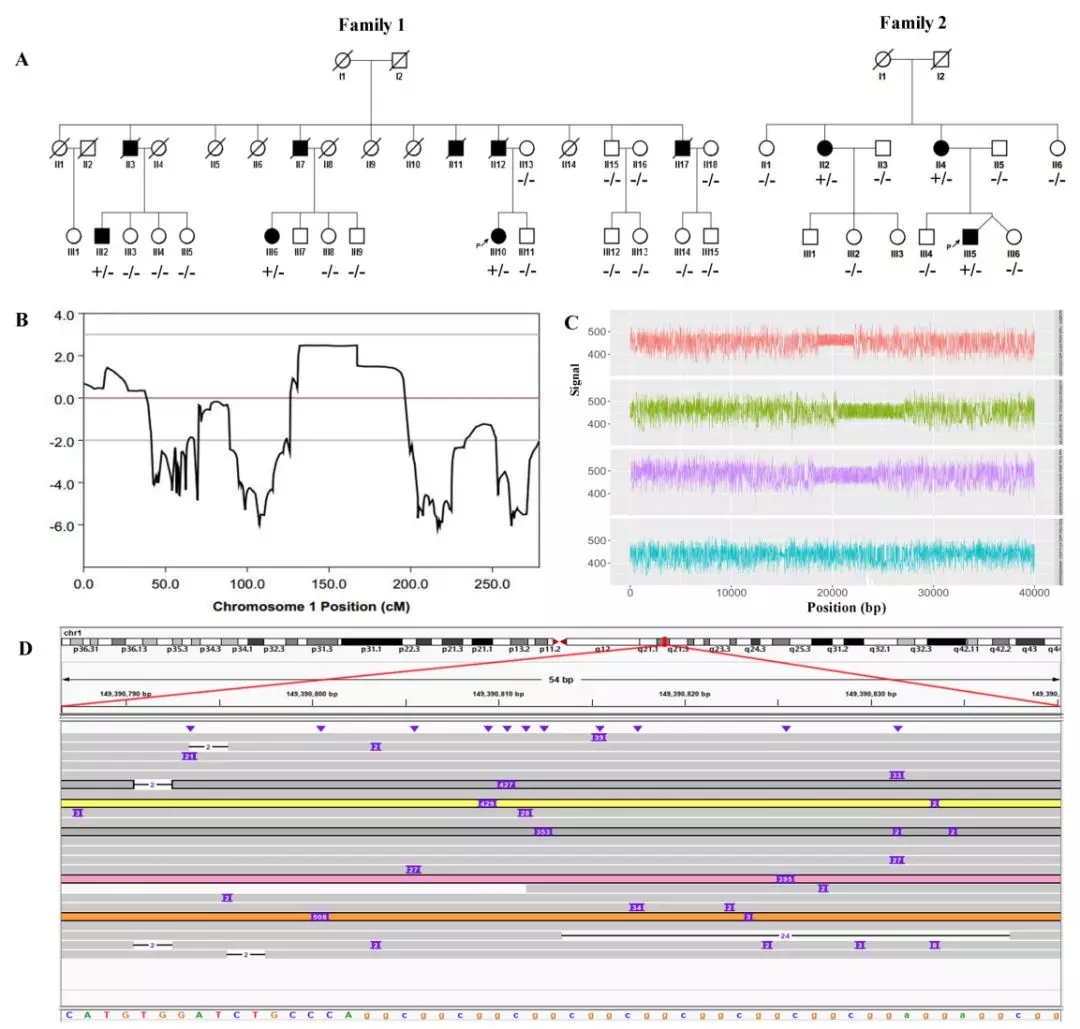
超级植物-蕨类的三代基因组攻略:ONT超长+Bionano Gen2+Hi-C+NextDenovo
/0 评论/在 科研动态 /通过 zu蕨类在地球上已经存在了3.35亿年,是现存最古老的植物之一。远在恐龙出现和大陆漂移之前,它们便占据着原始大陆的沼泽森林,通过羽状叶片储存大量太阳能。蕨类死亡后的遗骸被埋葬在泥泞的沼泽沉积物中,经过千万年压缩转化成当代工业革命的能量——煤。

蕨类具有重要系统发育地位,尽管蕨类的基因组数据量仍然有限,但现有数据强烈表明,它们的基因组动力学与所有其他陆地植物截然不同。蕨类基因组的典型特征是染色体数目多,这被认为是通过多倍体的多个全基因组复制(WGD)周期产生的。然而,与被子植物多倍体相比,蕨类的多倍体后二倍体化过程通常涉及基因沉默而不是DNA消除,从而导致染色体数目异常增多,同时保持二倍体基因的表达[1]。蕨类染色体的平均数目(n = 63.5)[2]是被子植物平均数目的三倍多(n = 21.55)[3]。而蕨类基因组大小平均为12Gb[3],最大甚至达到148Gb[4,5]
 蕨类基因组研究现状
蕨类基因组研究现状




表2 NextDenovo的组装案例

联系我们
科技服务联系方式:
售前及技术支持
Tel: 153 8703 7487
E-mail: inquiry@grandomics.com
售后及项目管理
Tel: 173 6290 2840
E-mail:pm-receive@grandomics.com
医学诊断联系方式:
售前及项目支持
Tel: 010-80765017
HR
Tel: 153 2745 1541
E-mail : hr@grandomics.com
官方微信公众号

希望组

希望组科技服务

希望组诊断服务
